|
|||||||||||||||||||||||||||||||||||||||||||||||||
|
|
С помощью BLASTP я нашла последовательности, сходные с последовательностью моего белка - сериновой D-аланил-D-аланин карбоксипептидазой
из бактерии Nostoc sp., штамм PCC 7524 (идентификатор в базе данных RefSeq WP_015141669.1). В дополнительных параметрах
поиска я изменила максимальное число находок на 20000, чтобы точно найти все последовательности. Условия поиска доступны по
ссылке.

Рисунок 1. Выравнивание белка D-аланил-D-аланин карбоксипептидазы из бактерии Nostoc sp., штамм PCC 7524 с белком из Anabaena variabilis, построенное BLASTP. 
Рисунок 2. Выравнивание белка D-аланил-D-аланин карбоксипептидазы из бактерии Nostoc sp., штамм PCC 7524 с ABC-транспортным ATP-связывающим белком из Enterococcus faecalis, построенное BLASTP. 
Рисунок 3. Выравнивание белка D-аланил-D-аланин карбоксипептидазы из бактерии Nostoc sp., штамм PCC 7524 с белком из Aliivibrio logei, построенное BLASTP. Гомологами исходной последовательности можно считать 4674 найденных последовательностей, придерживаясь критерия, что гомологичными являются находки с E-value < 1e-03 и query cover не менее 70%, то есть в построенное выравнивание вошло не менее 70% исходной последовательности. На рисунке 4 представлено графическое изображение результатов поиска. Показано всего 100 последовательностей, максимальное значение E-value установлено на 9e-04 (0,0009), однако отображенные находки имеют гораздо меньшее E-value (так как в графическое представление вошло 100 первых находок из списка, которые имеют наименьшее E-value), и все эти последовательности можно считать гомологичными исходной. 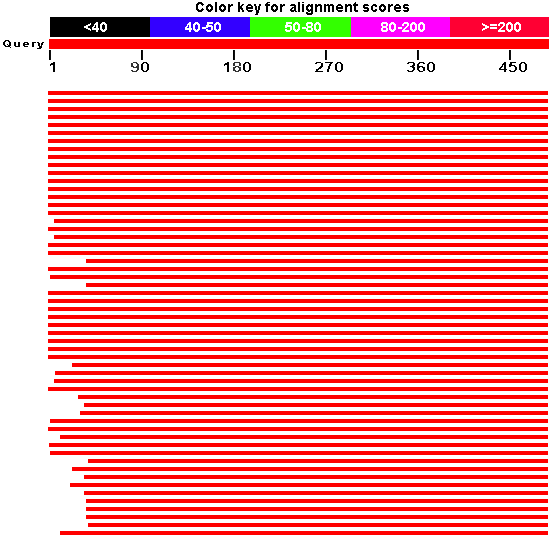
Рисунок 4. Графическое представление результатов поиска белка D-аланил-D-аланин карбоксипептидазы из бактерии Nostoc sp. в базе данных RefSeq с помощью BLASTP. Показано 100 находок, значение E-value не более 0,0009.
Также я провела поиск для отдельного таксона - Cyanobacteria. Все остальные условия поиска я оставила неизменными, просто дополнительно указала
таксон
(сслыка
на условия поиска). Всего найдено 482 последовательности. Для сравнения результатов двух поисков я выбрала одну из находок, которая встречается в обоих
случаях -
Для создания карты локального сходства я выбрала последовательность из генома Dickeya zeae (идентификатор в RefSeq WP_038903300.1) и построила
парное выравнивание с последовательностью моего белка с помощью BLAST. Полученная карта представлена на рисунке 5. Можно увидеть довольно длинный совпадающий
участок примерно с 80 остатка query (последовательность моего белка, отложена по горизонтали) и с 50 остатка гомологичной последовательности (по вертикали).
Места прерывания линии на рисунке соответсвуют гэпам в выравнивании, например, участок примерно с 270 по 280 остатки в query не соответствует никакому участку
во второй последовательности. Действительно, в выравнивании в этом месте в последовательности белка WP_038903300.1 стоит гэп длиной в 7 остатков. Также видны
две небольшие черточки, которые обозначают короткие совпадающие участки, выделенные как отдельные находки. Однако для них E-value слишком высокое (4,2 и 5),
а длина выравнивания слишком низкая (6 и 11 остатков), поэтому эти совпадения можно считать случайными. Для иллюстрации соответствия элементов карты локального
сходства и выравнивания на рисунке 6 представлено выравнивание этих последовательностей.
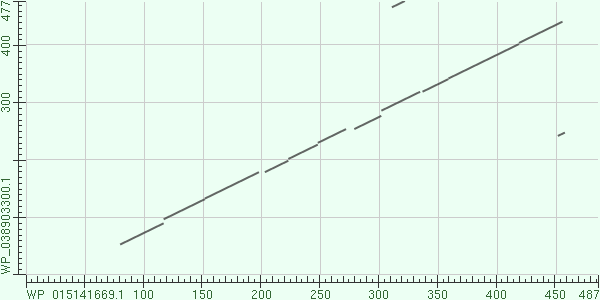
Рисунок 5. Карта локального сходства для выравнивания белка D-аланил-D-аланин карбоксипептидазы из бактерии Nostoc sp., штамм PCC 7524 (идентификатор в RefSeq WP_015141669.1, по горизонтали) с белком из бактерии Dickeya zeae (идентификатор WP_038903300.1, по вертикали). 
Рисунок 6. Выравнивание белка D-аланил-D-аланин карбоксипептидазы из бактерии Nostoc sp., штамм PCC 7524 (query) с с белком из бактерии Dickeya zeae (sbjct), построенное BLASTP. Я создала свою базу данных из множественного выравнивания align_06.fasta из практикума 8. Для этого я использовала команду makeblastdb, а из данного файла с выравниванием удалила все гэпы и получила список последовательностей. Таким образом, в моей базе данных было 9 последовательностей белков. Далее я произвела поиск моего белка в этой базе данных с помощью команды blastp (последовательность моего белка в формате fasta). Параметры лучшей находки представлены в таблице 2. Выравнивание показано на рисунке 7.
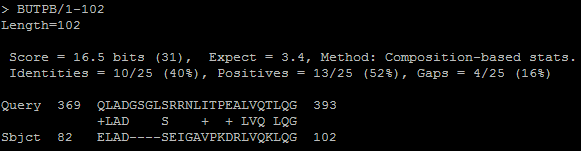
Рисунок 7. Выравнивание белка D-аланил-D-аланин карбоксипептидазы из бактерии Nostoc sp., штамм PCC 7524 с последовательностью BUTPB из новой базы данных. Также на рисунке приведены параметры этого выравнивания, представленные в таблице 2. Длина получившегося выравнивания небольшая, значение Bit score низкое относительно уже рассмотренных в этом практикуме выравниваний. Значение E-value также достаточно высокое, особенно для такой маленькой базы данных из 9 последовательностей. Стоит также учитывать, что вторая из находок имела практически такие же параметры, только немного хуже (например, bit score = 16,2 и E-value = 4). Все эти данные позволяют считать данную находку случайной, и нет оснований говорить о гомологии последовательностей. |
||||||||||||||||||||||||||||||||||||||||||||||||
|
© Наталия Кашко, 2015 |
|||||||||||||||||||||||||||||||||||||||||||||||||