|
|
Упражнения
UniProt
- Код референсного протеома бактерии Brucella abortus: UP000002719
- В референсном протеоме B. abortus белков систем секреции (secretion system) 15
- В референсном протеоме B. abortus белков систем секреции, кроме тех, которые помечены как не охарактеризованные (uncharacterized protein) 14
- В референсном протеоме B. abortus белков, которые связывают атом железа, 29
PubMed
- Статья Блюменталя (Blumenthal) с названием "Транс-сплайсинг и опероны C. elegans" ("Trans-splicing and operons in C. elegans"); PMID: 23175478
По запросу "Blumenthal[au] trans-splicing[ti] AND operons[ti] AND С. elegans[ti]" PubMed выдает 3 результата
- Список публикаций, в которых цитируется данная работа, в формате excel
PDB
- Список структур бактериальных белков из систем секреции типа IV (type IV secretion system) в формате excel.
Использовался запрос "TaxonomyTree Search for Bacteria (eubacteria) and StructTitleQuery: struct.title.comparator=contains struct.title.value=type IV secretion system", то есть структуры,
в названии которых есть "type IV secretion system", из организмов, принадлежащих таксону "Bacteria"
Тема 2. Белки, содержащие пирролизин
Пирролизин - 22-ая природная аминокислота, закодированная в геноме. Принятое обозначение - Pyl, O, структурная формула показана на рисунке 1. Впервые была обнаружена в Methanosarcina barkeri,
метаногенной архее, которая использует различные субстраты (CO2, уксусную кислоту, метиламины, метанол и метиловые тиолы) для получения энергии и синтезирует метан в качестве побочного
продукта [1].
В этой архее пирролизин входит в состав активных центров ферментов, участвующих в процессе метаногенеза. Позже было показано, что эта аминокислота встречается у всех метаногенов.
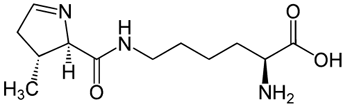
Рисунок 1. Структурная формула пирролизина.
В клетках пирролизин синтезируется из двух молекул L-лизина. За синтез отвечают продукты генов pylB, pylC и pylD (вместе обозначаются как pylBCD и обычно входят в состав оперона).
Один из возможных путей синтеза представлен на рисунке 2.
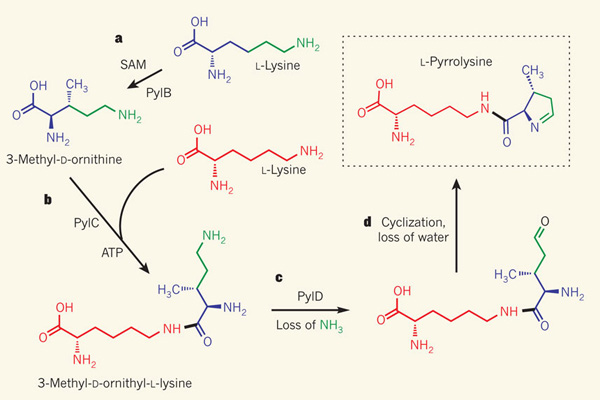
Рисунок 2. Путь синтеза L-пирролизина из двух молекул L-лизина [2]. Подробное описание представлено по
ссылке.
В мРНК пирролизин кодируется кодоном UAG, который в обычных случаях является стоп-кодоном. Для правильной трансляции этого стоп-кодона и вставки пирролизина в белок есть специальная тРНК с антикодоном CUA
(закодирована геном pylT) и аминоацил-тРНК-синтетаза класса II (pylS ген). Кассета pylTSBCD встречается у всех известных организмов, использующих пирролизин, и является необходимой и
достаточной для синтеза и вставки этой аминокислоты в белки. Показано, что клетки E. coli с трансформированной кассетой pylTSBCD способны самостоятельно синтезировать пирролизин и
транслировать в него UAG-кодон [3] [4].
Белки, содержащие пирролизин, я искала в базе данных UniProt по запросу "annotation:(type:non_std pyrrolysine)". Всего найдено 90 белков, из них 29 проверены и имеют записи в SwissProt. Результаты
поиска представлены в таблице excel.
Пирролизин встречается в белках у представителей доменов Археи (Archaea) и Бактерии (Bacteria). Среди архей эта аминокислота характерна для семейства Methanosarcinaceae, среди бактерий - для отдельных
представителей типа Фирмикуты (Firmicutes), например Desulfitobacterium hafniense и Acetohalobium arabaticum [5]. Кластеры pylTSBCD
у архей и бактерий немного отличаются, например, ген pylS у бактерий представлен двумя генами, pylSn и pylSc, кодирующими N-конец и C-конец аминоацил-тРНК-синтетазы соответственно.
Не так давно была обнаружена еще одна группа метаногенных архей - Methanomassiliicoccales, называемая "седьмой порядок метаногенов"
(7th Order of Methanogens) [6] [7]. В таблице результатов UniProt их белки представлены с 43 по 74 номер,
они предсказаны на основании отсеквенированных геномов Candidatus Methanomethylophilus alvus и Candidatus Methanomassiliicoccus intestinalis.
Основных белков, содержащих пирролизин, всего 3: монометиламин метилтрансфераза (mtmB), диметиламин метилтрансфераза (mtbB) и триметиламин метилтрансфераза (mttB).
Эти белки гомологичны у известных метаногенов. Однако у представителей Methanomassiliicoccales пирролизин входит в состав и других белков, например, Cas1 эндонуклеаза CRISP-системы (M9SDX5).
PDB-структуры известны для белка mtmB1 из археи Methanosarcina barkeri (O30642). Идентификаторы записей в PDB: 1L2Q, 1NTH, 1TV2, 1TV3, 1TV4 (таблица pdb-структур в формате
excel). Данные идентификаторы взяты из таблицы, полученной с помощью UniProt (последняя колонка "Cross-reference (PDB)"). Примечательно, что пирролизин был
открыт на основании X-ray анализа именно этого белка, и структура 1L2Q описана в работе [1]. Также в PDB есть множество записей о белках, закодированных генами
оперона pylTSBCD, в комплексе с пирролизином или с другими лигандами. Эти структуры можно найти в PDB по запросу "pyrrolysine", всего выдается 51 запись (таблица записей, полученных с помощью
такого запроса, в формате excel). Такой запрос показывает все записи, которые имеют хоть какое-нибудь отношение к пирролизину, потому что поиск
идет по всем строкам pdb-файла. В том числе выдаются структуры, представляющие разные этапы синтеза этой аминокислоты из лизина с участием белков pylD или pylC. Чтобы усовершенствовать
поиск, можно использовать параметры "Chemical ID(s): PYL and Polymeric type is Polymeric". По этому запросу должны выдаваться те структуры, в цепи у которых есть химическое вещество пирролизин.
Поиск по такому запросу выдает только 2 структуры: 1NTH и 1L2Q (таблица с результатами поиска в формате excel).
Поиск просто по пирролизину выдает также много записей о тРНК(Pyl) и аминоацил-тРНК-синтетазе pylRS. Эти структуры активно исследуются, потому что pylRS имеет низкую селективность к субстрату
и к антикодону тРНК (в отличие от других аминоацил-тРНК-синтетаз, которые связывают строго определенную тРНК со сторого определенной аминокислотой), таким образом, на основе этой ортогональной пары можно
создать механизмы для вставки неканонических аминокислот в белки [8].
Для поиска статей в PubMed я ввела запрос pyrrolysine[tiab] (ищет статьи, в которых встречается слово "pyrrolysine" в названии или в аннотации (abstract)). По нему мне выдали 111 статей, из которых 22
обзора (review), а у 48 статей доступен бесплатный полный текст (Free full text). Все найденные статьи представлены в таблице в формате csv. Далее можно
усовершенствовать запрос, отобрав только обзоры и статьи в бесплатном доступе. Для этого нужно записать запрос следующим образом: "pyrrolysine[tiab] AND review[pt] AND free full text[sb]".
По этому запросу выдается 8 находок (представлены в таблице). Ссылки на отдельные исследования я брала из найденных обзоров.
_______________________________________________________________________________________________
[1]. Hao B, et al. A new UAG-encoded residue in the structure of a methanogen methyltransferase. [PubMed]
[2]. Gaston MA, Zhang L, Green-Church KB, Krzycki JA. The complete biosynthesis of the genetically encoded amino acid pyrrolysine from lysine.
[PubMed]
[3]. Blight SK, et al. Direct charging of tRNA(CUA) with pyrrolysine in vitro and in vivo. [PubMed]
[4]. Longstaff DG, et al. A natural genetic code expansion cassette enables transmissible biosynthesis and genetic encoding of pyrrolysine.
[PubMed]
[5]. Prat L, et al. Carbon source-dependent expansion of the genetic code in bacteria. [PubMed]
[6]. Borrel G, et al. Comparative genomics highlights the unique biology of Methanomassiliicoccales, a Thermoplasmatales-related seventh order of methanogenic archaea that encodes pyrrolysine.
[PubMed]
[7]. Borrel G, et al. Unique characteristics of the pyrrolysine system in the 7th order of methanogens: implications for the evolution of a genetic code expansion cassette.
[PubMed]
[8]. Fekner T, Chan MK. The pyrrolysine translational machinery as a genetic-code expansion tool..
[PubMed]
|

