|
||||||||||||||||||||||||||||||||||
|
|
Я произвела поиск по последовательности моего белка D-аланил-D-аланин карбоксипептидазе (последовательность в формате fasta) на сайте Pfam, чтобы найти домены, встречающиеся в белке. Было найдено одно семейство - Peptidase_S13 (PF02113). Я сохранила seed выравнивание этого семейства и построила множественное выравнивание 9 последовательностей, входящих в состав seed, в программе JalView. Множественное выравнивание seed можно посмотреть в формате fasta, а также в проекте JalView (проект в формате jvp). На рисунке 1 представлено выравнивание с раскраской ClustalX. 
Рисунок 1. Множественное выравнивание последовательностей seed семейства Peptidase_S13 (PF02113) из банка Pfam. Черной рамкой выделен блок, с которым я в дальнейшем работала в практикуме. Раскраска ClustalX. Я сохранила консенсусную последовательность для всего выравнивания с помощью JalView. Она представлена в формате fasta. Далее я выбрала блок (позиции 432-448, на рисунке 1 обведен черной рамкой), для которого сохранила графическое представление LOGO. Оно было получено с помощью WebLogo 3 на сайте http://weblogo.threeplusone.com/. LOGO для данного блока представлено на рисунке 2. Консенсусная последовательность данного блока в формате fasta - по ссылке. 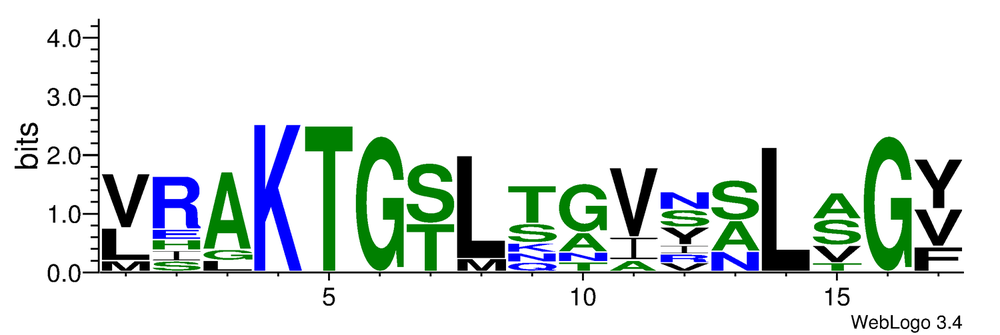
Рисунок 2. LOGO для одного из блоков в выравнивании последовательностей seed семейства Peptidase_S13 (PF02113). Построено с помощью программы WebLogo 3.4. Также я проверила, какая из последовательностей seed наиболее похожа на консенсусную. Для этого я построила парные глобальные выравнивания всех последовательностей из выравнивания с полученной консенсусной. Результаты приведены в таблице 1.
Как видно из таблицы 1, на консенсусную последовательность наиболее похож белок O06380_MYCTU из организма Mycobacterium tuberculosis.
Я составила сильный и слабый паттерны для выбранного блока и попыталась найти последовательности, содержащие
мотив с данным паттерном, в SwissProt. Для поиска я использовала сайт
http://prosite.expasy.org/scanprosite/.
По сильному паттерну найдено 6 последовательностей. Пять из них соответствуют гомологичным последовательностям
из семейства, с которым я работала в предыдущем практикуме по
PSI-BLAST. Шестая - синтаза пептидоглюкана из зеленой
водоросли Nephroselmis olivacea (идентификатор Q9TL36). Возможно, этот белок не является полностью
гомологичным моему белку (его длина 709 аминокислотных остатков, что примерно на 300 а.о. превышает длину моего
белка), однако он содержит данный мотив, который, скорее всего, отвечает за связывание каких-то компонентов
клеточной стенки. Для поиска я использовала следующий сильный паттерн:
|
|||||||||||||||||||||||||||||||||
|
© Наталия Кашко, 2015 |
||||||||||||||||||||||||||||||||||