Белок из первого семестра beta-lactamase domain-containing protein из организма Cyanothece sp. PCC 8801 (YP_002373080.1) был заменен в базе данных NCBI Protein на не избыточным белок MULTISPECIES: ribonuclease J из организма Cyanothece (WP_012596190), который имеет идентичную последовательность.
Название белка: RNase J1 (ribonuclease J).
PDB код: 3zq4.
Разрешение структуры: 3Å.
Файл с электронной плотностью белка 3zq4 (EDS map) доступен для скачивания на сайте PDBe. Наличие электронной плотности означает, что автор модели положил в PDB не только саму модель, но и экспериментальные данные. Экспериментальные данные называются "Файл структурных факторов" (Strucure factors). Он тоже доступен для скачивания с сайта PDB.
Построение изображения электронной плотности вокруг остова полипептидной цепиНа Рис. 1 изображен белок 3zq4. На Рис. 2 представлено изображение электронной плотности белка с уровнем подрезки 1.5. Белок достаточно большой и состоит из 4 аминокислотных цепей, поэтому в дальнейшем при визуализации электронной плотности я использовала только одну аминокислотную цепь D.
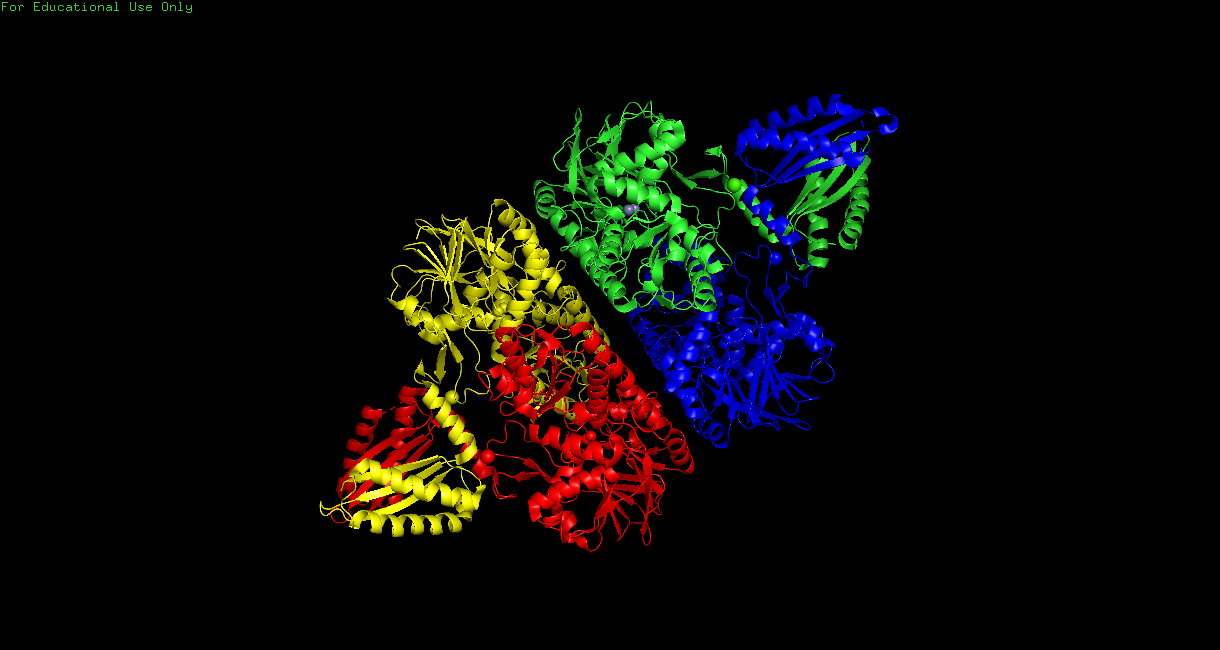
Рис. 1. Изображение белка 3zq4.
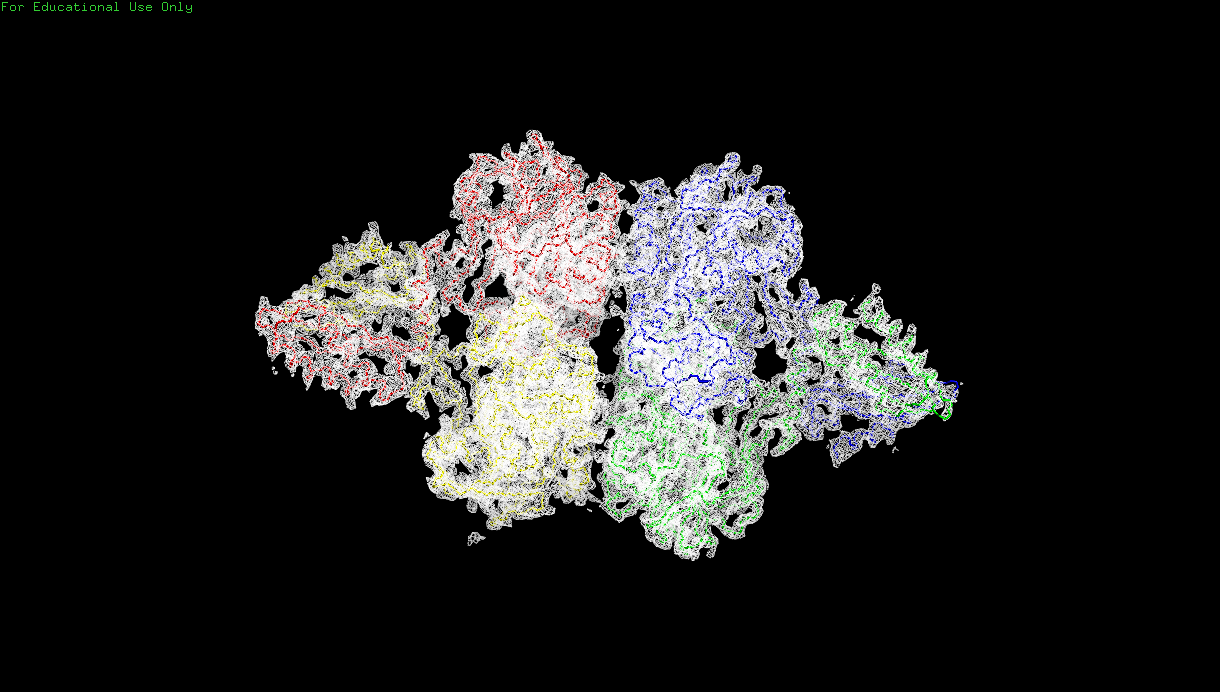
Рис. 2. Изображение электронной плотности вокруг остова полипептидных цепей белка 3zq4 с уровнем подрезки 1.5. Аминокислотная цепь D выделена синим цветом.
На Рис. 3 - 8 предсталены изображения электронной плотности вокруг остова полипептидной цепи D с уровнем подрезки от 0.5 до 3. При увеличении значении уровня подрезки полипептидная цепь хуже описывается отображаемой электронной плотностью. Начиная с уровня подрезки 2, появляются участки цепи, которые располагаются вне поверхности электронной плотности. В основном это участки петель белковой цепи.
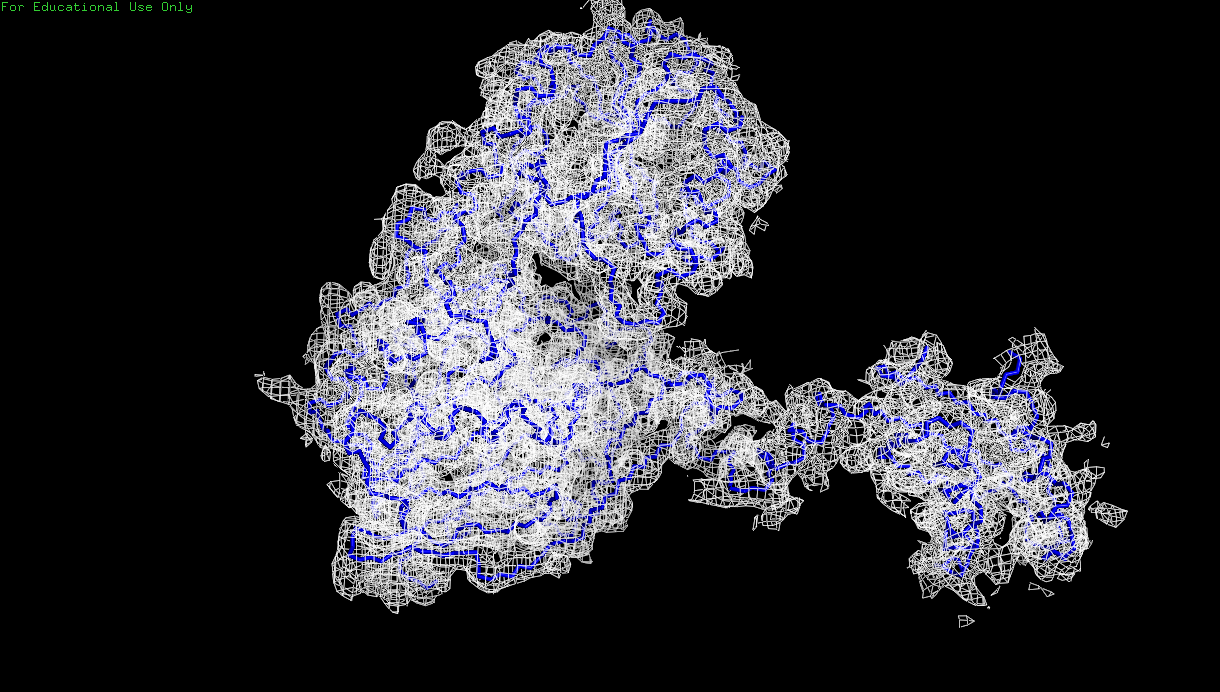
Рис. 3. Изображение электронной плотности вокруг остова полипептидной цепи D белка 3zq4 с уровнем подрезки 0.5.
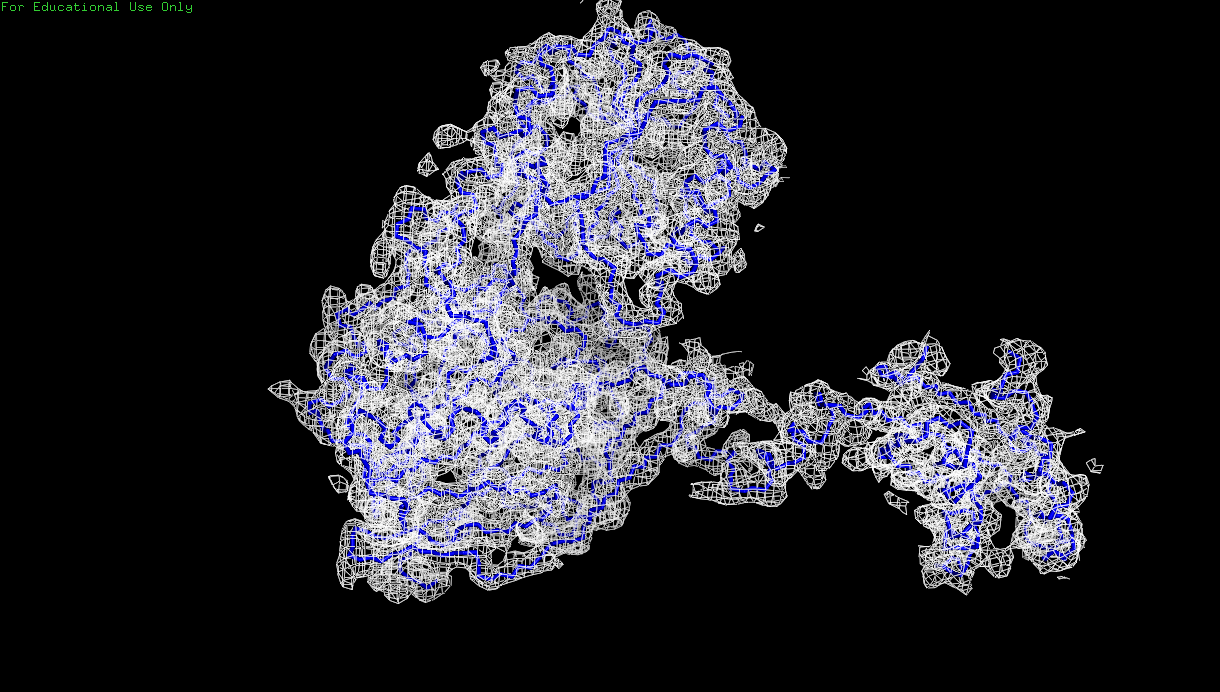
Рис. 4. Изображение электронной плотности вокруг остова полипептидной цепи D белка 3zq4 с уровнем подрезки 1.
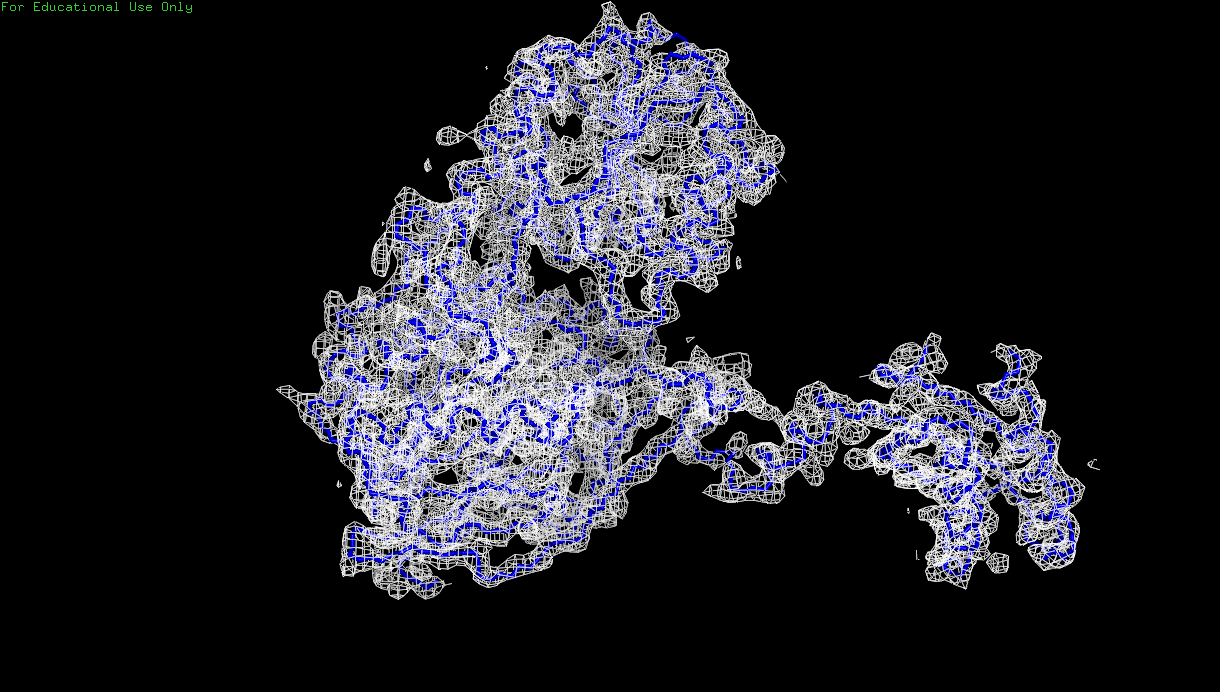
Рис. 5. Изображение электронной плотности вокруг остова полипептидной цепи D белка 3zq4 с уровнем подрезки 1.5.
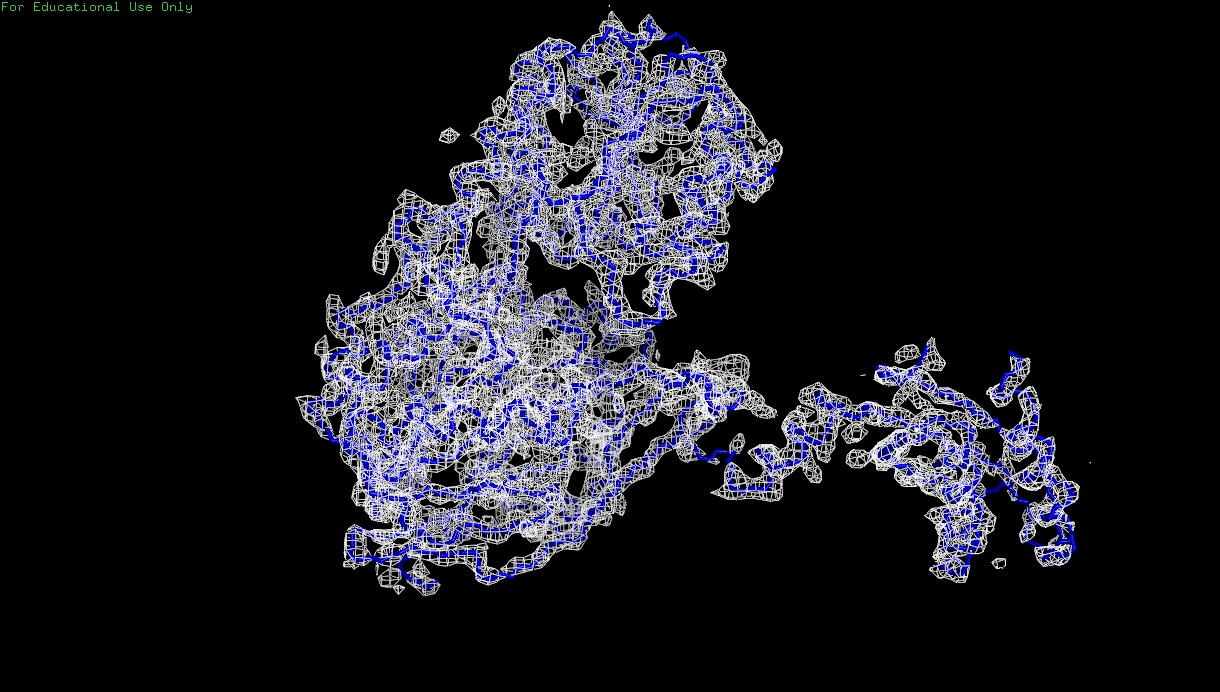
Рис. 6. Изображение электронной плотности вокруг остова полипептидной цепи D белка 3zq4 с уровнем подрезки 2.
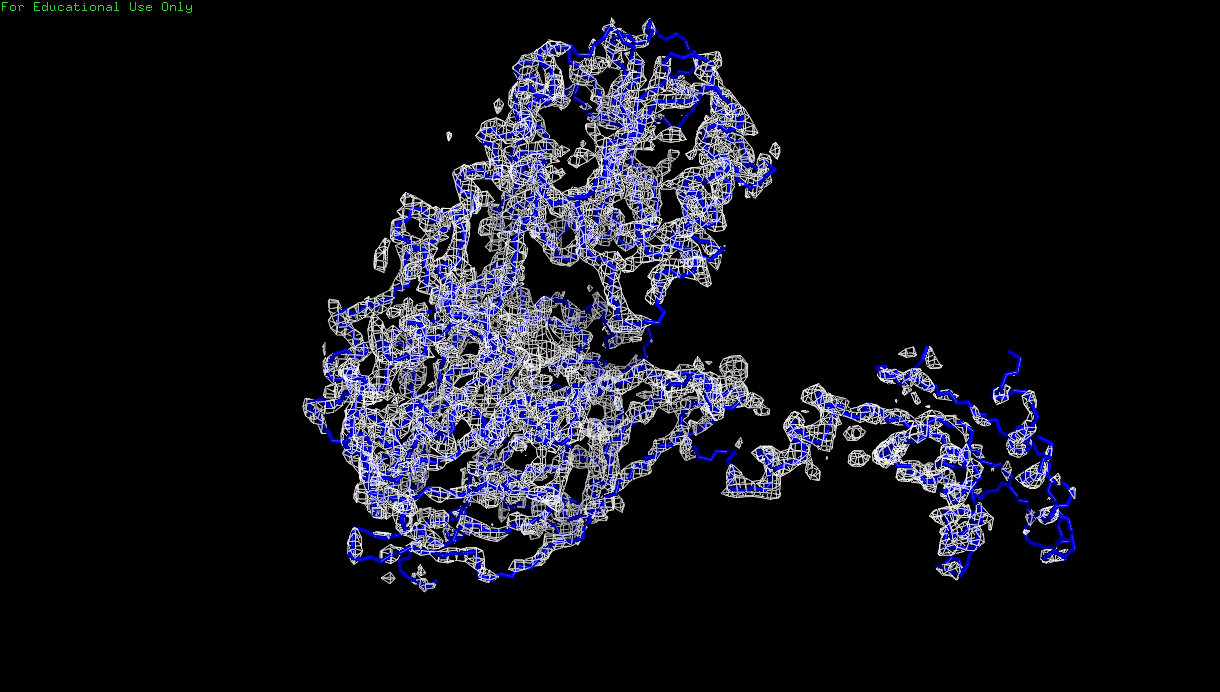
Рис. 7. Изображение электронной плотности вокруг остова полипептидной цепи D белка 3zq4 с уровнем подрезки 2.5.

Рис. 8. Изображение электронной плотности вокруг остова полипептидной цепи D белка 3zq4 с уровнем подрезки 3.
Построение изображения ЭП вокруг трех различных аминокислотных остатковНа Рис. 9 - 13 предсталены изображения электронной плотности вокруг трех аминокислотных остатков полипептидной цепи D (Asp164, Phe165 и Lys166) с уровнем подрезки от 0.5 до 3. Для данных аминокислотных остатков по респределению электронной плотности достаточно точно можно определить положение атомов и тип аминокислоты.
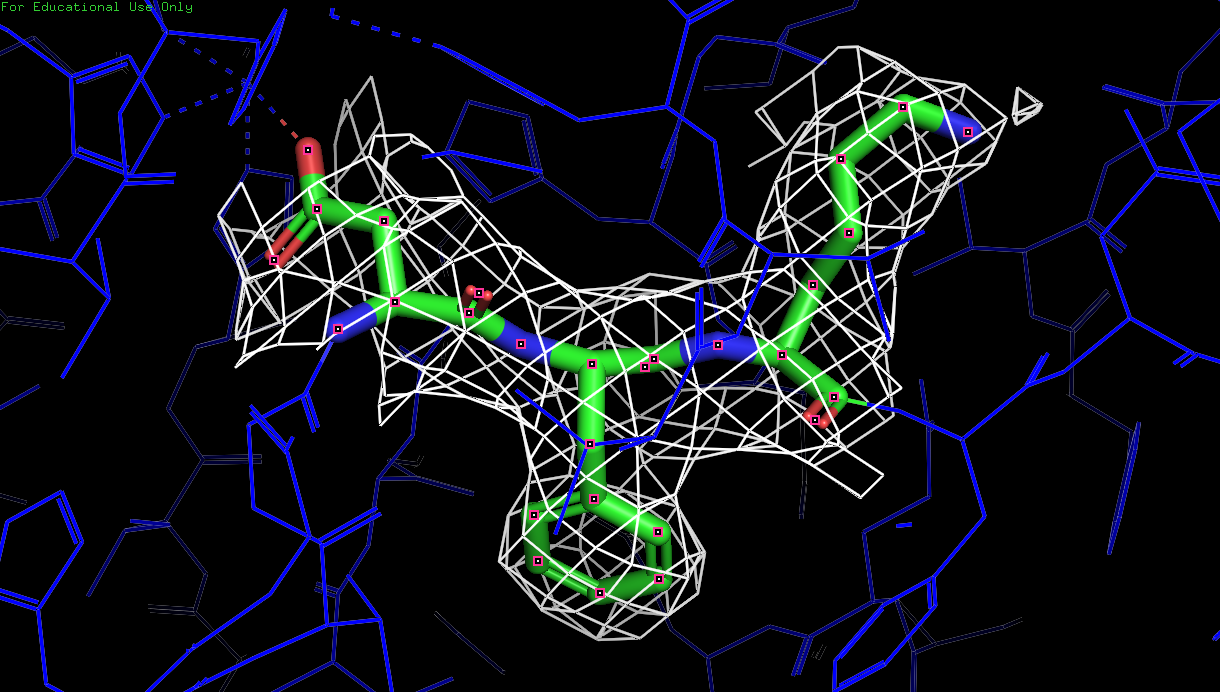
Рис. 9. Изображение электронной плотности вокруг остова полипептидной цепи D белка 3zq4 с уровнем подрезки 0.5.
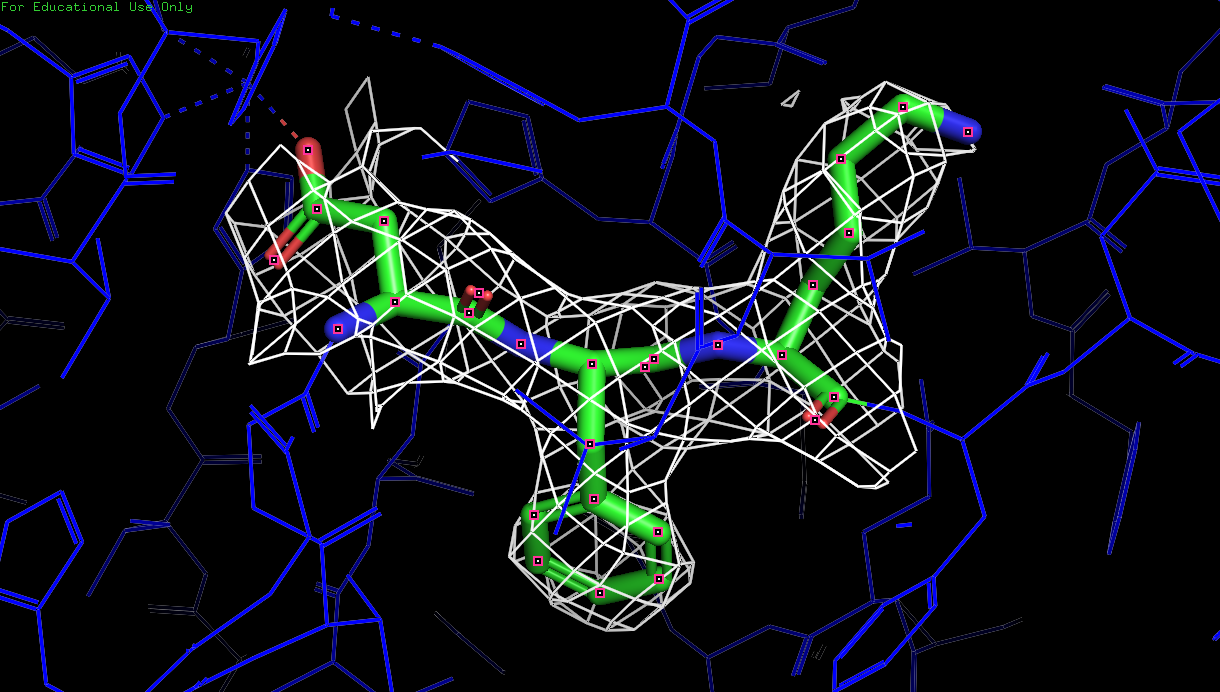
Рис. 10. Изображение электронной плотности вокруг остова полипептидной цепи D белка 3zq4 с уровнем подрезки 1.
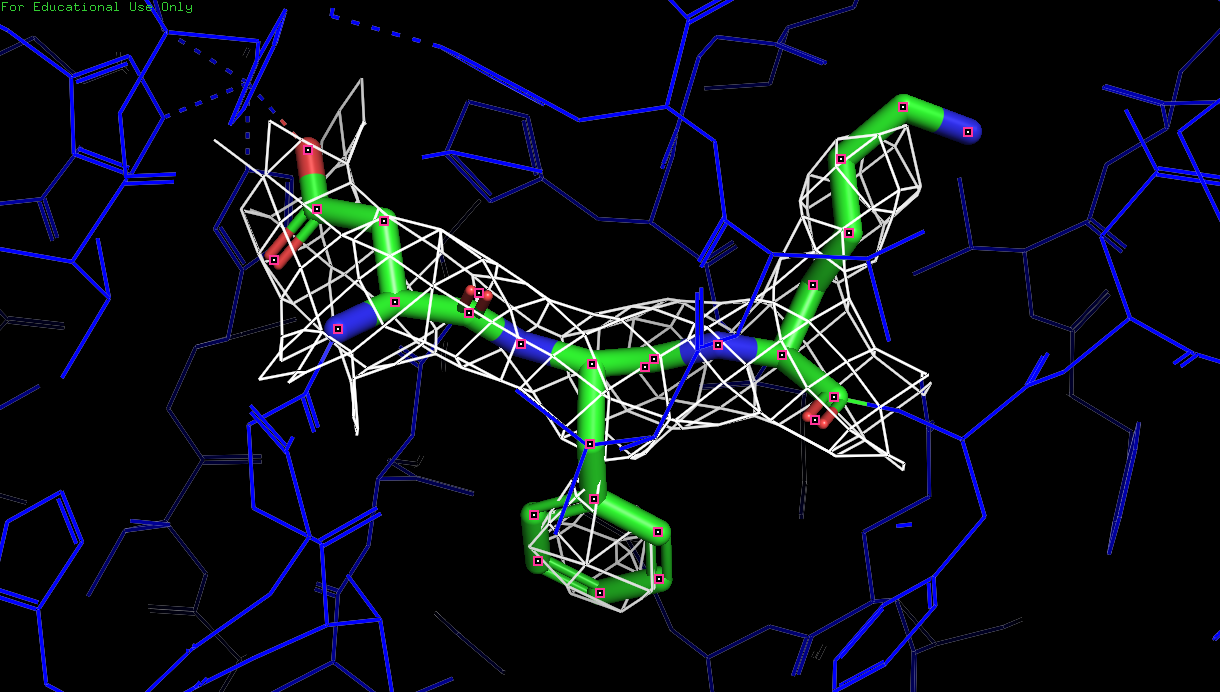
Рис. 11. Изображение электронной плотности вокруг остова полипептидной цепи D белка 3zq4 с уровнем подрезки 2.
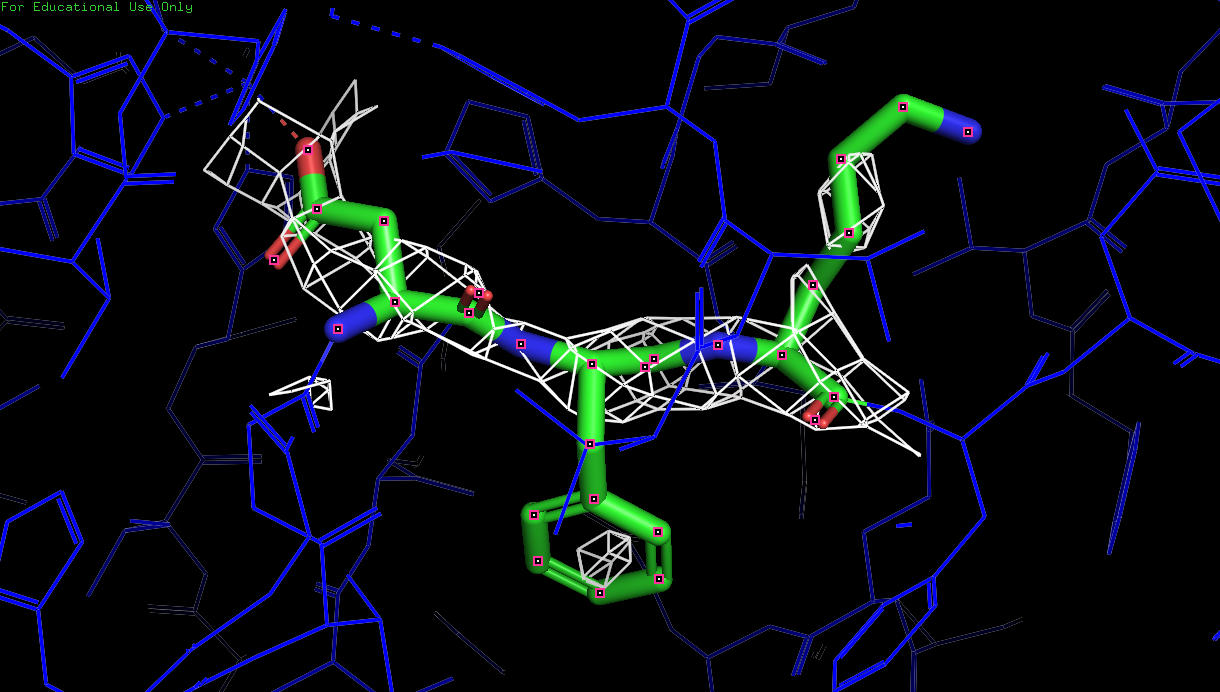
Рис. 12. Изображение электронной плотности вокруг остова полипептидной цепи D белка 3zq4 с уровнем подрезки 3.
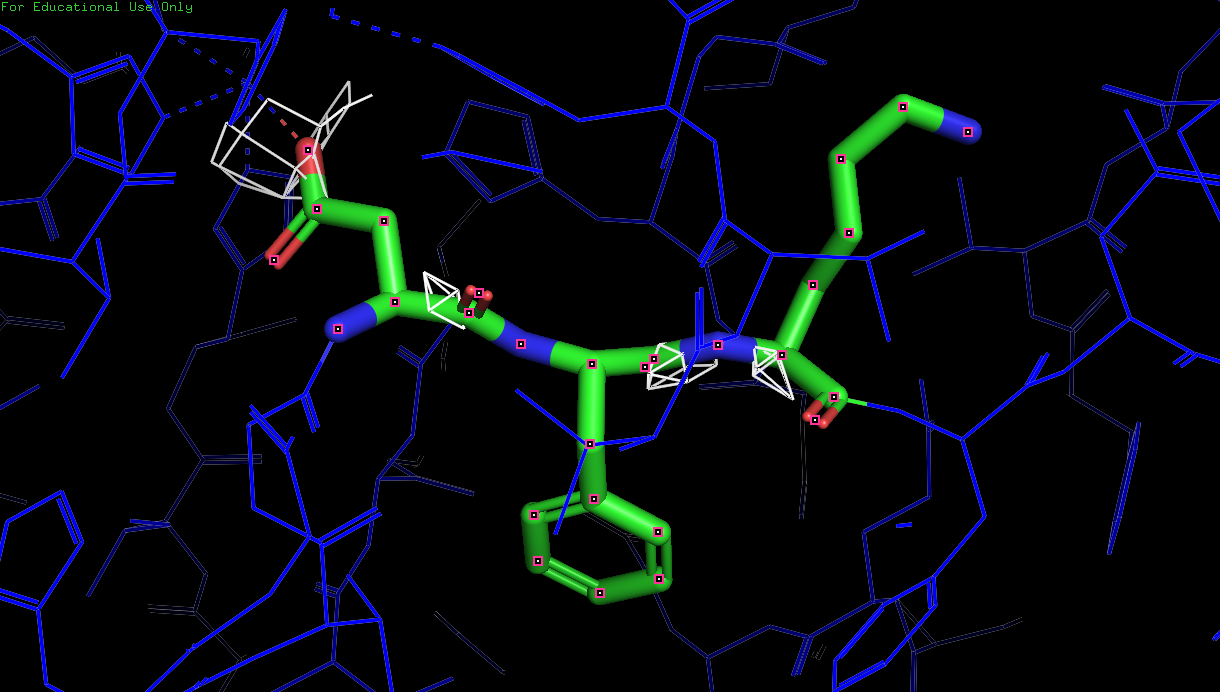
Рис. 13. Изображение электронной плотности вокруг остова полипептидной цепи D белка 3zq4 с уровнем подрезки 4.
Координаты атомов модели покрываются электронной плотностью, однако при больших уровнях подрезки аминокислотные остаткои на поверхности белка оказываются непокрытыми электронной плотностью.
Основные команды в PyMol:
fetch 3zq4
load 3zq4.ccp4, 3zq4_map
isomesh full_protein, 3zq4_map, 1, 3zq4, carve = 2