При использовании поиска по сходству структур в PDBeFold, были найдены структурные гомологи белка Bet v 1 (из березы, Betula verrucosa) 1BV1 (таблица 1).
Таблица 1. Характеристики некоторых структур, гомологичных белку 1BV1.
| PDB код | Название белка и организм | RMSD | Длина выравнивания | %seq |
|---|---|---|---|---|
| 1FM4 | Birch pollen allergen BET V 1L from Betula pendula | 0.90 | 158 | 92 |
| 2LPX | Strawberry Allergen Fra a 1e from Fragaria x ananassa | 2.01 | 148 | 55 |
| 1E09 | Major Cherry Allergen Pru av 1 from Prunus avium | 1.63 | 153 | 59 |
| 5MMU | Major apple allergen Mal d 1 from Malus domestica | 1.89 | 155 | 54 |
C помощью PDBeFold было получено множественное выравнивание белков из стаблицы 1. Выравнивание последовательностей, построенное по структурам - fasta.seq, файл с совмещёнными структурами - send.rasmol. Структурное множественное выравнивание представлено на рис. 1. Все гомологи довольно хорошо накладываются друг на друга.
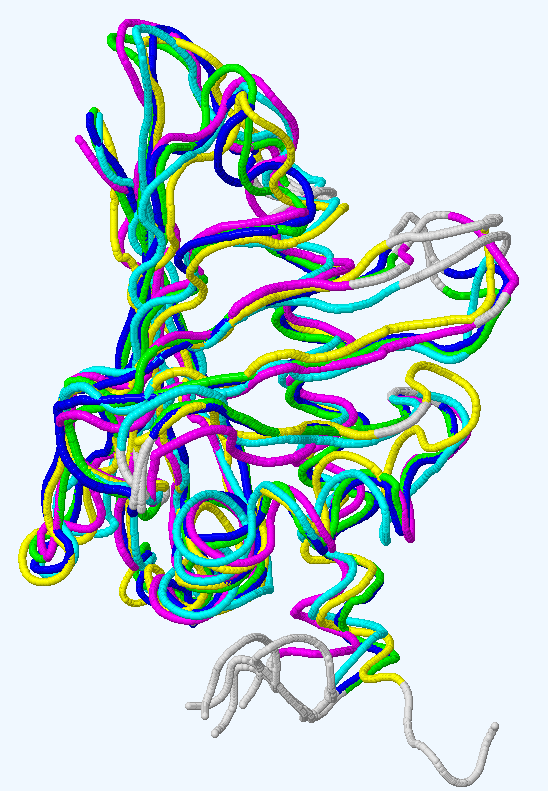
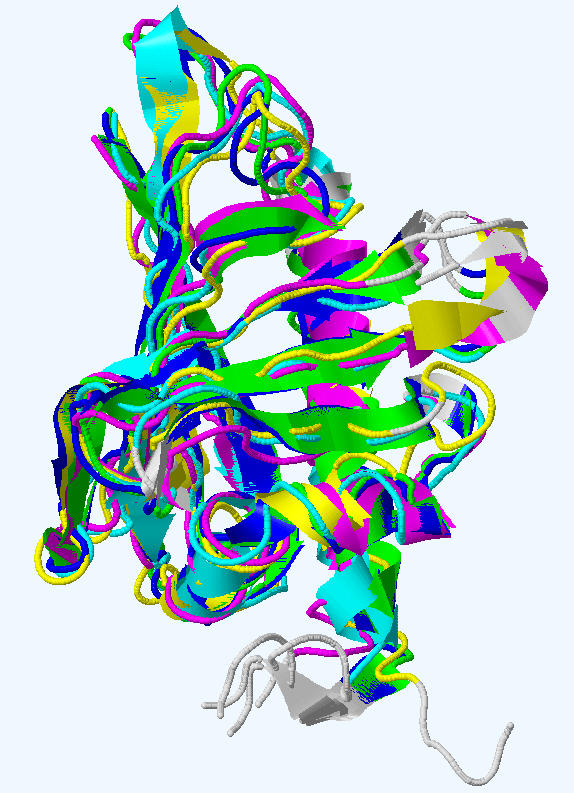
Рис. 1. Совмещение структур 1BV1 (зеленая цепь) со структурами гомологов: 5MMU (розовая), 1FM4 (синяя), 1Е09 (голубая) и 2LPX (желтая). Представлены изображения по trace (слева) и cartoon (справа).
Было построено множественное выравнивание полученных гомологов программой Muscle с параметрами по умолчанию. Часть выравнивания представлено на рис. 3 (полное выравнивание). Изображение части структурного выравнивания представлено на рис. 2.
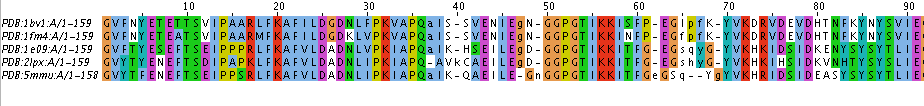
Рис. 2. Выравнивание по структуре (JalView).
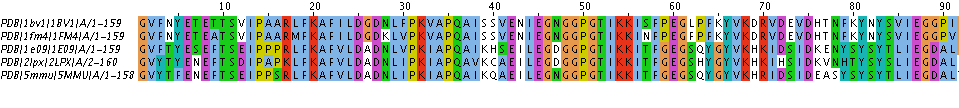
Рис. 3. Множественное выравнивание программой Muscle с настройками по умолчанию (JalView).
Видно, что в структурном выравнивании пристутсвуют гепы в позициях 37 и 40, а в выравнивании по последовательности этих гепов нет. Если посмотреть на эти остатки в структурном выравнивании, то становится понятно, что в структуре 2LPX действительно происходит смещение на одну аминокислоту в этом месте, поэтому 37 аланин по структуре не гомологичен остальным аланинам. Если смотреть только на выравнивание по последовательности, то этого не видно.
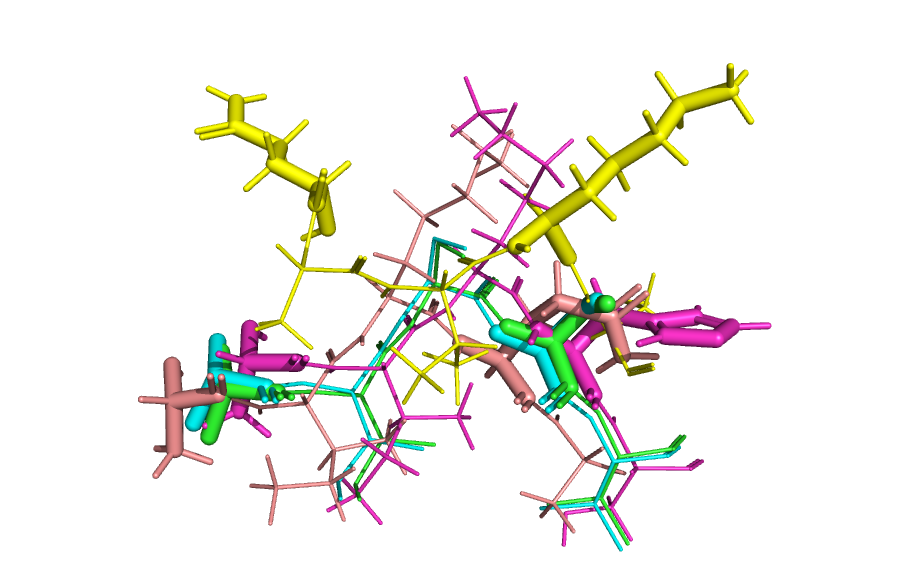
Рис. 4. Совмещение части структур 2LPX (желтая) со структурами гомологов: 5MMU (розовая), 1FM4 (синяя), 1Е09 (голубая) и 2LPX (желтая). 37 и 40 остатки (по выравниванию по последовательности) выделены sticks, а соседние остатки выделены lines.
Построение совмещения по заданному выравниванию
Для структуры 2f53 region d:115-191 константного домена T-клеточного рецептора из цепи альфа и для 2bnr region e:114-242 из цепи бета были построены карты бета-листов при помощи SheeP (рис. 5 и рис. 6). Для построения совмещения структур были выбраны С-альфа атомы консервативного цистеина (Cys136 для 2f53 и Cys143 для 2bnr) и спаренные с ним остатки. Структуры α-цепи и β-цепи довольно хорошо совместились.
Команды выранивания:
color orange, 2f53 and chain D and resi 115-191
color pink, 2bnr and chain E and resi 114-242
select alpha, 2f53 and chain D and resi 123-125+135-137+176-178 and name CA
select beta, 2bnr and chain E and resi 124-126+142-144+189-191 and name CA
pair_fit alpha, beta
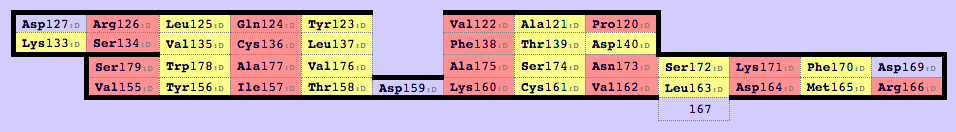
Рис. 5. Карта β-листов для α-цепи Т-клеточного рецептора (2f53 region d:115-191).
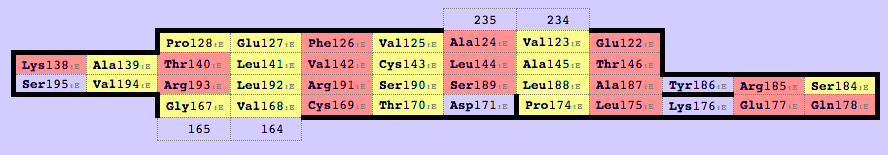
Рис. 6. Карта β-листов для β-цепи Т-клеточного рецептора (2bnr region e:114-242).
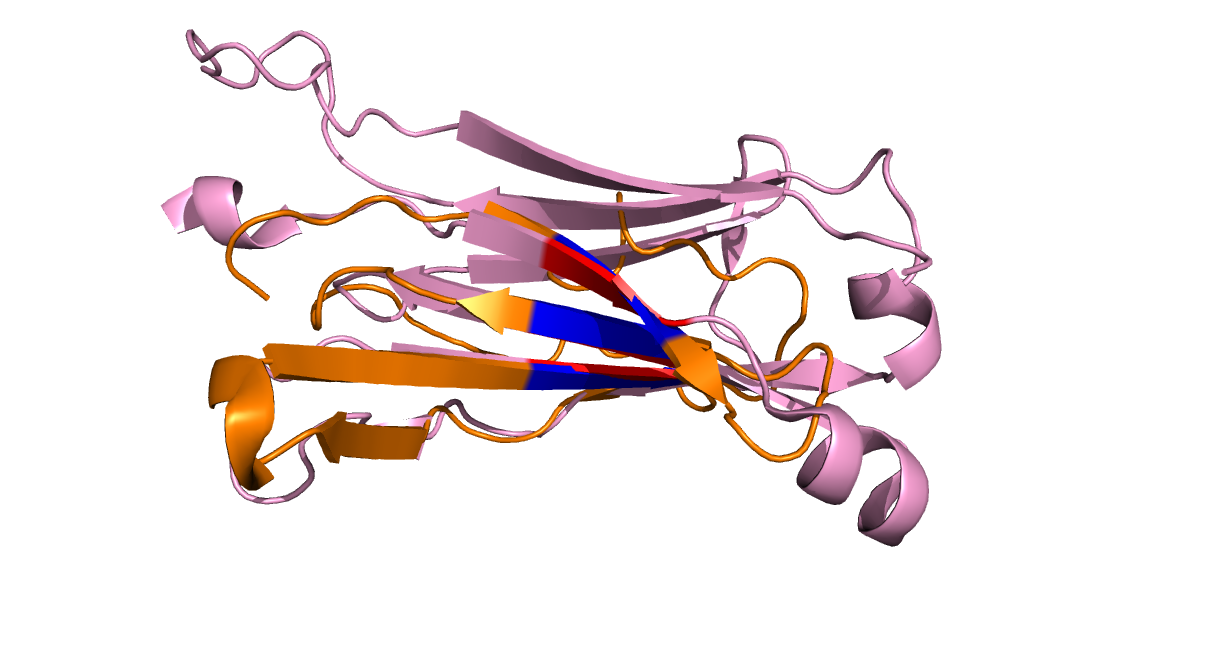
Рис. 7. Совмещение структур α-цепи (оранжевая) и β-цепи (розовая). Участки β-листов, использовавшиеся в качестве затравки выравнивания, выделены красным и синим цветом. RMS = 0.496 (9 to 9 atoms)