Геномное окружение. База данных STRING
В данном занятии я работал с информацией, которую может предоставить база данных STRING о белке, изучавшемся мною ранее - бета порфираназе из генома Zobellia galactanivorans. Самого белка в базе данных не оказалось, поэтому с помощью опции "search by protein sequence" был найден ближайший гомолог - GFO_1715 (гликозил гидролаза) из бактерии Gramella forsetii с e-value 6e-93.
На графе взаимодействий данного белка были показаны белки, находящиеся с данным на первом уровне близости (см. рис. 1). На рис. 2 изображена легенда изображения. На таблице 1 изображена краткая информация о функциях данных белков, взятая из базы KEGG. Исходя их этих данных получаем, что данные бекли не входят в один белковый комплекс, также она не являются белками, катализирующему последовательные реакции в организме, но, скорее всего, это группа белков, ответсвенных за крайне близкие функции в самом организме.
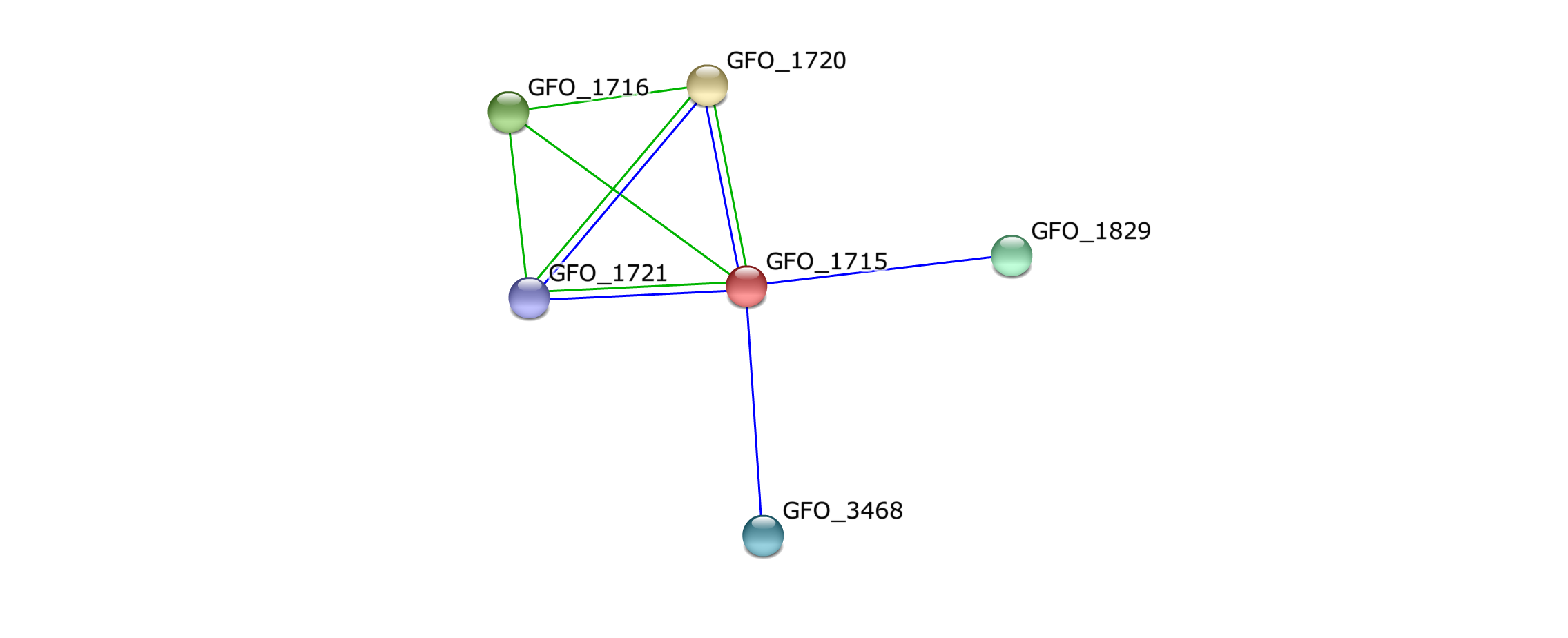
Рис. 1. Белки, находящиеся на первом уровне близости с белком GFO_1715. Зеленым обозначены связи Neighborhood - белки, входящие в консервативное окружение данного белка, синим - "Cooccurrence" - белки, встречающиеся с ним в одних организмах.

Рис. 2. Информация о белках
Таблица 1. Информация о белке GFO_1715 и его окружении.
| Идентификатор белка | KEGG | STRING |
| GFO_1715 | гипотетический белок | гликозил гидролаза |
| GFO_1720 | гипотетический белок | секретируемый белок |
| GFO_1716 | фитоноил-КОА диоксигеназа | фитоноил-КОА диоксигеназа |
| GFO_1829 | гликозил гидролаза | гликозил гидролаза |
| GFO_3468 | гликозил гидролаза | гликозил гидролаза |
| GFO_1721 | гипотетический белок | секретируемый белок, содержащий домен с неизвестной функцией |
Далее была получена информация о геномном окружении выбранного белка. На рис.3 приведено дерево организмов, в котором встречаются найденные белки с точностью до филума. Полностью раскрытое дерево - tree_all.png.
Часть найденных белков встречаются рядом только в одном организме - бактерии Gramella forsetii, откуда и взят исходный белок для анализа. Поэтому говорить о совместной встречаемости данных белков затруднительно. Можно заметить, что они выполняют схожую функцию, и, скорее всего, служат для выполнения одной и той же задачи, т.е их наличие рядом друг с другом оправдано. Хотя это может и являться лишь следствием того, что они являются результатами многократной дупликации первоначального гена. Сшивок между белками при этом не происходит. Интересно, что между участок, на котором они встречаются, делится на два отдельных группой из трех белков - GFO_1717 - алкоголь-дегидрогеназой, GFO_1718 - ацетилэстераза, GFO_1719 - гликозил гидролазой. Эти белки также есть на графе взаимодействия с GFO_1715, но не входят в первый уровень близости.

Рис. 3. Геномное окружение белка GFO_1715 с точностью до филума.
Далее был получен график совместной встречаемости гомологов найденных белков с точностью до филума (рис. 4.). Результаты позволяют предположить, что данный набор белков редко встречается среди организмов всех трех доменов , скорее всего, это можно объяснить необычностью субстратов данного белка (сложные углеводы красных водорослей - порфираны)(цвет "квадратика" показывает, наиболее близкого гомолога в данном таксоне, "полнота" собственно фигур до квадратов обозначает распространненость белка среди представителей данного таксона).

Рис. 4. График совместной встречаемости гомологов найденных белков с точностью до филума.
