RIP - Ribosome inactivating protein

Для работы был выбран домен PF00161. Данный домен имеет название RIP(Ribosomoe Activating Protein), входит в состав множества огранических ядов, которые действуют за счет остановки трансляции с рибосомы. К примеру, данный домен входит в состав известного белкового яда, добываемого из из клещевины - рицина.
Домен насчитывает 12 архитектур, содержащих суммарно 945 белковых последовательностей. Ссылка на список архитектур домена
Информация об архитектурах всех последовательностей, включающих данный домен представлена на первом листе (DOMAINS) в таблицы Excel. Данные были получены с помощью первого скрипта (swisspfam-to-xls.py), из данных к заданию.
Из базы данных Uniprot по идентификаторам были получены все последовательности, включающие домен RIP.
С помощью второго из данных скриптов (uniprot-to-taxonomy.py) были получены данные о таксономии этих последовательностей. Они приведены на той же странице (TAXONOMY) таблицы Excel.
С помощью Python из этих данных были составлены доменные архитектуры для каждой последовательности (ссылка на скрипт). Результат был добавлен в ту же таблицу Excel на страницу 3 (Architecture), где для каждого домена (столбцы) показана встречаемость в каждой последовательности (строки), а также в скобках длина каждого случая встречаемости. Колонка, соответствующая исходному домену, выделена зеленым.
В качестве исследуемого таксона был выбран таксон Cellular, в качестве подтаксонов - Eukaryota и Bacteria.
С помощью скрипта choose.py были выбраны только представители, входящие в таксоны Bacteria и Eukariota. Далее с помощью сервиса Muscle для них было построено выравнивание. Скачать выравнивание можно по этой ссылке
Построение филогенетического дерева последовательностей домена
Методом Maximum Likelihood было реконструировано эволюционное дерево данных белков - ссылка на структуру дерева в .nwk формате
Для подтверждения правильности построения дерева использовался bootstrap анализ с 100 репликами
Изображения полученного дерева и бутстрепного дерева приведены на рис. 1 и рис. 2
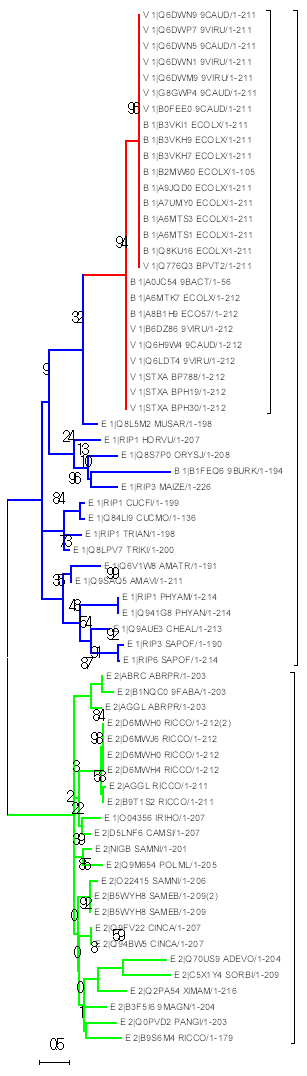
Дерево, построенное методом Maximum Likelihood

Бутстреп дерево для данной выборки
Дерево отчетливо делится на 3 множества - красным выделено множество прокариот и вирусов, имеющих первую доменную архитектуру, синим - второе множество, состоящее из эукариот, имеющих первую доменную архитектуру, зеленым - эукариоты, имеющие вторую доменную архитектуру.
Можно предложить следующий сценарий эволюции домена - изначально он имелся у бактерий с первой архитектурой. Использовался для подавления размножения других видов бактерий. В вирусы он попал за счет того, что лямбда-подобные фаги вырывают из генома хозяина большие части генома. В эукариоты этот домен попал во время эндосимбиоза, в следствии которого появились Viridiplantae. Далее у части (в том числе и у клещевины) он был включен в новую доменную архитектуру.
Построение профиля подсемейства и характеристика качества его работы
В качестве подсемейства были выбрана группа, выделенная на дереве зеленым цветом. По ним с помощью программы hmm2biuld был построен профиль последовательностей и затем он был откалиброван (программа hmm2calibrate).
По этому профилю был проведен поиск по всем белкам, содержащих RIP с помощью программы hmm2search без дополнительных параметров.
Далее с помощью скрипта create_ROC_and_EP.py были получены ROC-кривая (рис.3) и Enrichment plot (рис. 4) для модели, при поиске во всех последовательностях, содержащих домен RIP.
На их основании можно утверждать, что модель подходит для поиска белков данного подсемейства.
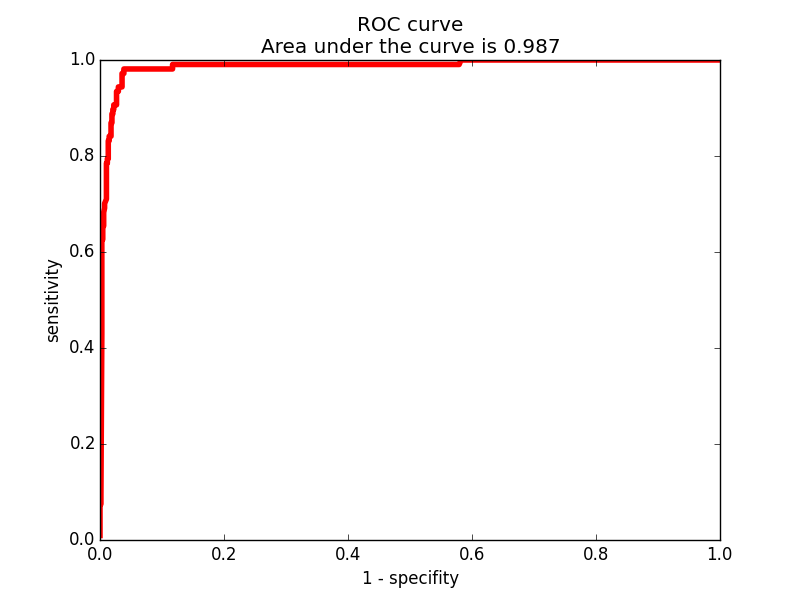
ROC-кривая для модели
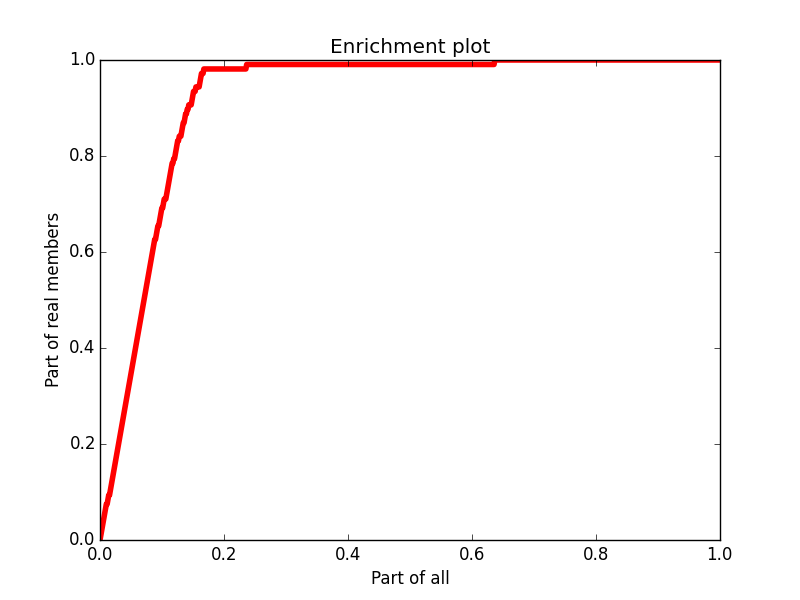
EP для модели
Тектовые отчеты построения ROC и EP
Более подробные результаты при пороге e-value 4.9e-49 приведены в таблице 1. В данном документе Excel представлены выполнения практикума.
На самом деле |
принадлежит подсемейству | не принадлежит | сумма |
| Выше порога по профилю | 106 | 78 | 184 |
| Ниже порога | 1 | 596 | 597 |
| сумма | 107 | 574 | 681 |