| Учебный сайт Саши Погорельской |

|
|
||||||
| Главная | Семестры | Скрипты | Обо мне | Ссылки | ||
|
|
Комплексы нуклеиновых кислот и белков Одним из способов предсказания вторичной структуры тРНК является испольхование программы einverted пакета EMBOSS. Данные, максимально близкие к настоящим (исходя из работы find_pair, см. предыдущее задание), получились при следующих параметрах работы программы: gap penalty 12, minimum score threshold 15, match score 3, mismatch score-4. Полученная информация прдеставлена в таблице 1. Другим методом прдесказания вторичной структуры является алгоритм Зукера. Он реализован в программе mfold. Я запускала программу 4 раза, чтобы получить описание, наиболее близкое данным find_pair, использованной в предыдущем практикуме. При этом я меняла процент возможного отличия вычисленной энергии от оптимальной структуры. По умолчанию, это число равно 5%, данные в таблице 1 представлены для P=20%. Плоская структура, полученная при помощи программы mfold прдеставлена на рисунке 1. Таблица 1.Данные предсказания вторичной структуры тРНК, полученные с помощью различных сервисов
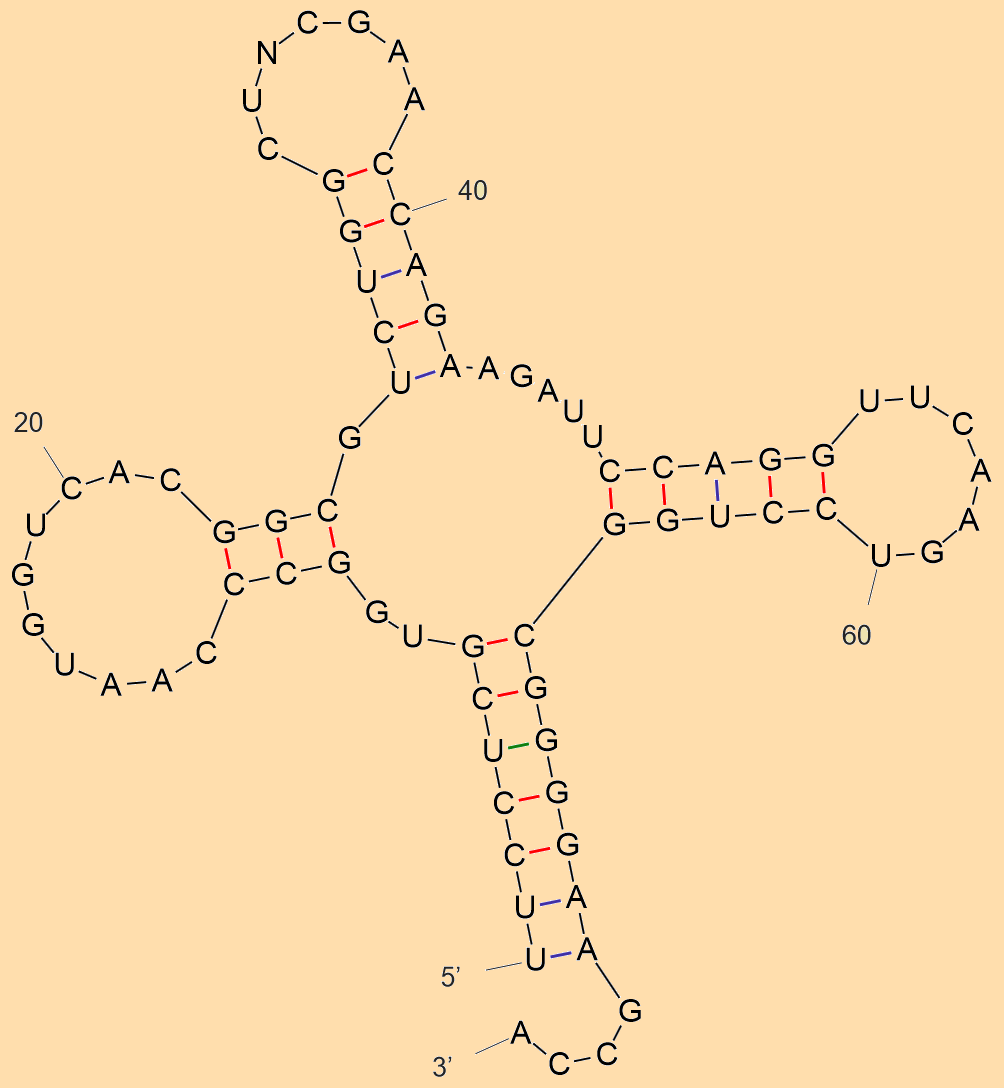
Рисунок 1. Предсказанная mfold вторичная структура транспортной РНК, идентифиатор PDB: 1F7U Дальше я буду работать с комплексом ДНК-белок, идентификатор pdb: 1TRO. Чтобы вспомнить как определяется наборы атомов в Jmol надо было написать скрипт, визуализующий различные атомы. Результаты представлены на рисунках 2-6, скрипт представляет их через паузы. 
Рисунок 2. Трехмерная структура комплекса ДНК-белок (иднтификатор pdb: 1TRO). Рисунок получен с помощью Jmol 
Рисунок 3. Трехмерная структура ДНК из комплекса ДНК-белок (иднтификатор pdb: 1TRO). Рисунок получен с помощью Jmol 
Рисунок 4. Трехмерная структура ДНК из комплекса ДНК-белок (иднтификатор pdb: 1TRO). Выделено множество атомов кислорода 2'-дезоксирибозы. Рисунок получен с помощью Jmol 
Рисунок 5. Трехмерная структура ДНК из комплекса ДНК-белок (иднтификатор pdb: 1TRO). Выделено множество атомов кислорода в остатке фосфорной кислоты. Рисунок получен с помощью Jmol 
Рисунок 6. Трехмерная структура ДНК из комплекса ДНК-белок (иднтификатор pdb: 1TRO). Выделено множество атомов азота в азотистых основаниях. Рисунок получен с помощью Jmol ЗАдавая другие группы атомов в Jmol я получила информацию о контактах ДНК и белка в комплексе. Критерием было расстояние между взаимодействующими атомами в 4,5 ангстрем. Данные представлены в таблице 2. Заметно, что контактов с атомами малой бороздки меньше всего. Возможно, это связано с тем, что объемным спиралям белка легче взаимодействовать с большой бороздкой ДНК, поэтому в процессе эволюции развивались именно эти контакты. Таблица 2.Данные о внутрикомплексных контактах ДНК и белка. Информация получена с помощью скрипта Jmol
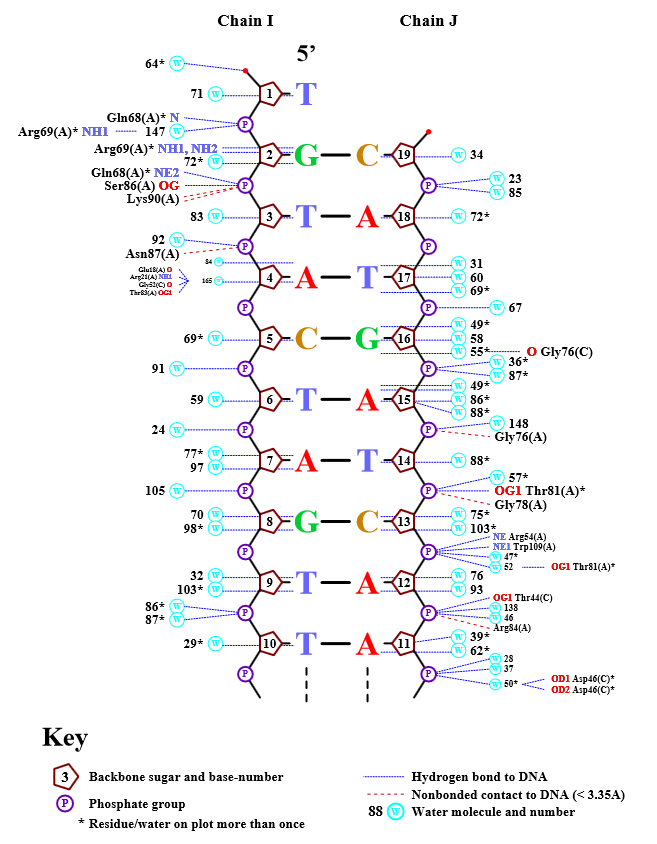
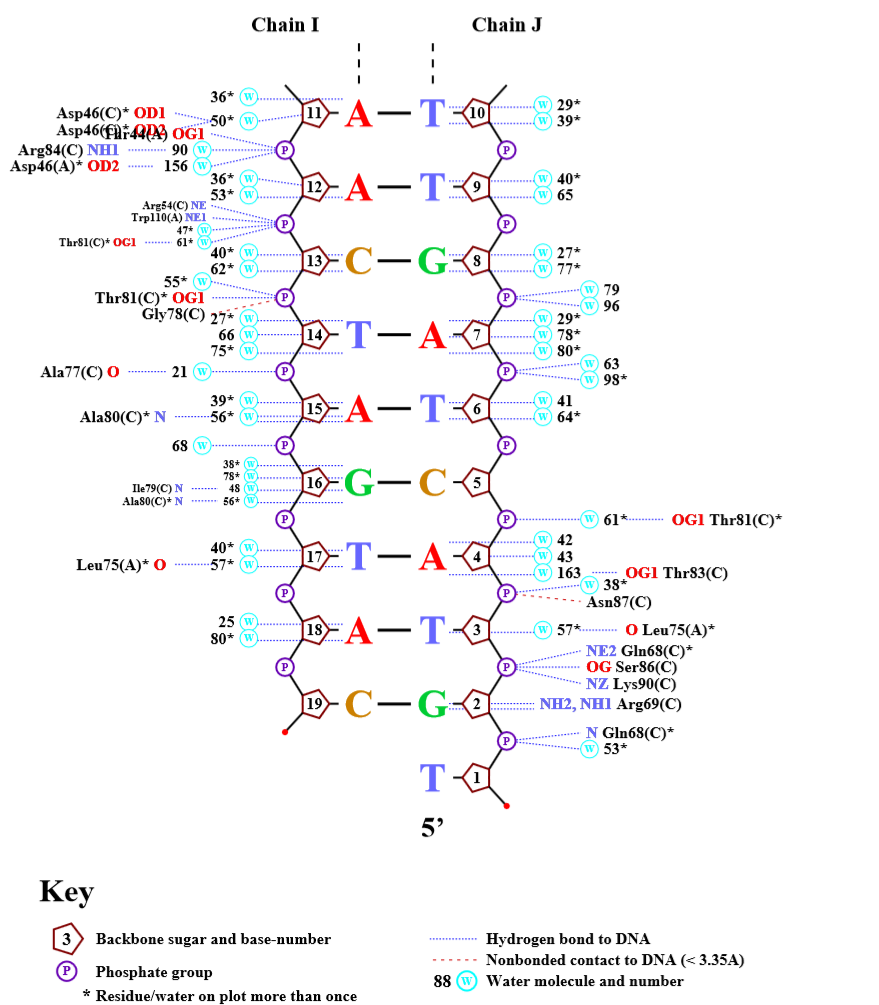
Рисунок 7. Популярная схема ДНК-белковых контактов в комплексе (иднтификатор pdb: 1TRO). Рисунок получен с помощью программы nucplot Больше всего на схеме контактов ДНК и аспарагиновой кислоты (Asp46 цепи С) - 5. Поэтому, на мой взгляд, это самый важный аминокислотный остаток для распознавания ДНК. 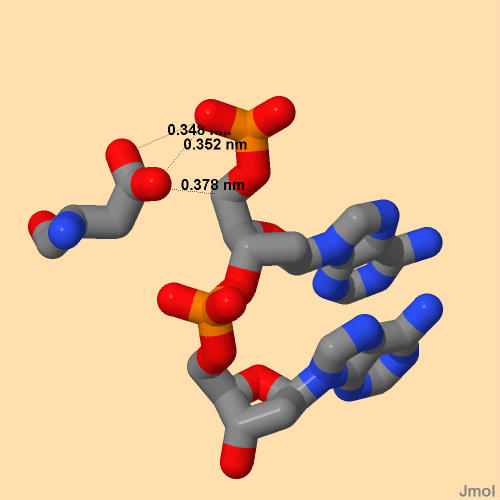
Рисунок 8. Asp46(цепочка C) и 2 аденина (11 и 12 цепочки J). Пунктиром показаны контакты белка и ДНК, показана длина связи. Рисунок получен с помощью Jmol |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| © Pogorelskaya Sasha | Last modification date: 19.02.15 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||