| Учебный сайт Саши Погорельской |

|
|
||||||
| Главная | Семестры | Скрипты | Обо мне | Ссылки | ||
|
|
A-, В- и Z- формы ДНК. Структура РНК С помощью программы fiber (пакета 3DNA на сервере kodomo) можно построить различные формы молекул ДНК с заданной последовательностью или повторяющейся последовательностью. pdb-файлы, созданные данной программой можно скачать здесь: A-форма, B-форма, Z-форма. Одна из нитей ДНК в них включает в себя 5 раз повторенную последовательность "GATC". 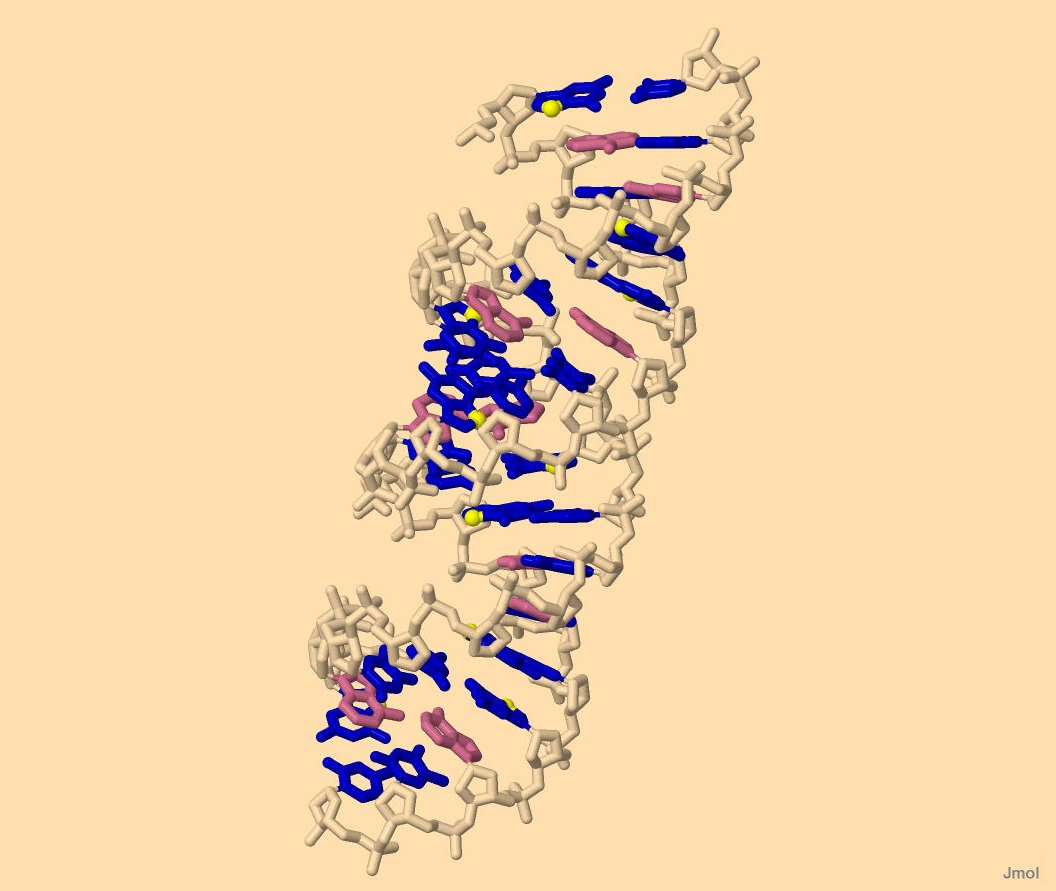
Рисунок 1. Трехмерная структура A-формы ДНК.Азотистые основания покрашены в синий цвет, кроме аденина (розовый), атомы N7 гуанина покрашен в желтый. Изображение получено с помощью Jmol Одним из средств визуального представления трехмерных структур является Jmol, использовавшийся в предыдущем семестре. Изображение, полученное с помощью этой программы представлено на рисунке 1. Для A-формы ДНК представлена разная покраска по отомам сахаро-фосфатного остова (светло-желтый цвет), азотистых оснований (синий для гуанина, цитозина и тимина и розовый для аденина) и атомов N7 гуанина (желтый). Исходным был файл, полученный ранее. При этом использовались следующие команды:
Для выполнения следующего задания мне нужно было скачать с сайта PDB структуры с идентификаторами 1f7u и 1tro, включающие комплекс балка и транспортной РНК и ДНК-белковый комплекс соответственно. Чтобы проверить целостность структур нуклеиновых кислот в этих файлах, я представила в Jmol их сахаро-фосфатный остов в ленточной модели. Эти изображения приведены на рисунках 2 и 3. 
Рисунок 2. Трехмерная структура сахаро-фосфатного остова транспортной РНК, идентифиатор PDB: 1F7U.Изображение получено с помощью Jmol Использованные команды:

Рисунок 3. Трехмерная структура сахаро-фосфатного остова ДНК из ДНК-белкового комплекса, идентифиатор PDB: 1tro. Изображение получено с помощью Jmol Использованные команды:
Из рисунка 2 видно, что структура транспортной РНК не содержит разрывов в скелете цепи, а ДНК содержит. Вероятнее всего это объясняется тем, что эта структура составлена из двух практически аналогичных частей белка, объединяющихся в комплекс. По рисунку 3 заметно, что разрыв очень легко устраняется, значит, это может одна молекула (без разрыва). Возможно, это не было просчитано при создании этого файла. Следующим заданием было определние большой и малой бороздки ДНК и расположение атомов азотистых оснований относительно них. Изобржание 32 цитозина для B-формы представлено на рисунке 4. На рисунке 5 тот же фрагмент ДНК немного повернут. Заметно, что в сторону малой бороздки обращен только атом кислорода O2, а в сторону большой - N4 и C5. Остальные атомы располагаются в плоскости между бороздками. Плоская структура цитозина с покрашенными атомами представлена на рисунке 6.
Использованные команды (с точностью до поворота):
В A-форме все атомы цитозина находятся в большой бороздке, кроме N4 - на границе, но направлен он в сторону малой бороздки. В Z-форме все атомы в большой бороздке. Таблица 1.Сравнение свойств различных форм ДНК. Измерения произведены с помощью Jmol в структурах, полученных в начале занятия
Таблица 2.Сравнение торсионных углов цитозина (32 цитозин, изображенный на рисунках 4-5) различных форм ДНК. Измерения произведены с помощью Jmol в структурах, полученных в начале занятия
Также я сравнила значения торсионных углов различных азотистых оснований для молекулы ДНК из ДНК-белкового комплекса (идентификатор PDB: 1TRO) и транспортной РНК (идентификатор PDB: 1F7U). Скачать таблицу с этими данными можно здесь. В таблице 3 приведены значения торсионных углов в тРНК. При это были удалены азотистые основания, для которых нельзя посчитать углы (крайние в цепи и в спирали для РНК). Основания с наибольшими отклонениями торсионных углов от средних выделены желтым цветом. В наибольшей степени это характерно для цитозина и гуанина. Таблица 2.Сравнение торсионных углов нуклеотидов тРНК (идентификатор PDB: 1F7U). Удалены нуклеотиды, для которых нельзя посчитать углы, нижняя строка - средние значения по обеим цепям. 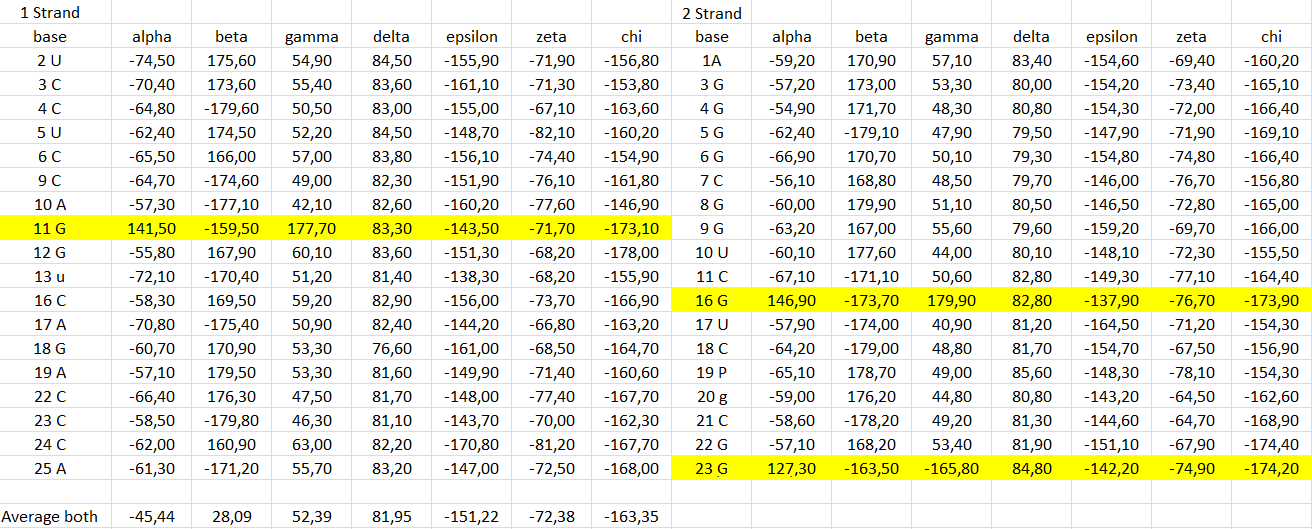
Вторичная структура транспортной РНК содержит несколько шпилек, одна из них представлена на рисунке 7. Она образована атомами 928-931 нуклеотидов и после поворота цепи 939-942. Все шпильки, входящие в состав этой тРНК прдеставлены на рисунке 8, обведены в рамочки. 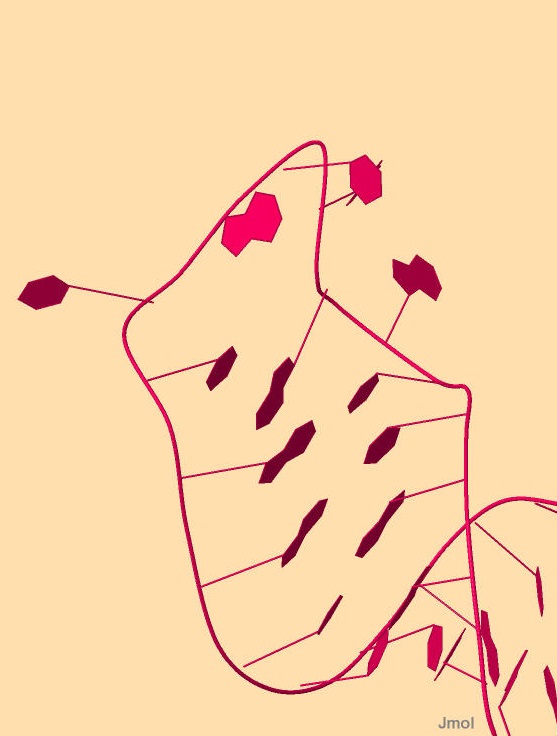
Рисунок 7. Одна из шпилек транспортной РНК. Идентификатор PDB: 1F7U. Изображение получено с помощью Jmol 
Рисунок 8. Шпильки транспортной РНК. Идентификатор PDB: 1F7U. Данные получены с помощью программы find_pair и analyze На рисунке 8 можно также увидеть присутствие неканонических пар оснований. Отдельно они подчеркнуты на рисунке 9. 
Рисунок 9. Неканонические пары транспортной РНК. Идентификатор PDB: 1F7U. Данные получены с помощью программы find_pair и analyze Помимо водородных связей в шпильках есть одиночные связи между основаниями для поддержания пространственной структуры. Пары, образующие такие связи представлены на рисунке 10, они подчеркнуты. 
Рисунок 10. Дополнительные водородные связи в транспортной РНК. Идентификатор PDB: 1F7U. Данные получены с помощью программы find_pair и analyze В выходном файле программы analyze также есть информация о величине стекинговых взаимодействий, она прдеставлена на рисунке 11. При этом пары с самым сильным перекрыванием подчеркунты карсным, а с самым слабым - черным. Стандартное изображение 7 структуры на рисунке 11 прдеставлено на рисунке 12. 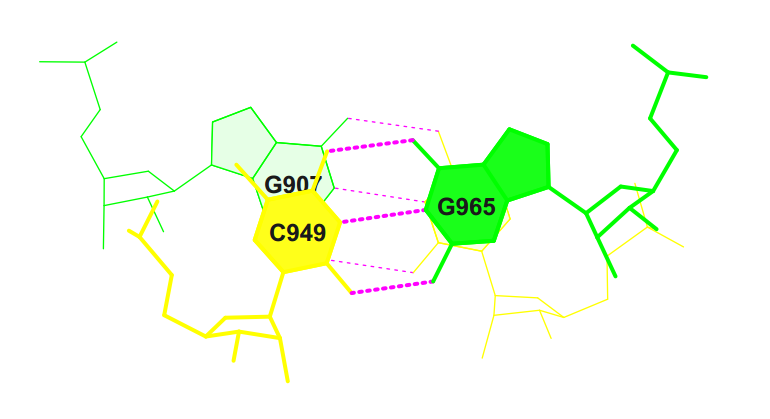
Рисунок 11. Стандартное изображение стекинг-взаимодействия в тРНК. Идентификатор PDB: 1F7U. Данные получены с помощью программы find_pair и analyze |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| © Pogorelskaya Sasha | Last modification date: 19.02.15 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||