Предсказание структур амилоидов с помощью AlphaFold2
В этом практикуме мы работаем с AlphaFold2 с помощью ColabFold. Наша задача - исследовать, насколько хорошо AlphaFold2 справляется с предсказанием структур амилоидов - агрегатов из белков, образующих собой как бы один бета-лист. Накопление в организме различных амилоидных фибрилл связывают со многими заболеваниями, в том числе с болезнью Альцгеймера и болезнью Паркинсона.
В качестве примера мы будем предсказывать структуру мономера и мультимеров фрагмента альфа-синуклеина. Этот белок из 140 аминокислот взаимодействует с фосфолипидами и в растворе обычно считается внутренне неупорядоченным (intrinsically disordered). Он особенно активно накапливается в ядрах нейронов, а также в пресинаптических окончаниях, где, по-видимому, участвует в заякоривании в мембране SNARE-комплекса, осуществляющего "сливание" везикул с нейротрансмиттером с мембраной и, как следствие, высвобождение их содержимого в пресинаптическую щель. За связывание липидов отвечает N-концевой участок данного белка (примерно с 1 по 60 остатки), а за взаимодействие с другими белками - C-конец (остатки 96-140).
Альфа-синуклеин интересует нас потому, что разные молекулы данного белка могут агрегировать, переходя в бета-форму. Непосредственно участвует в данном процессе, собственно, гидрофобный участок данного белка, расположенный между описанными выше N-концевым и C-концевым (т.е. примерно с 60 по 95 остатки). Показана связь накопления фибрилл альфа-синуклеина с болезнью Паркинсона, мультисистемной атрофией и другими заболеваниями - в том числе, в некоторых случаях, с болезнью Альцгеймера1.
Для предсказания структуры с помощью AlphaFold2 мы будем использовать фрагмент белка с 38 по 95 остатки. На рис. 1 данный участок выделен в "нормальной" форме альфа-синуклеина, связанного с мицеллой.
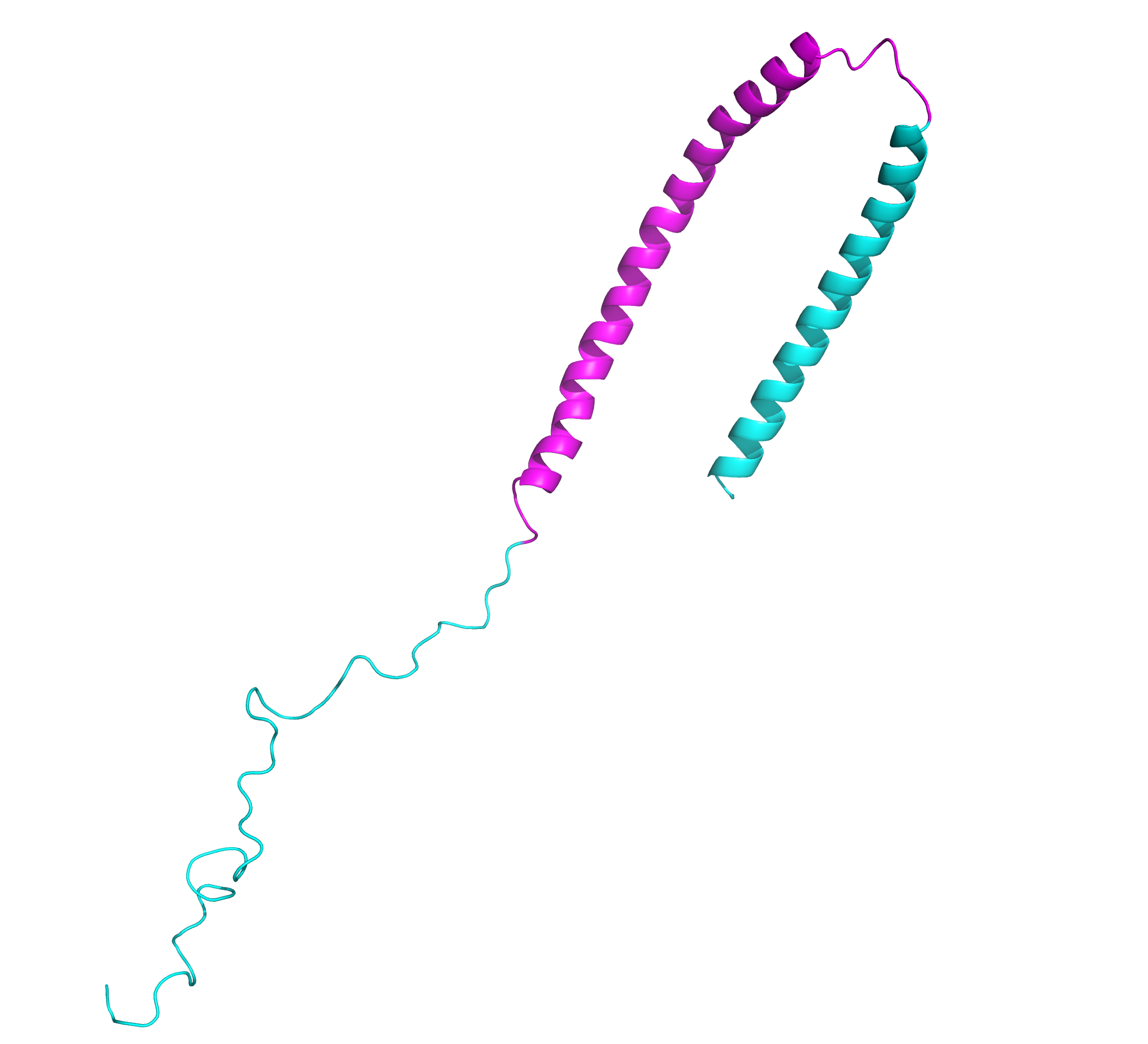
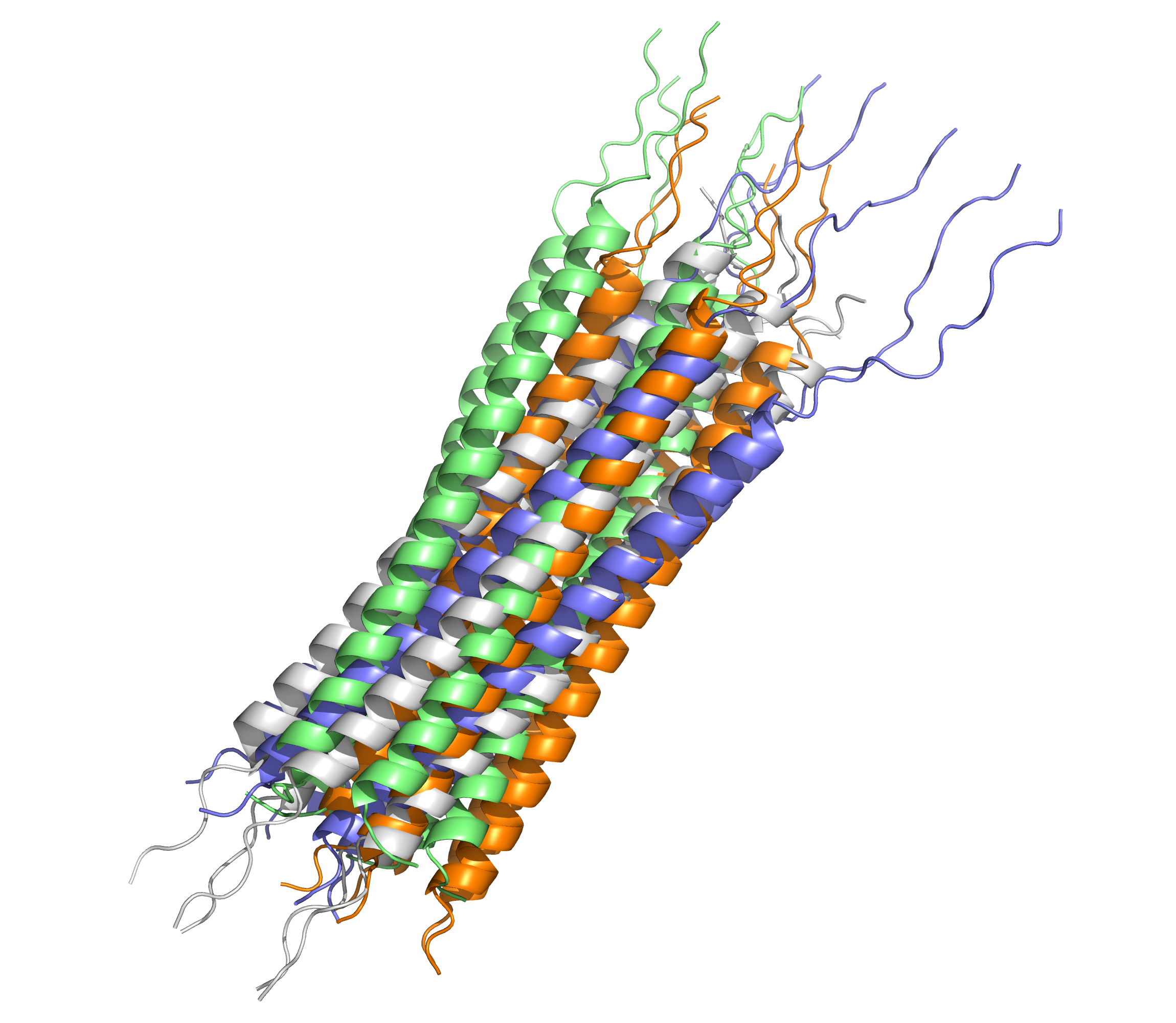
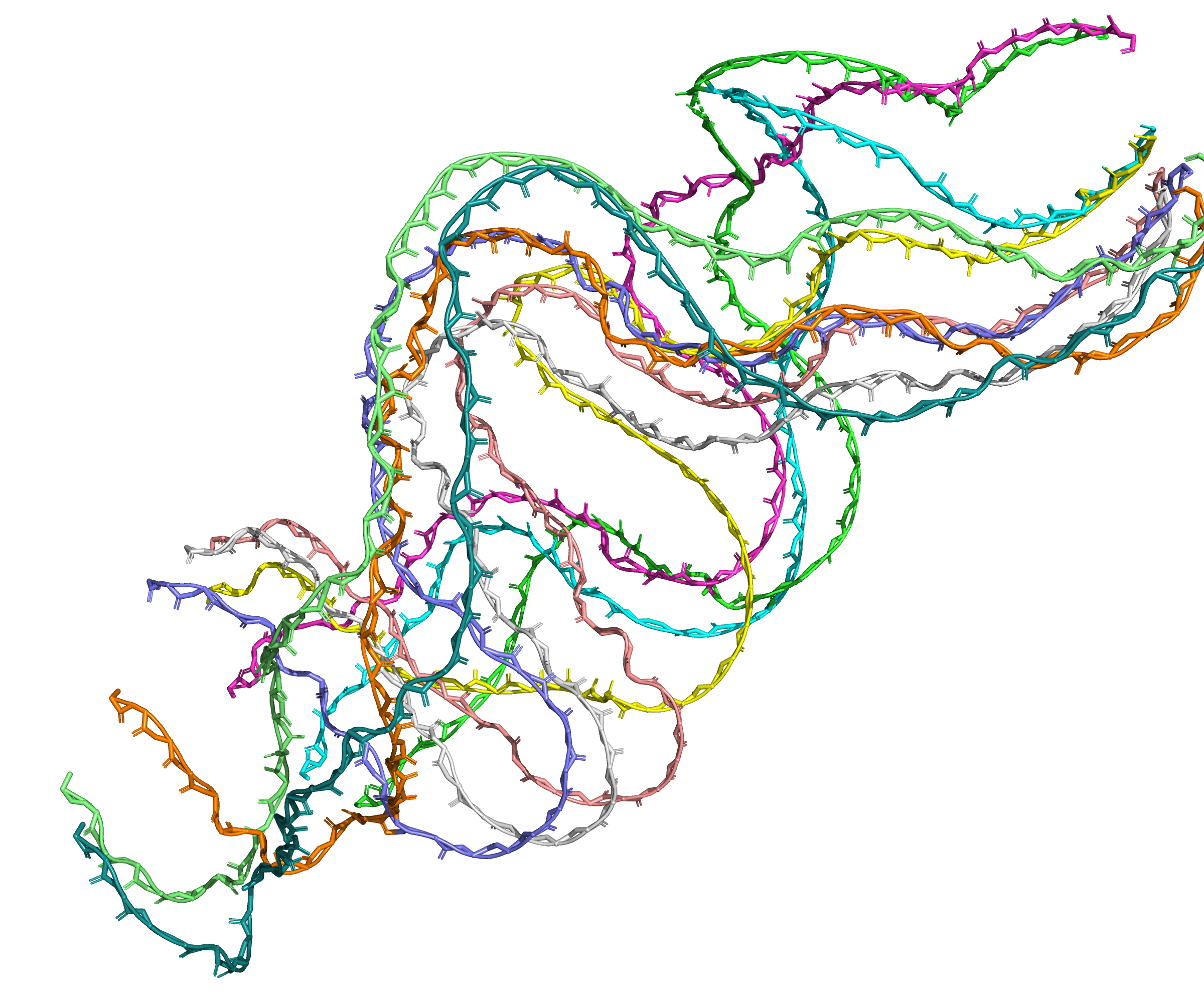
То, что остатки именно в этом участке белка формируют амилоидные структуры, подтверждает структура фибриллы на рис. 2.

Предсказание структуры мономера
AlphaFold2 построил пять моделей для мономера данного фрагмента альфа-синуклеина. Три лучших из них по метрикам качества AlphaFold2 представляют собой просто длинную спираль с небольшими неупорядоченными участками на концах, а худшие две - два спиральных участка, соединенных петлей.
Сравнив эти результаты с данными о структуре данного фрагмента из PDB (структура 1XQ8, см. выше), обнаруживаем, что наибольшую схожесть имеет третья по рангу модель AlphaFold2 (см. рис. 1). Поскольку данный белок часто считают внутренне неупорядоченным, что повышает вероятность, что полученная ЯМР структура 1XQ8 может быть далеко не единственной возможной формой даже в норме, вполне возможно, что все три модели AlphaFold2 без петли внутри в норме могут обнаруживаться в клетках. Впрочем, оснований утверждать, что это обязательно так, у нас нет.
Предсказание структуры мультимера
Надо сказать, что сам по себе AlphaFold2 не создавался с задачей хорошо предсказывать структуру белок-белковых комплексов и вообще многокомпонентных мультимеров. В ColabFold для работы с мультимерами реализованы две модификации AlphaFold2: AlphaFold-Multimer и AlphaFold2-ptm. Если первая из них - действительно отдельная модификация2, то второй вариант - это просто сам AlphaFold2 с нестандартными параметрами. Мы попробовали с помощью обоих этих алгоритмов предсказать структуру мультимеров альфа-синуклеина из пяти и десяти мономеров.
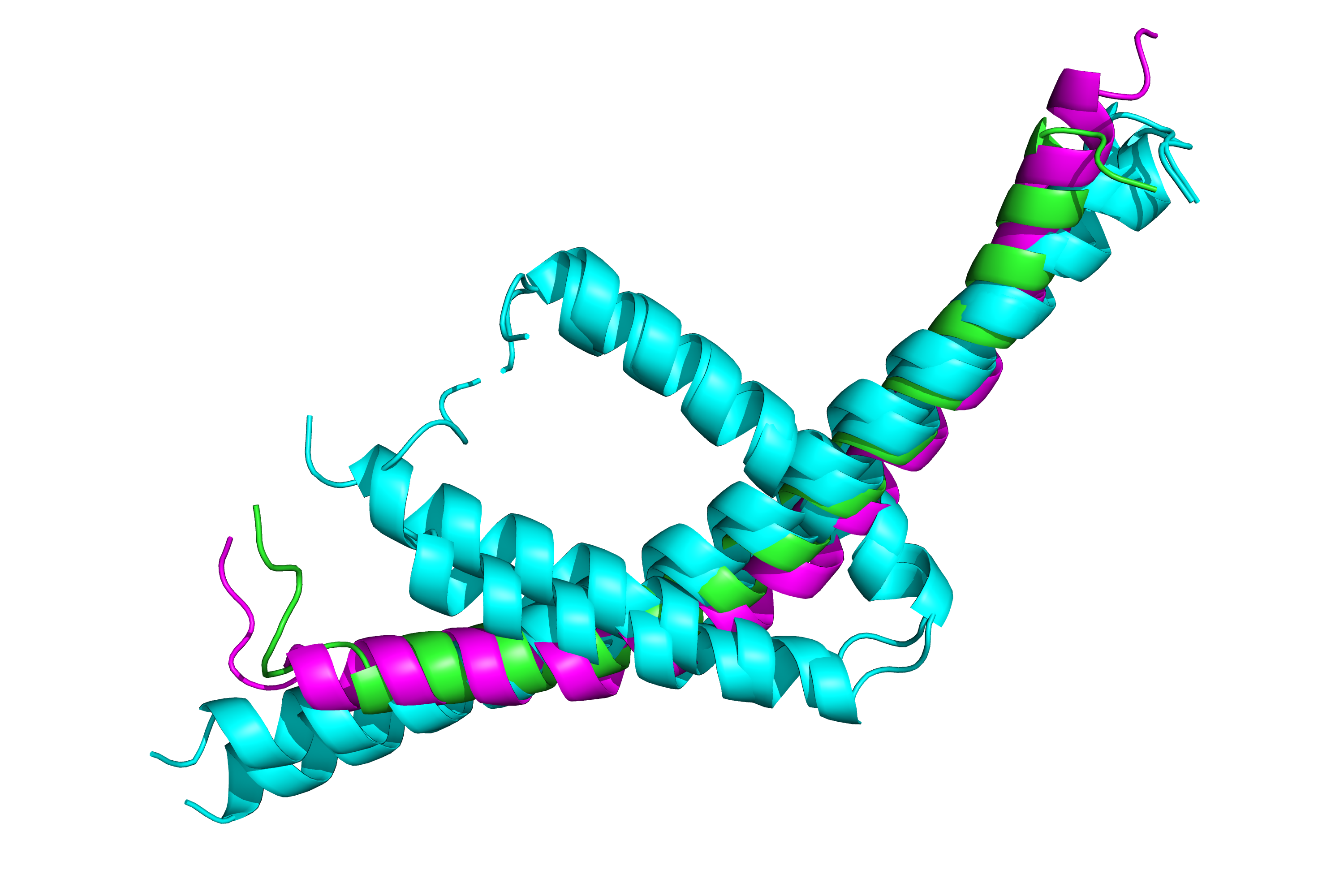
AlphaFold-Multimer, который является вариантом по умолчанию для комплексов в ColabFold, очень плохо справился со своей задачей. Что для пяти, что для десяти мономеров, он просто расположил спирали друг рядом с другом, при этом разные белки еще и "наползли" друг на друга (рис. 3-5).

Это довольно странно, что у AlphaFold2-Multimer возникли настолько серьезные проблемы с предсказанием, поскольку авторы статьи, где предложен данный алгоритм, как раз-таки специально заботились о лоссе в связи с наличием нескольких белков:
AlphaFold uses a Frame Aligned Point Error (FAPE) loss, whereby the distances between ground truth and predicted atoms are computed in the local reference frame of every residue. In AlphaFold, this loss was clamped at 10 Å. For training the multimer model we made changes to the loss function used: for the intra-chain amino acid pairs of the complex we keep the same 10 Å clamping; for the inter-chain pairs we use an unclamped FAPE loss. This provides a better gradient signal for incorrect interfaces. Moreover, we add extra positional encodings denoting whether a given pair of amino acids corresponds to different chains and whether they belong to different homomer or heteromer chains. 2
Возможно, проблема не с самим AlphaFold2-Multimer, а с его реализацией в ColabFold, поскольку туда он был добавлен только в ноябре 2021.
Нельзя не сказать, что проблема обучения моделей для структур из нескольких белков и сложности, возникающие в том числе из-за возможного перекрывания разных составных частей - по-видимому, актуальная проблема в структурной биоинформатике, и авторы опубликованной в ноябре 2021 модели для докинга белков друг с другом разрабатывали целый отдельный сложно устроенный лосс для этих целей3.
Тем не менее, если мы попробуем использоваться AlphaFold2-ptm, т.е. просто с AlphaFold2, запущенным в ColabFold с особенными параметрами для работы с комплексами, мы увидим куда более радостную картину.
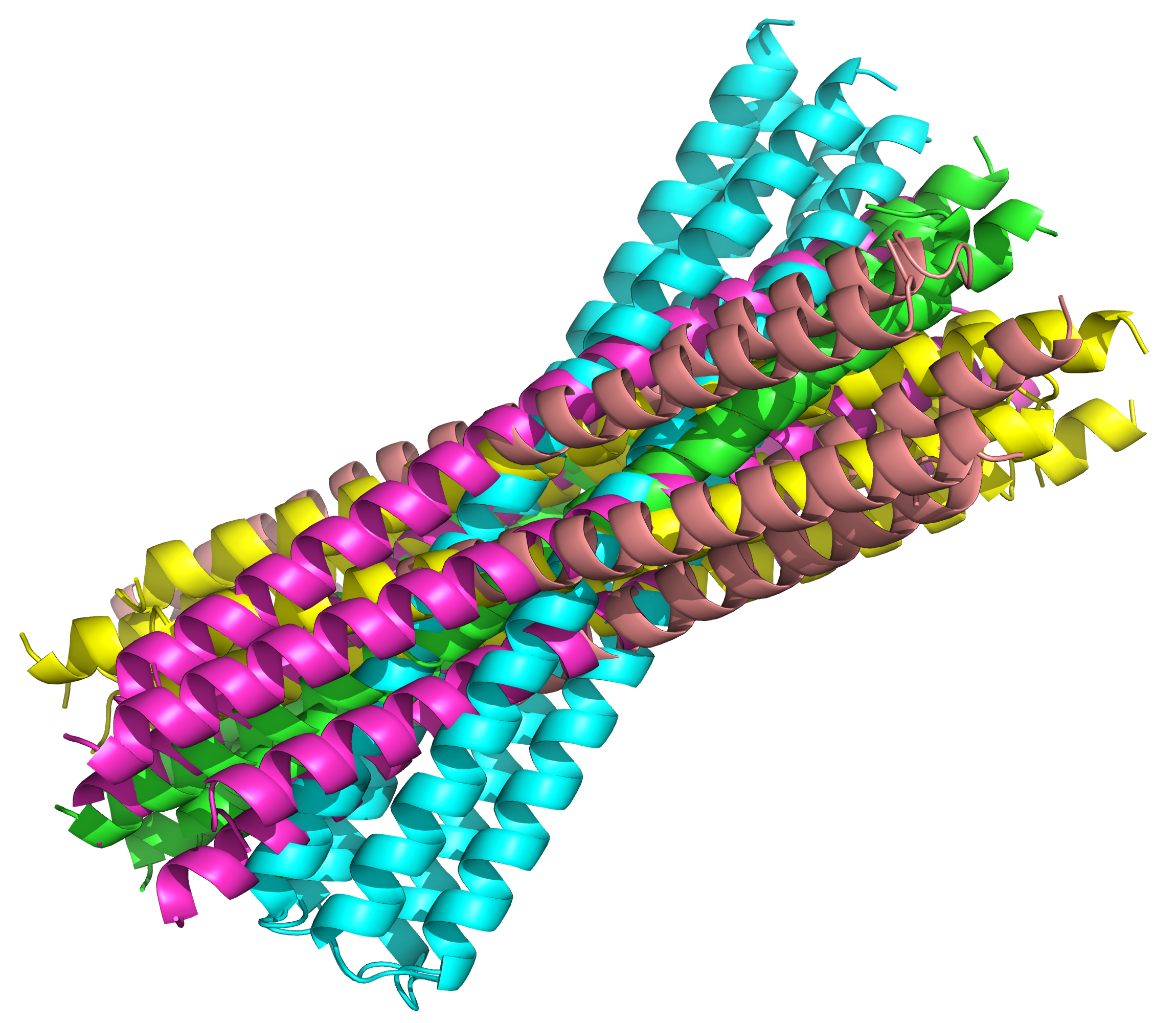
Для мультимера из пяти мономеров четыре из пяти моделей будут просто состоять из расположенных рядом спиральных белков, т.е. примерно тот же результат, что и у AlphaFold2-Multimer, но хотя бы без перекрываний (см. рис. 6).
При этом одна из моделей, хоть и обладающая самым низким рангом, будет представлять собой что-то более интересное (см. рис. 7): здесь AlphaFold2 не разметил вторичную структуру и расположил белки рядом друг с другом - это уже отдаленно может напоминать бета-лист из этих белков (пусть и очень уж отдаленно).
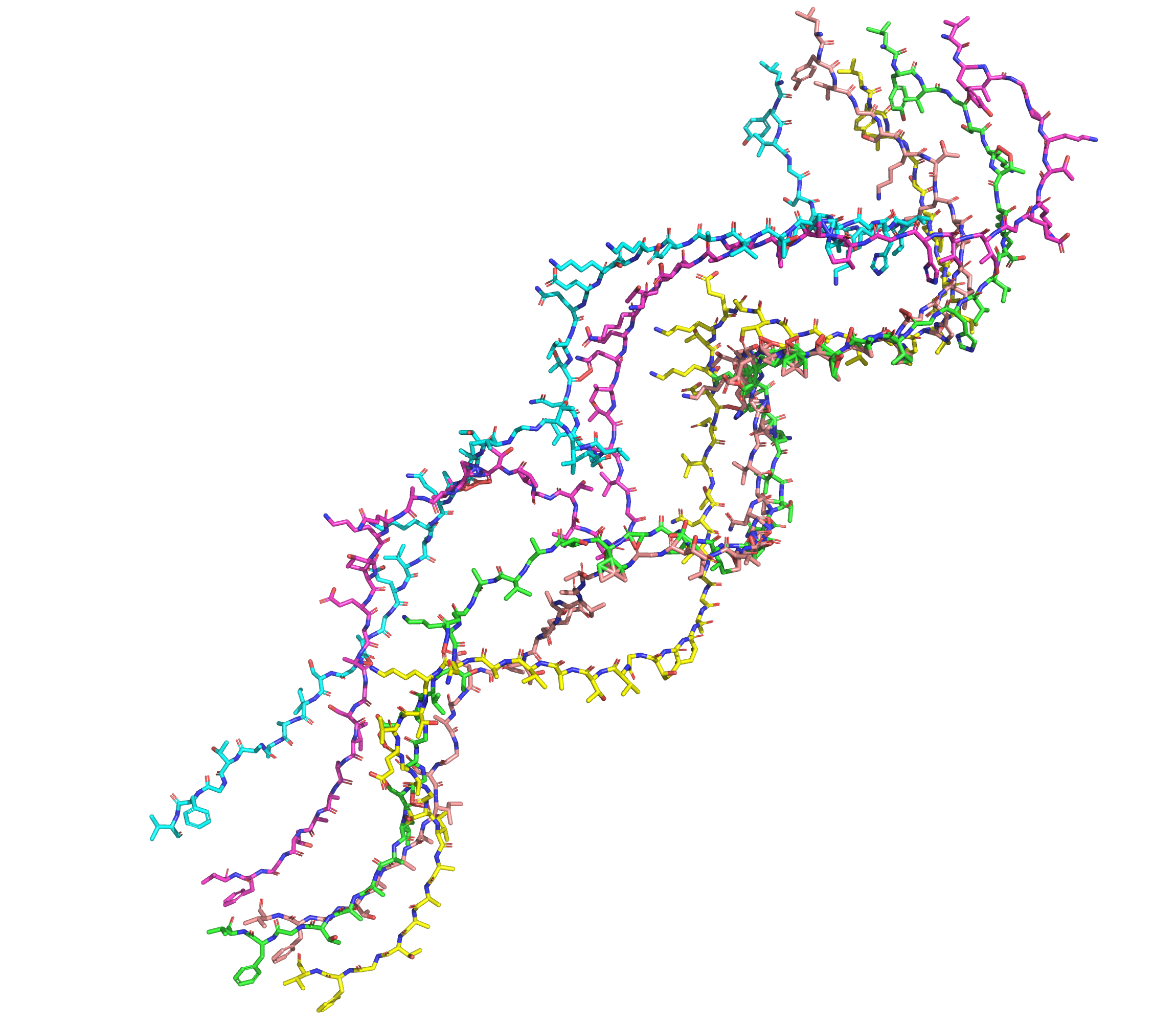
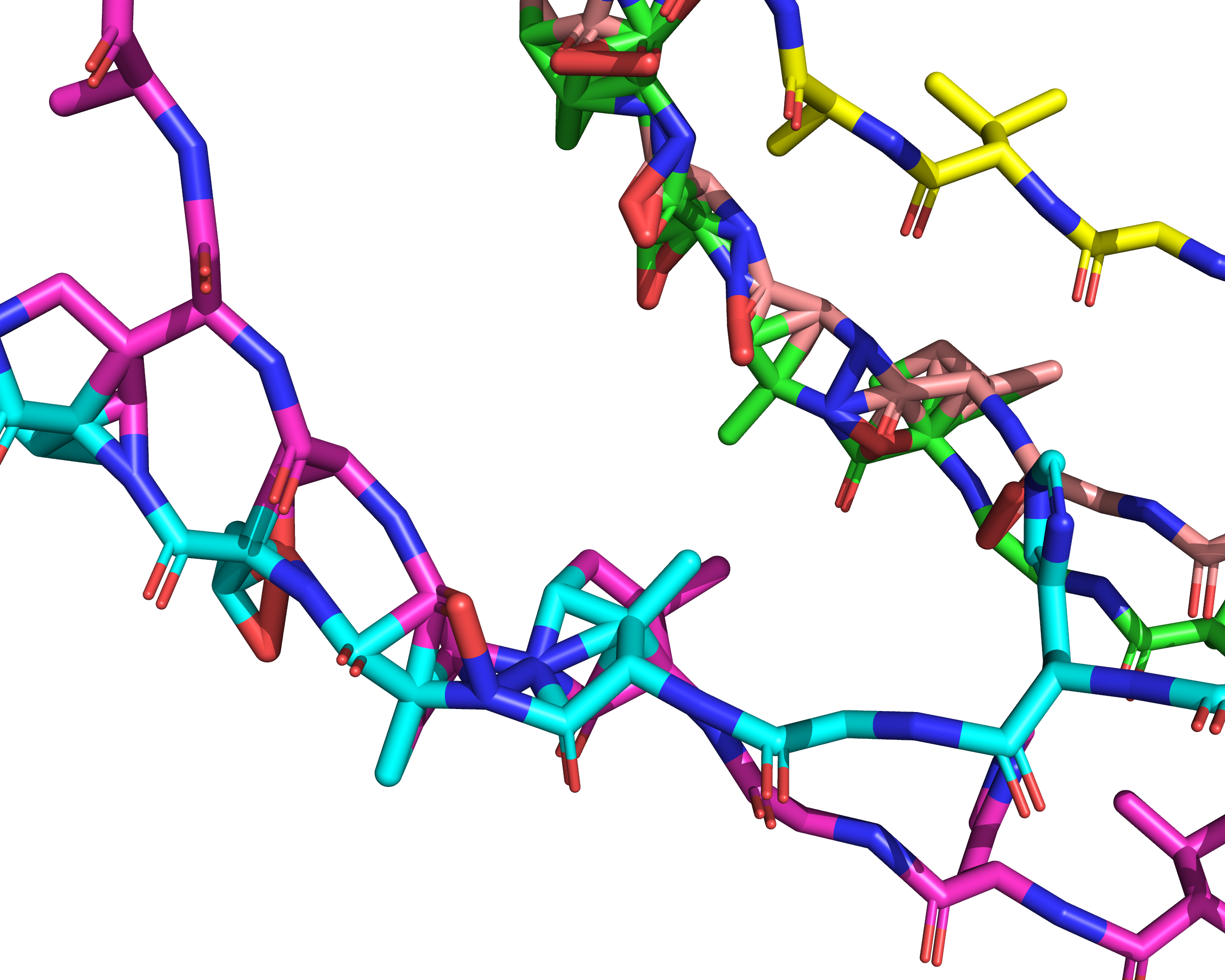
Тем не менее, в этой модели AlphaFold2-ptm уже есть перекрывание и даже будто бы ковалентные связи между разными цепями (рис. 8). А значит наша гипотеза о том, что перекрывание - исключительно баг AlphaFold2-Multimer, не верна, и это какая-то системная проблема.
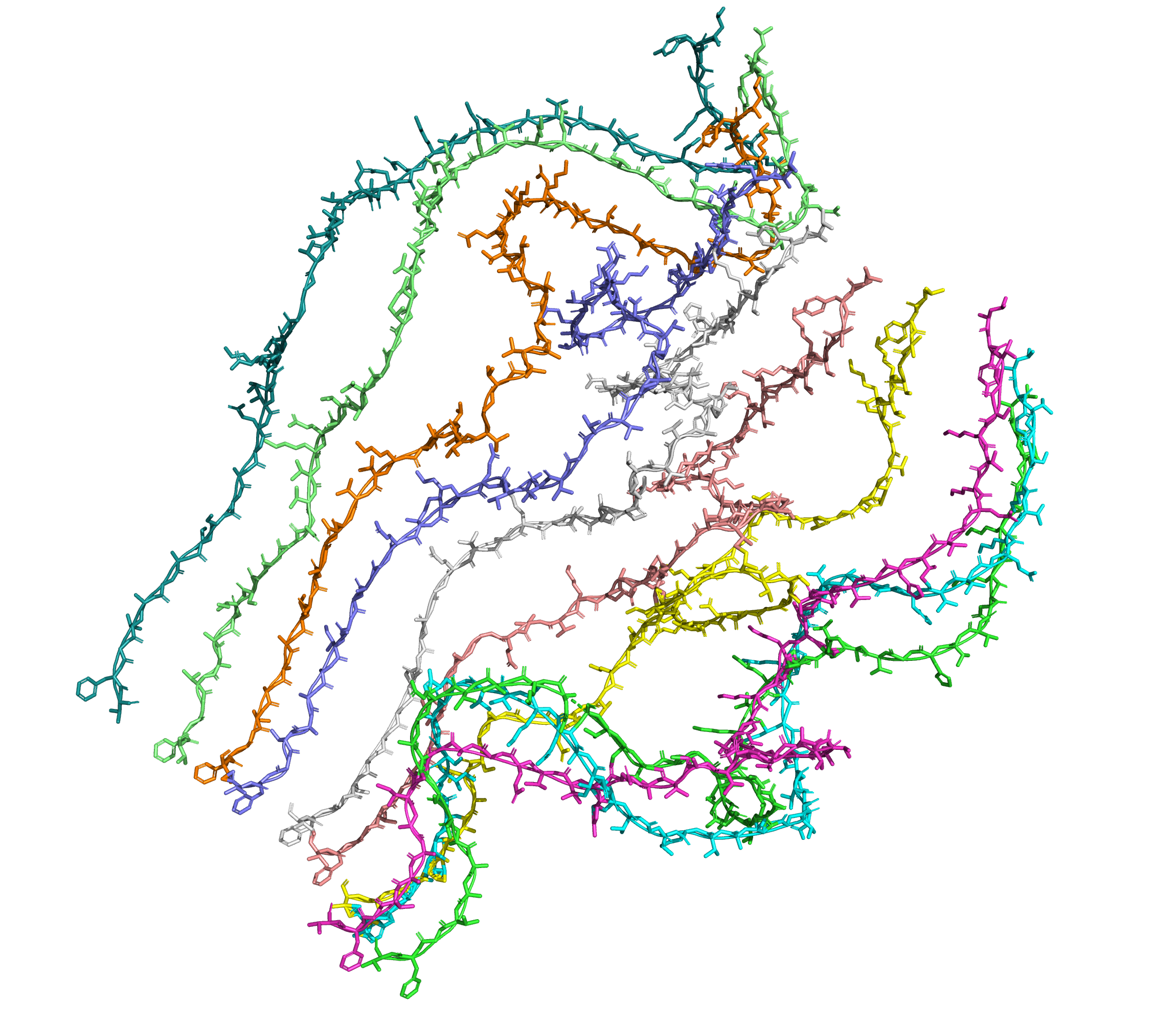
В случае мультимера из 10 мономеров только две модели представляют собой сгруппированные спиральные белки. Так, модель с рангом 2 даже по автоматической разметке вторичной структуре предполагает наличие небольшого бета-листового участка (рис. 9).

Вторичная структура моделей с рангом 4 и 5 никак не размечена, однако и они в целом похожи на бета-листы из белков (см. рис. 10-12).

В целом, можно заключить, что предсказание структур мультимеров - задача решенная пока что в еще меньшей степени, чем просто предсказание структур белков, а алгоритм AlphaFold2-Multimer и, в некоторых случаях, AlphaFold2-ptm либо не учитывают совершенно необходимых ограничений на расположение компонент комплекса сам по себе, либо плохо реализованы в ColabFold. Тем не менее, способностью "угадывать" возможность взаимодействий, в том числе бета-листов, между разными белками, AlphaFold2 обладает. Чтобы сказать, следует ли из этого, что с помощью AlphaFold2 можно предсказывать, является ли белок амилоидным, нужно как минимум сделать контрольные "эксперименты" и запустить AlphaFold2-ptm с не-амилоидными белками с целью проверить, не будет ли он их тоже располагать вытянутыми друг рядом с другом, как он это сделал для альфа-синуклеина, однако потенциально это можно попробовать сделать и, может быть, даже получится показать, в каких случаях AlphaFold2-ptm позволяет предсказывать "амилоидность".
Список литературы
1. Chiti F, Dobson CM. Protein Misfolding, Amyloid Formation, and Human Disease: A Summary of Progress Over the Last Decade. Annu Rev Biochem. 2017 Jun 20;86:27-68. doi: 10.1146/annurev-biochem-061516-045115. Epub 2017 May 12. PMID: 28498720.
2. Protein complex prediction with AlphaFold-Multimer. Richard Evans, Michael O’Neill, Alexander Pritzel, Natasha Antropova, Andrew Senior, Tim Green, Augustin Žídek, Russ Bates, Sam Blackwell, Jason Yim, Olaf Ronneberger, Sebastian Bodenstein, Michal Zielinski, Alex Bridgland, Anna Potapenko, Andrew Cowie, Kathryn Tunyasuvunakool, Rishub Jain, Ellen Clancy, Pushmeet Kohli, John Jumper, Demis Hassabis. bioRxiv 2021.10.04.463034; doi: https://doi.org/10.1101/2021.10.04.463034.
3. Independent SE(3)-Equivariant Models for End-to-End Rigid Protein Docking. Octavian-Eugen Ganea, Xinyuan Huang, Charlotte Bunne, Yatao Bian, Regina Barzilay, Tommi Jaakkola, Andreas Krause. arXiv:2111.07786v1 [cs.AI] 15 Nov 2021.