Anna Zheltova
A-, B-, Z- form DNA (A-, B-, Z-формы ДНК)
Complexes of DNA-protein (Комплексы ДНК-белок)
Reading Sanger sequencing (Прочтение последовательностей по Сэнгеру)
Nucleotide databanks (Нуклеотидные банки данных)
Aligning genomes (Выравнивание геномов)
The genes of prokaryotes (Гены прокариот)
The genes of eukaryotes (Гены эукариот)
Search for snp (Поиск полиморфизмов)
Задание 1. Построить карту сходства хромосом двух родственных бактерий.
Был использован алгоритм blastn для выравнивания 2 последовательностей (blast2seq) с сайта NCBI.
Было проведено выравнивание каждой хромосомы одной бактерии с каждой хромосомой другой бактерии (алгоритм работал с параметрами по умолчанию).
Для выполнения данного задания я взяла два разных вида рода Brucella, у каждого из которых имеются 2 хромосомы:
o Brucella ovis ATCC 25840 ( хромосома 1 ,хромосома 2 ) и:
• Brucella suis bv. 2 strain Bs143CITA ( хромосома 1 ,хромосома 2 )
1) Brucella_ovis_ATCC_25840_chromosome1 и Brucella suis bv. 2 strain Bs143CITA chromosome I
По горизонтали Brucella_ovis_ATCC_25840, по вертикали Brucella suis bv. 2 strain Bs143CITA

Это выравнивание прямых последовательностей, красной рамкой выделена инверсия протяженного участка, синей рамкой выделено место, в котором произошла либо делеция, либо вставка.
2) Brucella ovis ATCC 25840 chromosome II и Brucella suis bv. 2 strain Bs143CITA chromosome II
По горизонтали Brucella_ovis_ATCC_25840, по вертикали Brucella suis bv. 2 strain Bs143CITA
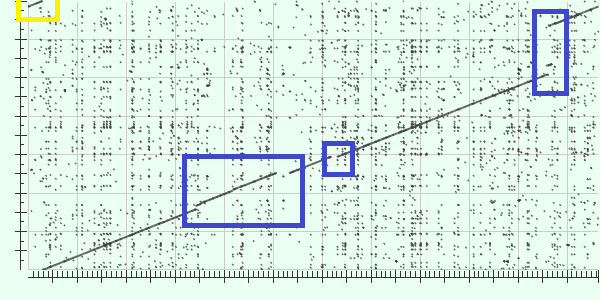
Это выравнивание прямых последовательностей, синими рамками выделены места, в которых произошли либо делеции, либо вставки, желтой рамкой выделен участок, в котором произошла транслокация начального участка у одного из видов в начало или в конец.
3) Brucella ovis ATCC 25840 chromosome I и Brucella suis bv. 2 strain Bs143CITA chromosome II
По горизонтали Brucella_ovis_ATCC_25840, по вертикали Brucella suis bv. 2 strain Bs143CITA
Query cover=14%
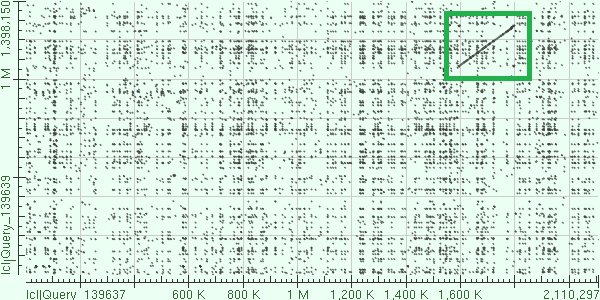
Как и ожидалось, в выравнивании наблюдается один крупный участок, соответствующий крупному эволюционному событию (в зеленой рамке)
4) Brucella ovis ATCC 25840 chromosome II и Brucella suis bv. 2 strain Bs143CITA chromosome I
по горизонтали Brucella_ovis_ATCC_25840, по вертикали Brucella suis bv. 2 strain Bs143CITA
Query cover=6%

Как и ожидалось, не было обнаружено никаких крупных участков.
• Brucella ceti TE28753-12 ( хромосома 1 ,хромосома 2 )
1) Brucella ovis ATCC 25840 chromosome I и Brucella ceti TE28753-12 chromosome 1
по горизонтали Brucella ovis ATCC 25840, по вертикали Brucella ceti TE28753-12
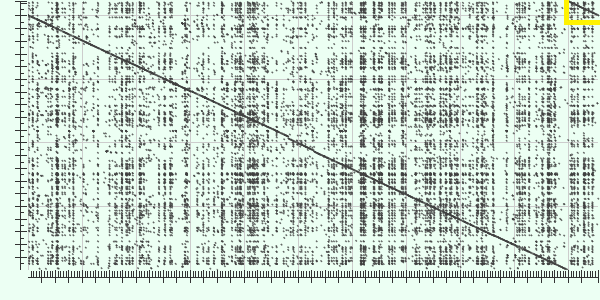
Наблюдается как будто инвертированная линия, вероятно, из-за того, что в геноме штамма Brucella ceti TE28753-12 была секвенирована комплементарная цепь. Желтой рамкой выделен участок, в котором произошла транслокация участка у одного из видов в начало или в конец.
2) Brucella ovis ATCC 25840 chromosome II и Brucella ceti TE28753-12 chromosome 2
по горизонтали Brucella ovis ATCC 25840, по вертикали Brucella ceti TE28753-12
Наблюдается как будто инвертированная линия, вероятно, из-за того, что в геноме штамма Brucella ceti TE28753-12 была секвенирована комплементарная цепь. Желтой рамкой выделен участок, в котором произошла транслокация участка у одного из видов в начало или в конец. Синими рамками выделены места, в которых произошли либо делеции, либо вставки.
3) Brucella ovis ATCC 25840 chromosome I и Brucella ceti TE28753-12 chromosome 2
Query cover=5%
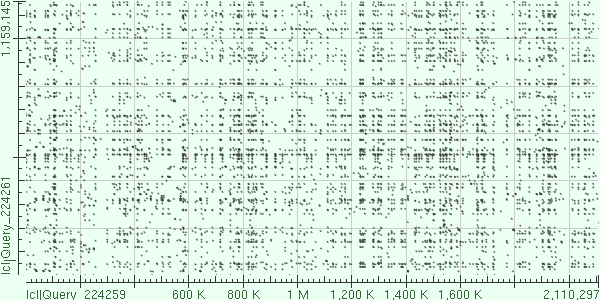
Как и ожидалось, не было обнаружено никаких крупных участков.
4) Brucella ovis ATCC 25840 chromosome II и Brucella ceti TE28753-12 chromosome 1
Query cover=6%

Как и ожидалось, не было обнаружено никаких крупных участков.
• Brucella microti CCM 4915 ( хромосома 1 ,хромосома 2 )
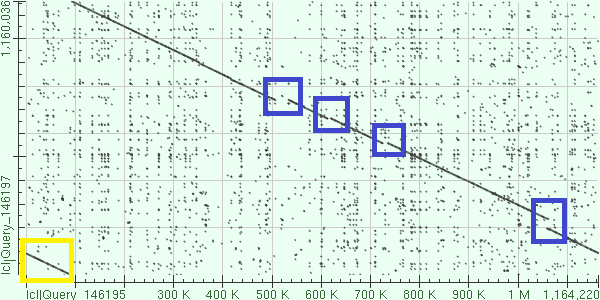
1) Brucella ovis ATCC 25840 chromosome I и Brucella microti CCM 4915 chromosome 1
По горизонтали Brucella ovis ATCC 25840, по вертикали Brucella microti CCM 4915
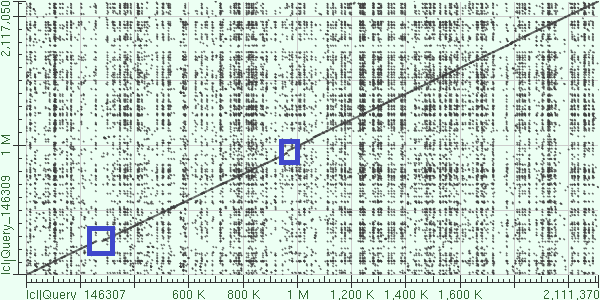
Это выравнивание прямых последовательностей, синими рамками выделены места, в которых произошли либо делеции, либо вставки.
3) Brucella ovis ATCC 25840 chromosome II и Brucella microti CCM 4915 chromosome 2
По горизонтали Brucella ovis ATCC 25840, по вертикали Brucella microti CCM 4915
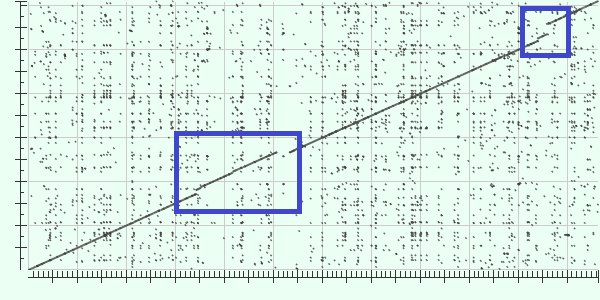
Это выравнивание прямых последовательностей, синими рамками выделены места, в которых произошли либо делеции, либо вставки.
3) Brucella ovis ATCC 25840 chromosome I и Brucella microti CCM 4915 chromosome 2
Query cover=5%

Как и ожидалось, не было обнаружено никаких крупных участков.
4) Brucella ovis ATCC 25840 chromosome II и Brucella microti CCM 4915 chromosome 1
Query cover=6%
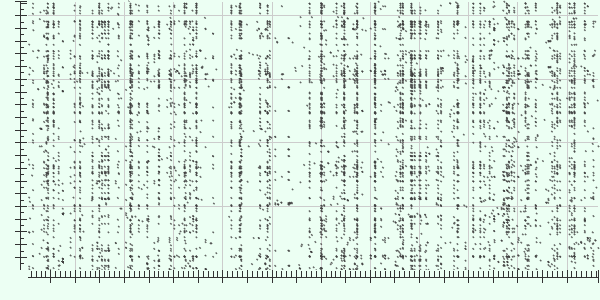
Как и ожидалось, не было обнаружено никаких крупных участков.
Задание 2. Опишите сходство и различие геномов близкородственных бактерий
Были выбраны штаммы, геном каждого представлен с двумя хромосомами:
• Brucella canis HSK A52141
• Brucella canis strain SVA13
• Brucella canis ATCC 23365
Для выполнения данного задания использовался метод построения нуклеотидного пангенома (NPG) с помощью пакета NPG-explorer.
Нуклеотидный пангеном - специальный формат для множественного выравнивания геномов, ориентированный на геномы близкородственных бактерий или архей.
Был создан файл genomes.tsv , в котором содержится информация о том, откуда брать последовательности геномных ДНК и аннотации генов.
Затем я с помощью команд создала файл npge.conf, в котором содержатся параметры программы. Я изменила параметр MIN_IDENTITY на рекомендованное программой для моего случая значение 0.895.
Далее я получила нуклеотидный пангеном и много файлов с аналитической информацией, которую я визуализовала, скачав файл qnpge.exe и запустив на своем компьютере.
Результаты сравнения геномов с помощью пакета NPGE
1. Глобальные блоки (G-блоки) представляют собой синтеничные участки - объединения стабильных блоков.
Число g-блоков: 7 (по данным из файла blocks.gbi ).
• g6x152
• g6x144
• g6x124
• g6x109
• g6x103
• g6x102
• g6x100
В каждом блоке содержится по 6 фрагментов.
Из файла blocks.blocks была получена информация о порядке расположения блоков в хромосоме для каждого генома.
файл в Excel с получившимися данными
Для 1 хромосомы:
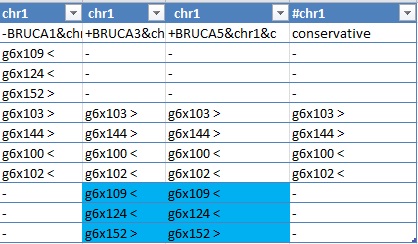
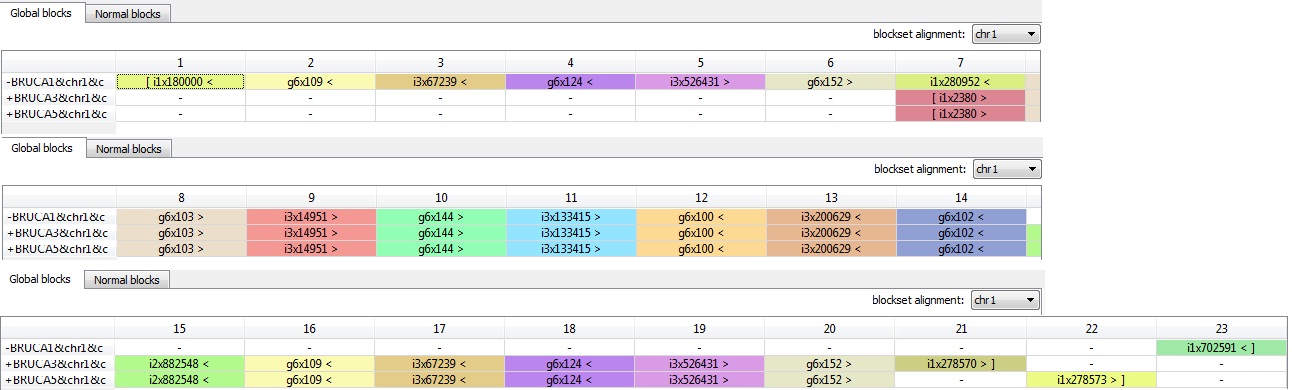
• Только три блока g6x103, g6x152, g6x144,были расположены на прямой цепи (символ ">"), оставшиеся - на обратной (символ "<")
• Если принять за эталон расположение блоков в штамме Brucella canis HSK A52141 (1 столбец BRUCA1), то можно сделать следующие выводы:
o У штаммов Brucella canis strain SVA13 (в таблице – BRUCA3) и Brucella canis ATCC 23365 (в таблице – BRUCA5) наблюдается транслокация трех первых блоков (g6x109, g6x124, g6x152). Данное эволюционное изменение выделено в таблице синим цветом.
Участок выравнивания i- и g-блоков хромосомы 1, показывающий наличие крупных эволюционных изменений в геномах бактерий всех рассматриваемых штаммов.
Для хромосомы 2:
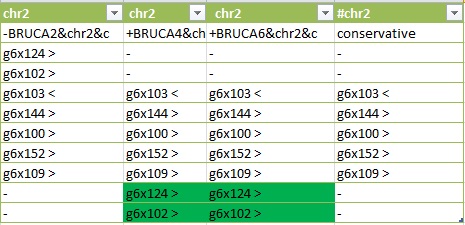
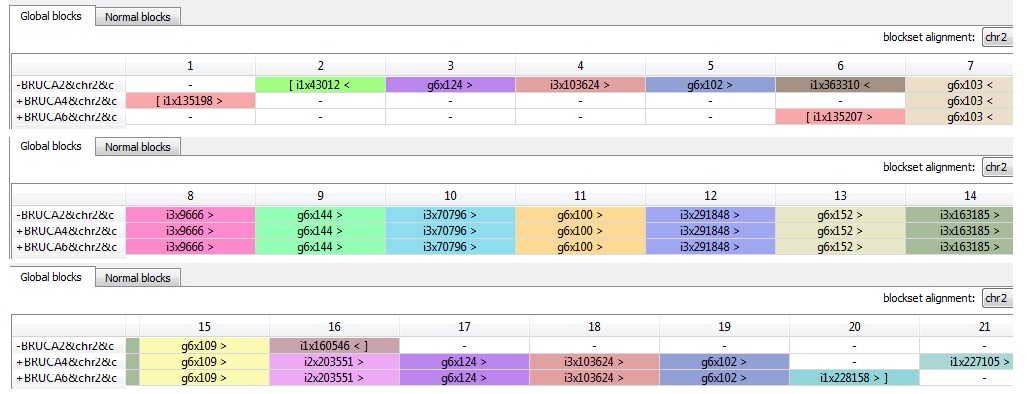
• Все блоки были расположены на прямой цепи (символ ">"), кроме блока g6x103 (на обратной (символ "<"))
• Если принять за эталон расположение блоков в штамме Brucella canis HSK A52141 (1 столбец BRUCA2), то можно сделать следующие выводы:
o У штаммов Brucella canis strain SVA13 (в таблице – BRUCA4) и Brucella canis ATCC 23365 (в таблице – BRUCA6) наблюдается транслокация двух первых блоков (g6x124, g6x102). Данное эволюционное изменение выделено в таблице зеленым цветом.
2. S-блоки
Стабильные блоки (S-блоки) - ядро генома
• Число S-блоков: в 6 исследуемых геномах по 7 s-блоков в каждом
• Cуммарная длина (процент от длины генома в среднем): 4998 (0.05%)
• Процент консервативных позиций в объединенном выравнивании s-блоков (т. е. сходство геномов): 0.918432
3. Описание повторов на примерах r-блоков:
Наиболее часто встречающийся блок r21x104.
Он содержит 21 фрагмент, имеет длину 104 позиции. Всего 21 повтор во всех геномах (от 3 до 4 раз во всех геномах в каждой хромосоме).
Данные файла pangenome.bi :

4. Описание крупных делеций на примерах h-блоков:
h-блоки - "полустабильные" блоки.
Для рассмотрения были взяты:
• h3x169552 (содержит 3 фрагмента, длина 169552 позиции). Данный блок присутствует только во 2-ых хромосомах всех штаммов.
Похожая ситуация наблюдалась для всех h3-блоков (либо присутствовали только в первой хромосоме, либо только во второй у всех штаммов)
Поэтому были рассмотрены все h2-блоки:
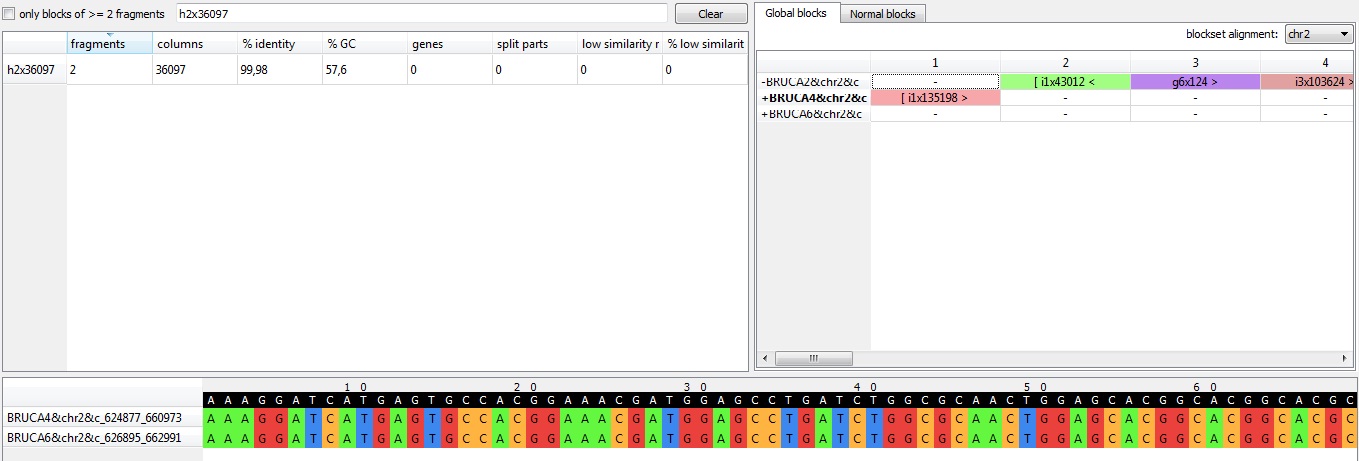
• h2x36097 (содержит 2 фрагмента, длина 36097 позиций). Данный блок присутствует в 2 хромосоме штаммов Brucella canis strain SVA13 (в файлее – BRUCA4) и Brucella canis ATCC 23365 (в файле - BRUCA6), но отсутствует в 1 хромосоме всех штаммов и в 2 хромосоме штамма Brucella canis HSK A52141 (в файле – BRUCA2).Отсутствие данного блока в штамме Brucella canis HSK A52141 может быть объяснено делецией этого участка в данном штамме (или наоборот вставкой данного участка в двух других штаммах).
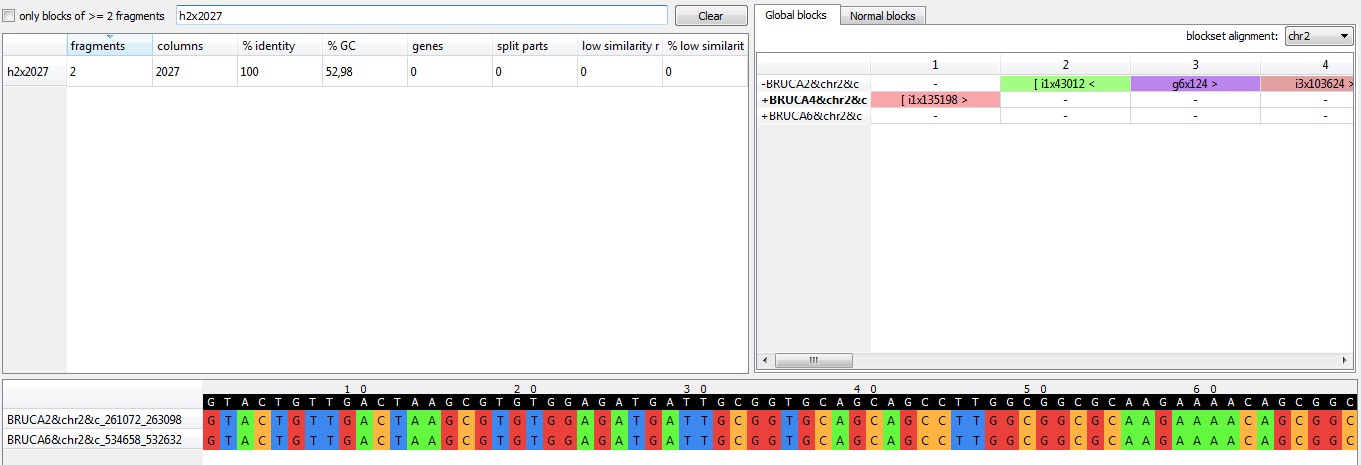
• h2x2027 (содержит 2 фрагмента, длина 2027 позиций). Данный блок присутствует в 2 хромосоме штаммов Brucella canis HSK A52141 (в файле – BRUCA2) и Brucella canis ATCC 23365 (в файле - BRUCA6), но отсутствует в 1 хромосоме всех штаммов и в 2 хромосоме штамма Brucella canis strain SVA13 (в файлее – BRUCA4).Отсутствие данного блока в штамме Brucella canis strain SVA13 может быть объяснено делецией этого участка в данном штамме (или наоборот вставкой данного участка в двух других штаммах).
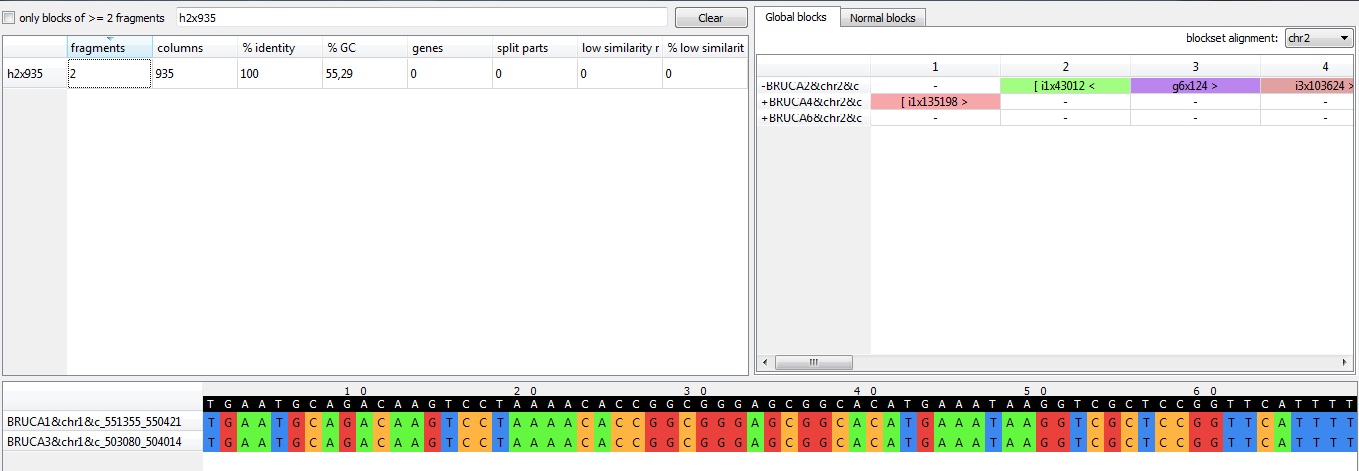
• h2x935 (содержит 2 фрагмента, длина 935 позиций). Данный блок присутствует в 1 хромосоме штаммов Brucella canis HSK A52141 (в файле – BRUCA1) и Brucella canis strain SVA13 (в файлее – BRUCA3), но отсутствует в 2 хромосоме всех штаммов и в 1 хромосоме штамма Brucella canis ATCC 23365 (в файле – BRUCA5). Отсутствие данного блока в штамме Brucella canis ATCC 23365 может быть объяснено делецией этого участка в данном штамме (или наоборот вставкой данного участка в двух других штаммах).
5. U-блоки - уникальные последовательности из одного генома.
К сожалению, не было найдено ни одного U-блока. Возможно, данные геномы имеют очень значительное сходство, а следовательно не имеют u-блоков.
6. Примеры расхождений между аннотациями генов из одного блока.
• В блоке r6x107 qnpge выделяет только 2 гена, один из которых принадлежит штамму Brucella canis HSK A52141 (в файле – BRUCA1), а другой Brucella canis ATCC 23365 (в файле – BRUCA5). Первый кодирует транспортер (cation diffusion facilitator family transporter).
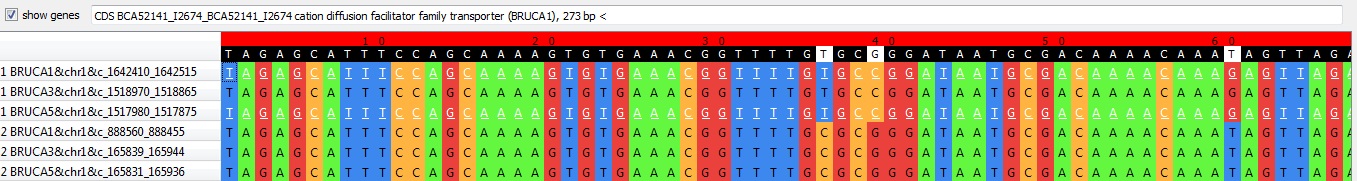
Второй - антранилат-синтазу.
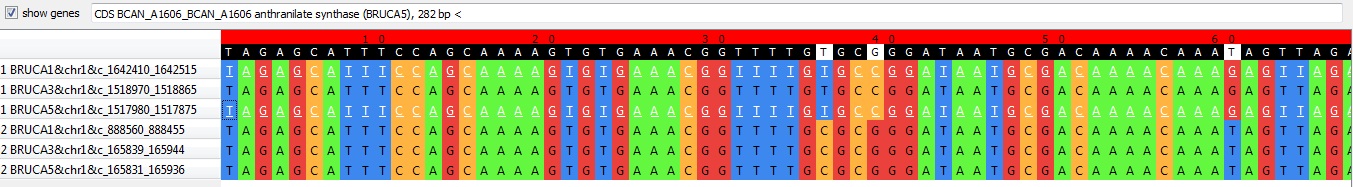
• В блоке m3x18 qnpge выделяет только 2 гена, один из которых принадлежит штамму Brucella canis HSK A52141 (в файле – BRUCA1), а другой Brucella canis ATCC 23365 (в файле – BRUCA5). Первый кодирует peptide chain release factor 2
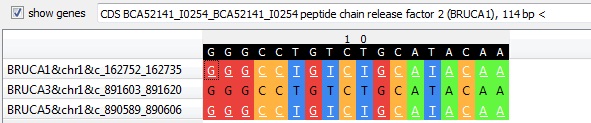
Второй – транспозазу (transposase for insertion sequence element)
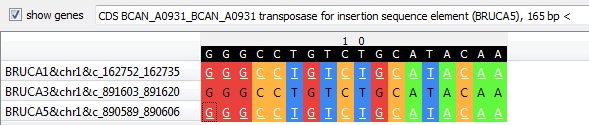
В принципе, NPG-explorer b qnpge помогают «облегчить жизнь» при биоинформатическом анализе.