Anna Zheltova
A-, B-, Z- form DNA (A-, B-, Z-формы ДНК)
Complexes of DNA-protein (Комплексы ДНК-белок)
Reading Sanger sequencing (Прочтение последовательностей по Сэнгеру)
Nucleotide databanks (Нуклеотидные банки данных)
Aligning genomes (Выравнивание геномов)
The genes of prokaryotes (Гены прокариот)
The genes of eukaryotes (Гены эукариот)
Search for snp (Поиск полиморфизмов)
Задание 1. Аннотирование последовательности и сравнение её с аннотацией генов в записи GenBank
Для аннотации была выбрана единственная кольцевая хромосома бактерии Escherichia coli UMN026.
Таксономия организма

GenBank: BA000007.2
Аминокислотная последовательность (GenBank) в fasta-формате.
Для аннотирования последовательности данной хромосомы использовался сервер RAST. С его помощью была получена Excel таблица аннотаций RAST .
Файл с аннотацией генов в формате gbk.
Доли разных функциональных субсистем генов в последовательности хромосомы

RAST аннотировал все гены, среди которых были гены РНК. Таких генов RAST аннотировал 124. Данные гены не учитывались при дальнейшем выполении задания. Рассматривались только гены белков.
Таблица аннотаций генов белков из NCBI, обработанная в Excel.
Таблица аннотаций генов белков из RAST, обработанная в Excel.
Таблица сравнения аннотаций генов.
• RAST аннотировал 5595 генов белков
• GenBank аннотировал 5204.
• Число совпадающих аннотаций: 4133.• Число генов, расположенных на обратной цепи, у которых не совпадают старт-кодоны, а стоп-кодоны совпадают: 402
• Число генов, расположенных на прямой цепи, у которых не совпадают старт-кодоны, а стоп-кодоны совпадают: 426
• Число генов, аннотированных GenBank, но не RAST: 409
• Число гены, аннотированные RAST, но не GenBank: 796
Для 3-х генов с не совпадающими аннотациями была проверена аннотация с помощью blast.
Использовался blastp по банку Swiss_prot для штаммов E.coli
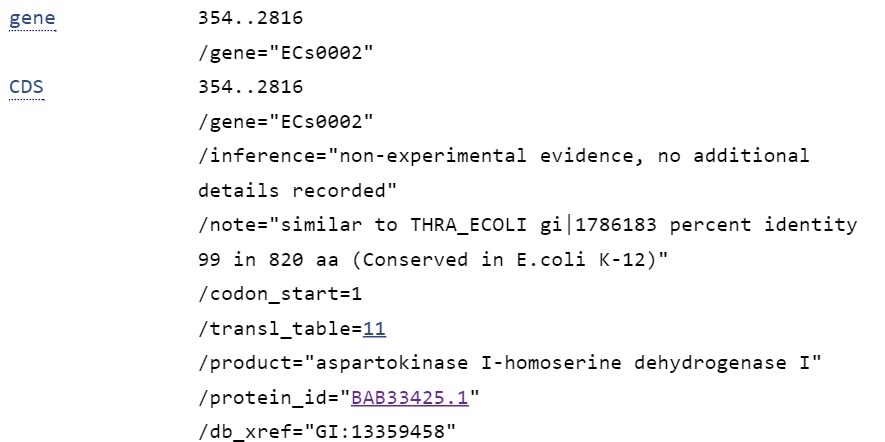
o Белок bifunctional aspartokinase I/homoserine dehydrogenase I, расположенный на прямой цепи, у которого совпадают стоп-, но не совпадают старт-кодоны.

Для GenBank
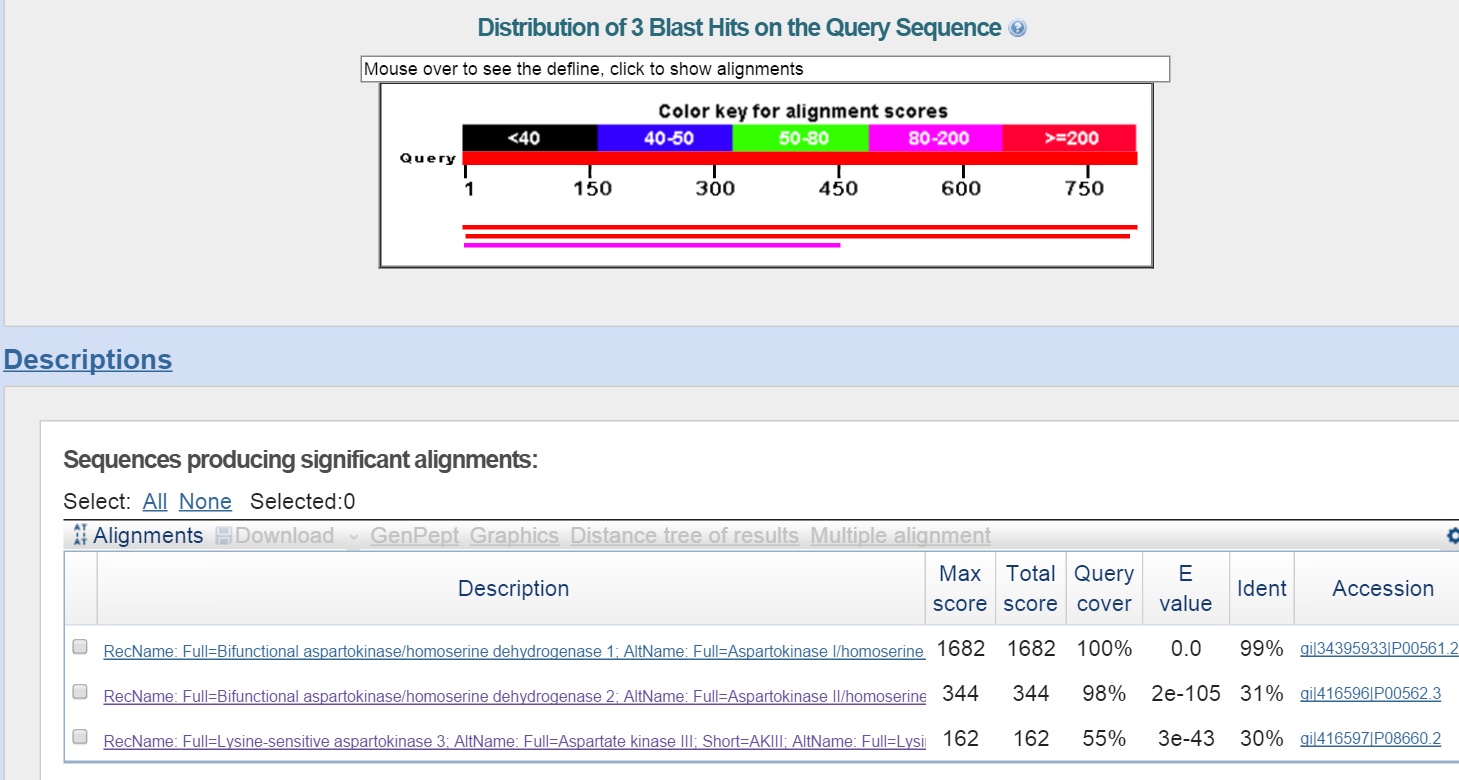
Было найдено три находки. Все они принадлежали штамму Escherichia coli K-12.
Для RAST
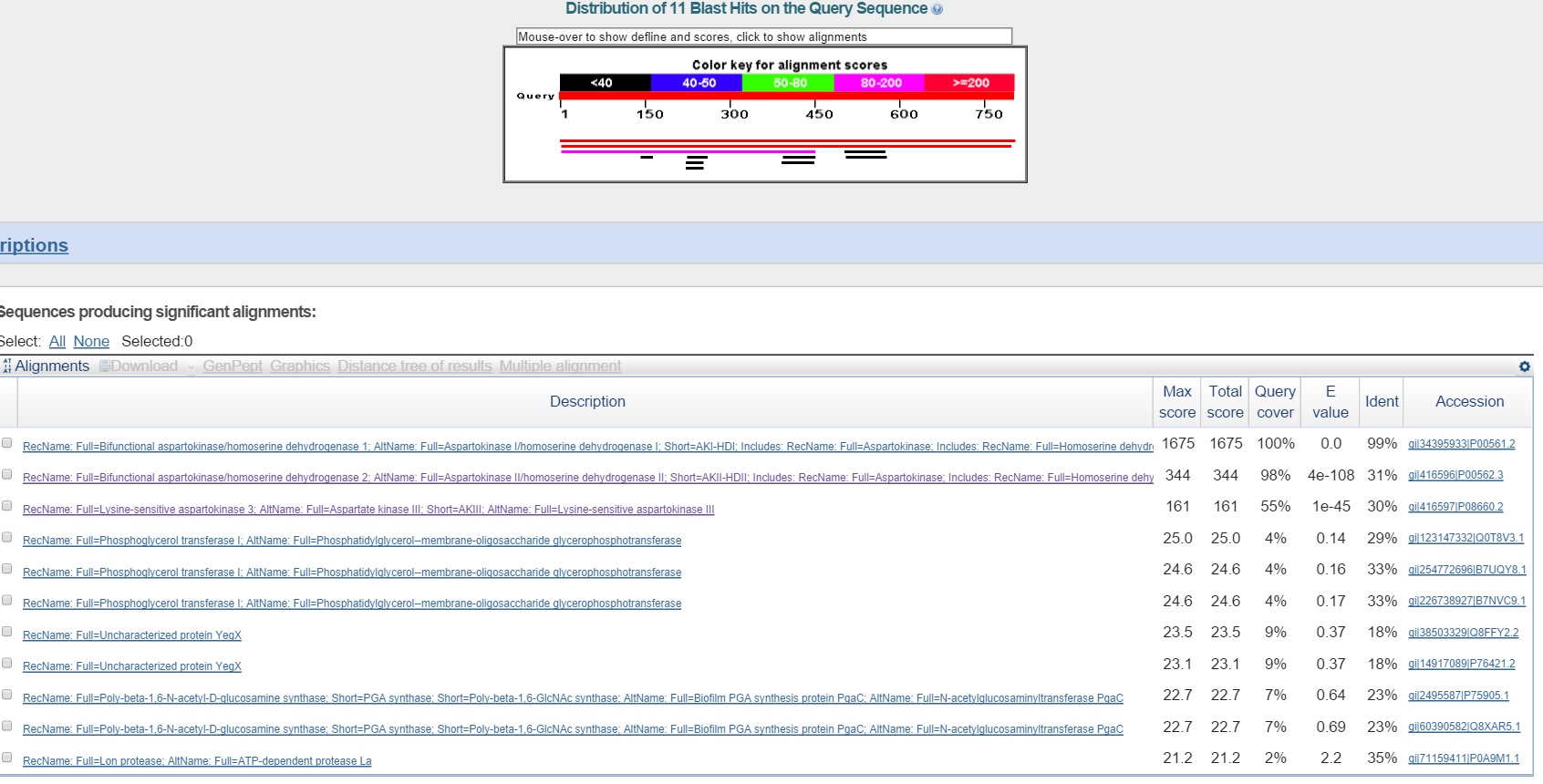
Лучшая находка принадлежала штамму Escherichia coli K-12.
Для лучшей находки аннотации GenBank

Для лучшей находки аннотации RAST

Так как для RAST не совпадают старт- и стоп-кодоны, предпочтительнее выбор в качестве старт-кодона, предложенного GenBank.
o Белок transcriptional regulator, расположенный на обратной цепи, у которого не совпадают старт-кодоны, а стоп-кодоны совпадают.

Для GenBank
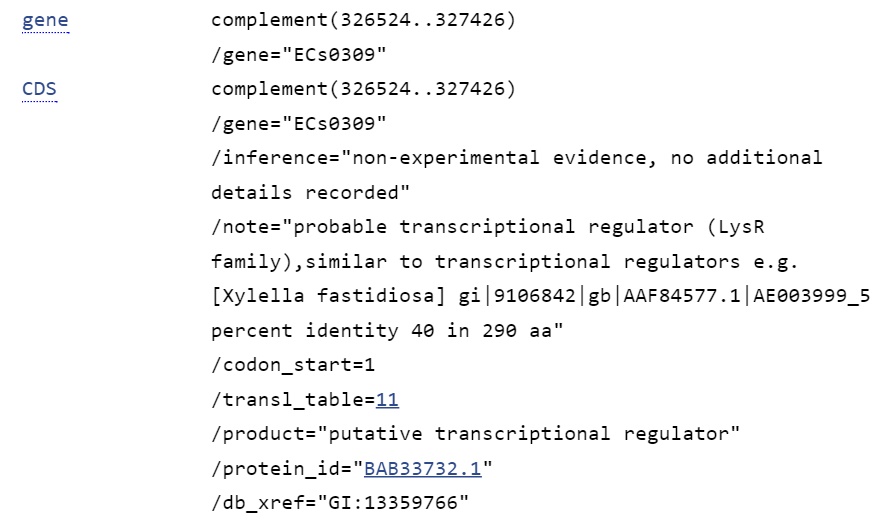
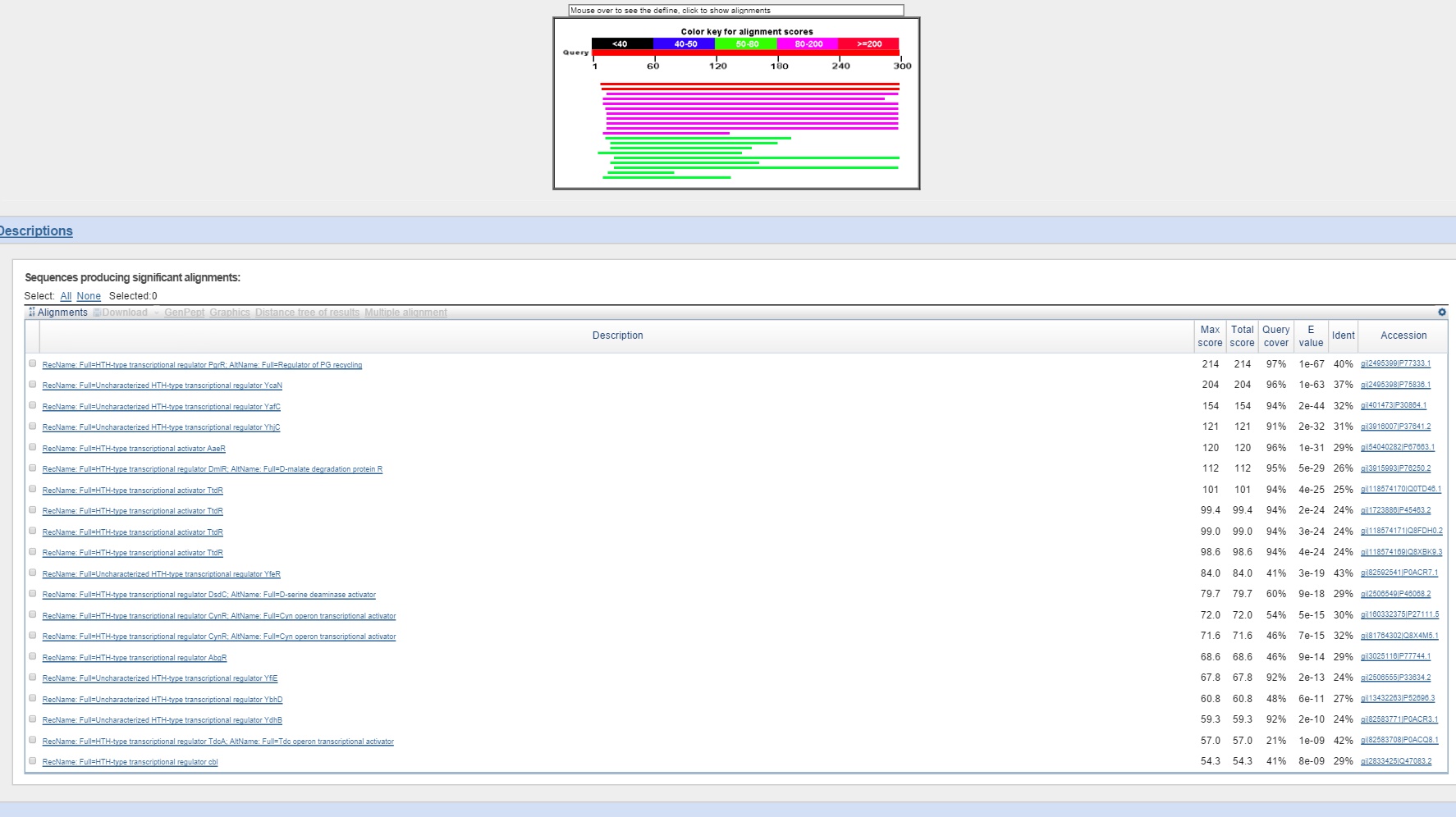
Лучшая находка принадлежала штамму Escherichia coli K-12.
Для RAST
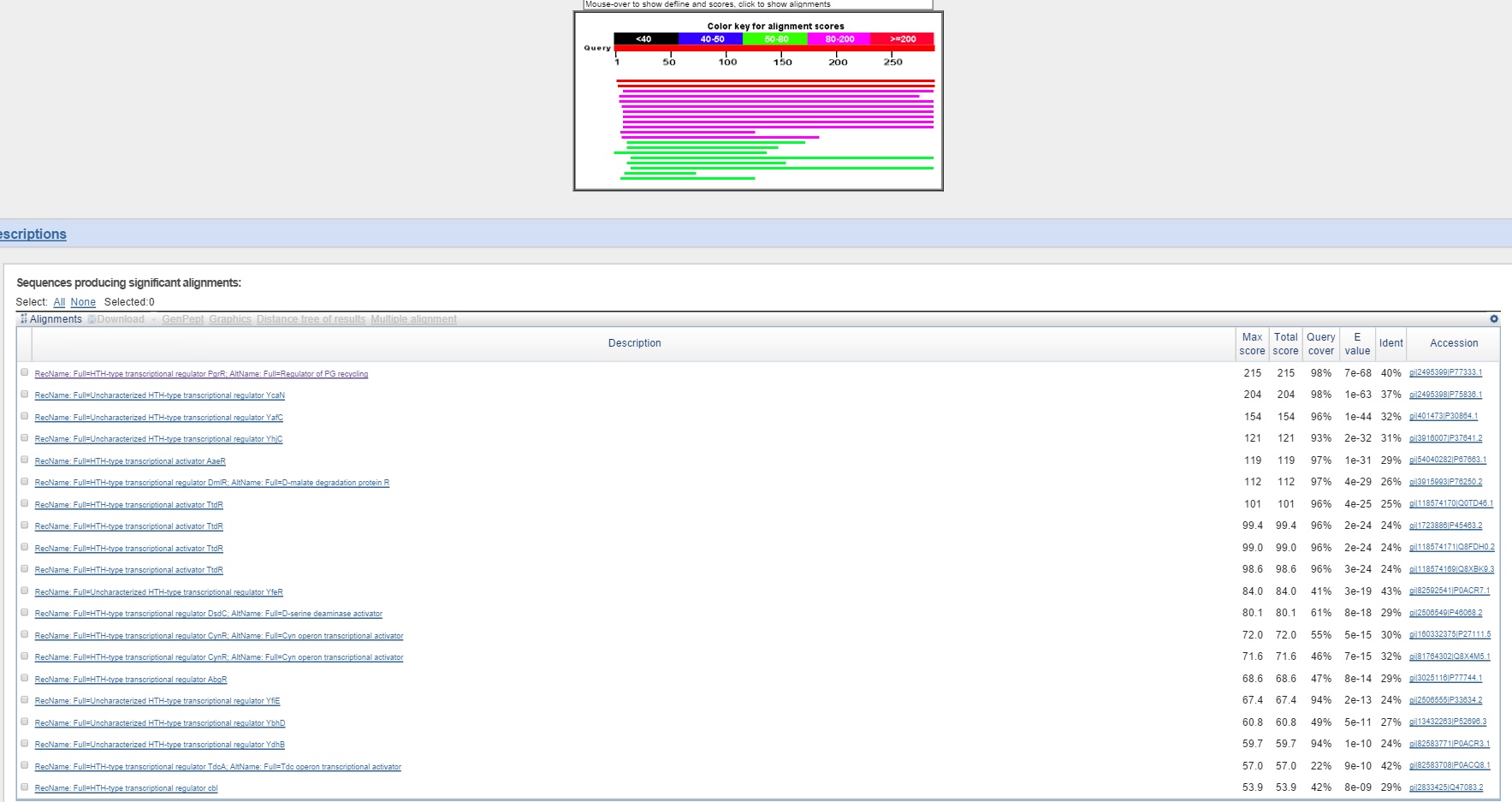
Лучшая находка принадлежала штамму Escherichia coli K-12.
Для лучшей находки аннотации GenBank

Для лучшей находки аннотации RAST
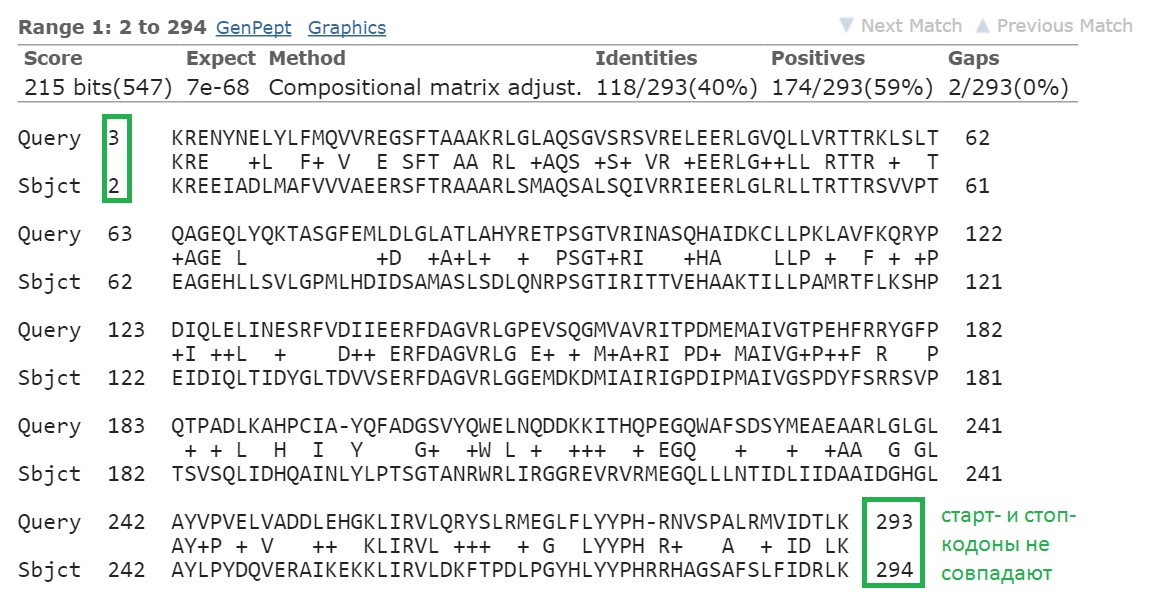
Результаты RAST чуть лучше, но определенности нет: старт-кодоны в обоих случаях не совпадают. Обе находки плохие.
o Белок Inosine/xanthosine triphosphatase (EC 3.6.1.-) (RAST) или NTPase (GenBank), расположенный на обратной цепи, у которого не совпадают старт-кодоны, а стоп-кодоны совпадают. Так же несовпадение названий продуктов.

Для GenBank

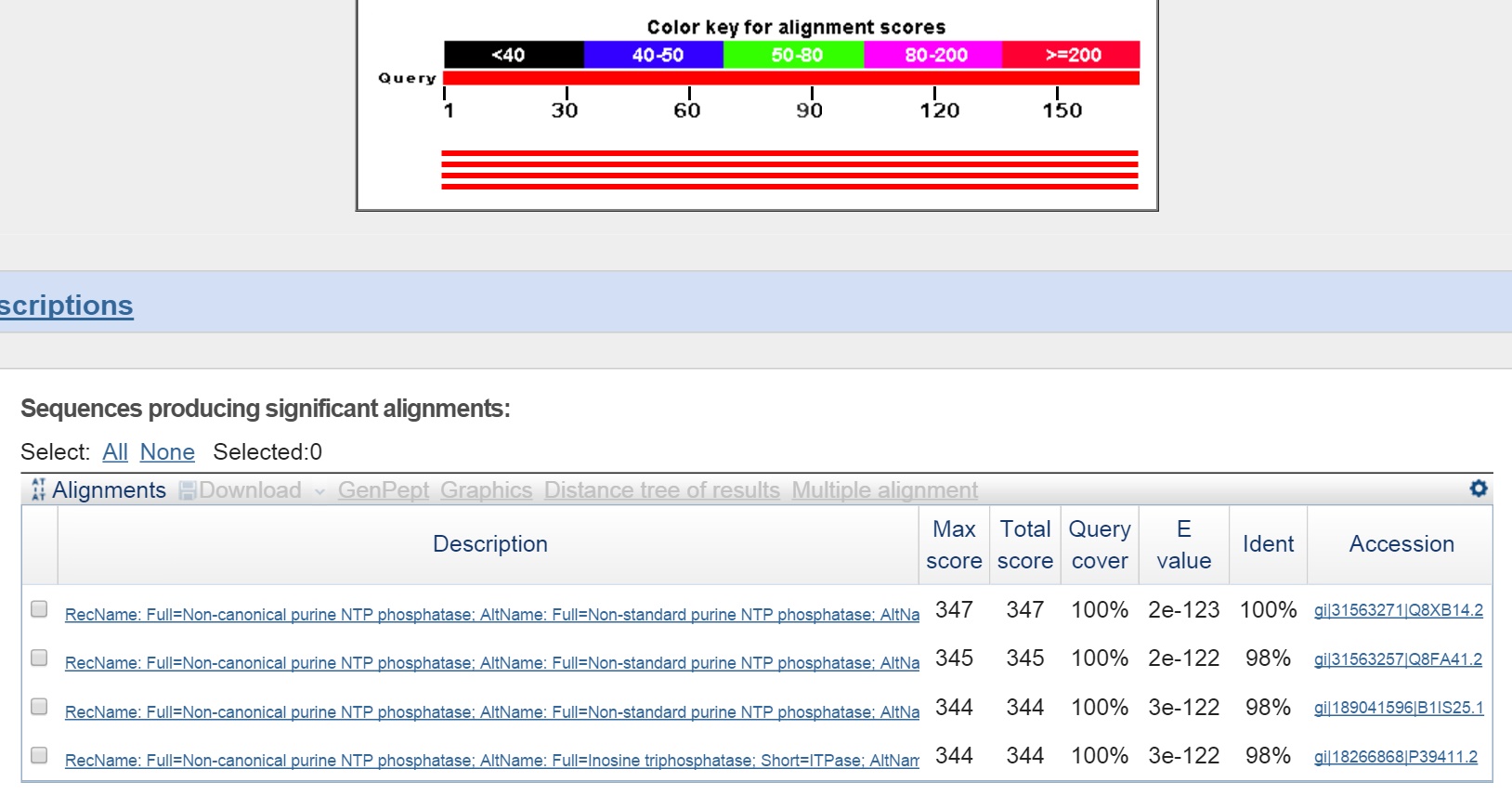
Лучшая находка принадлежала исследуемому штамму Escherichia coli O157:H7
Для RAST
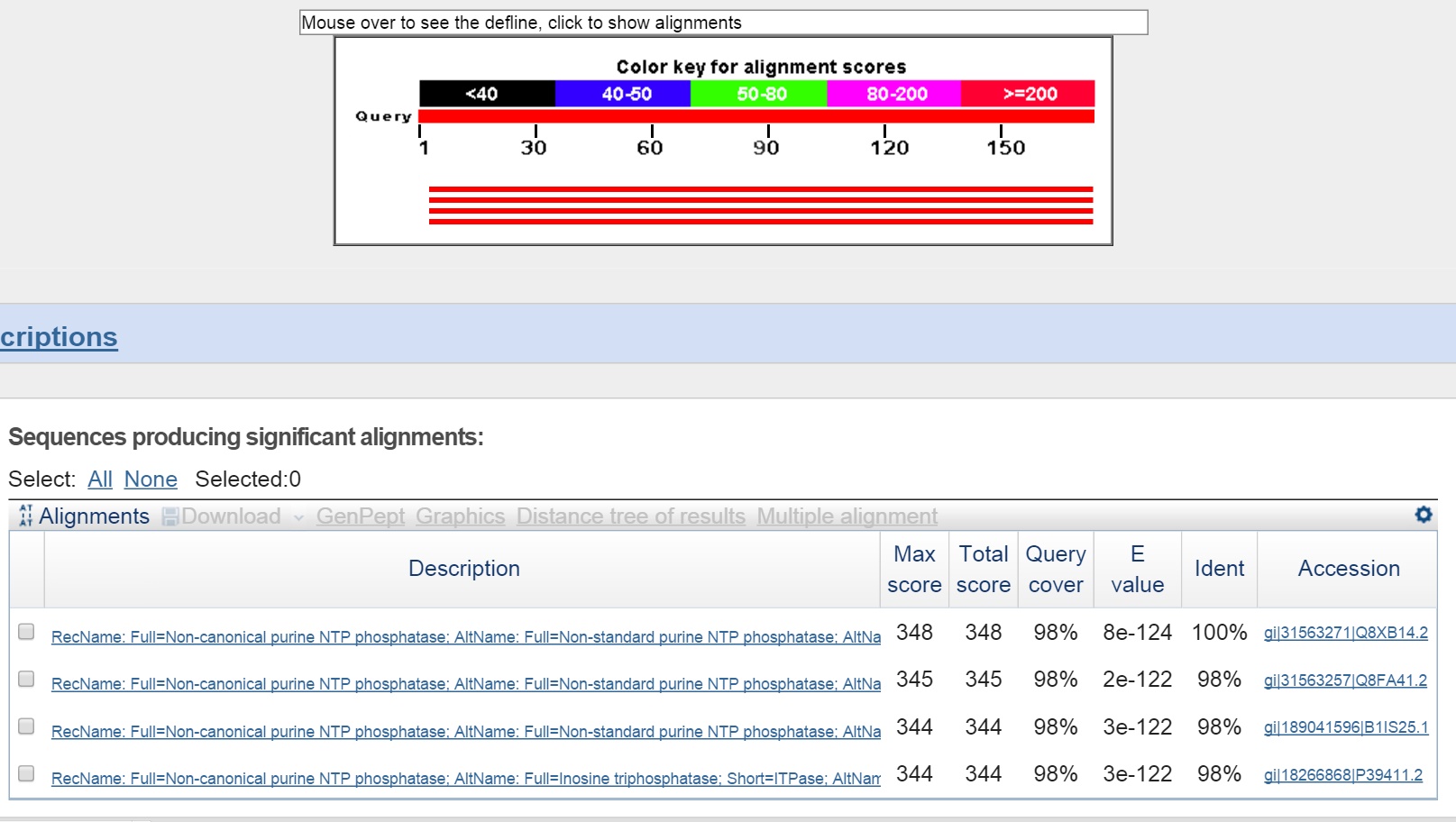
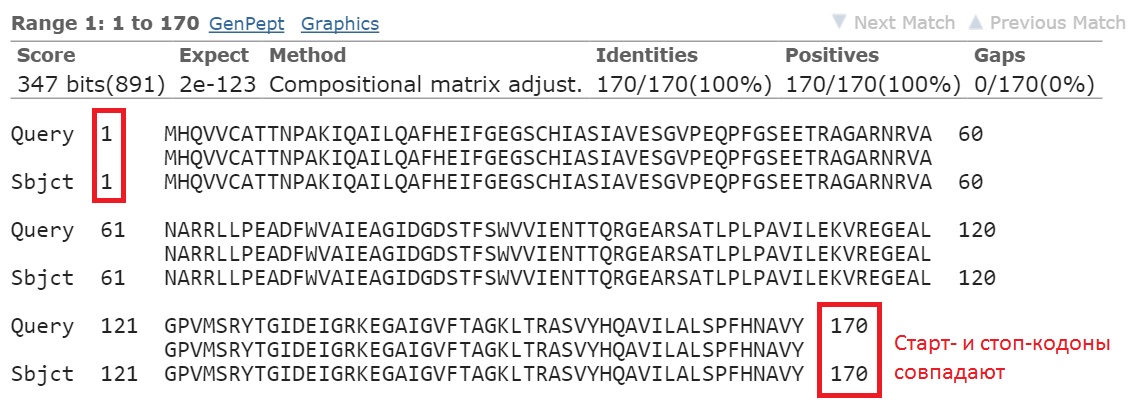
Лучшая находка принадлежала исследуемому штамму Escherichia coli O157:H7Для лучшей находки аннотации GenBank
Для лучшей находки аннотации RAST
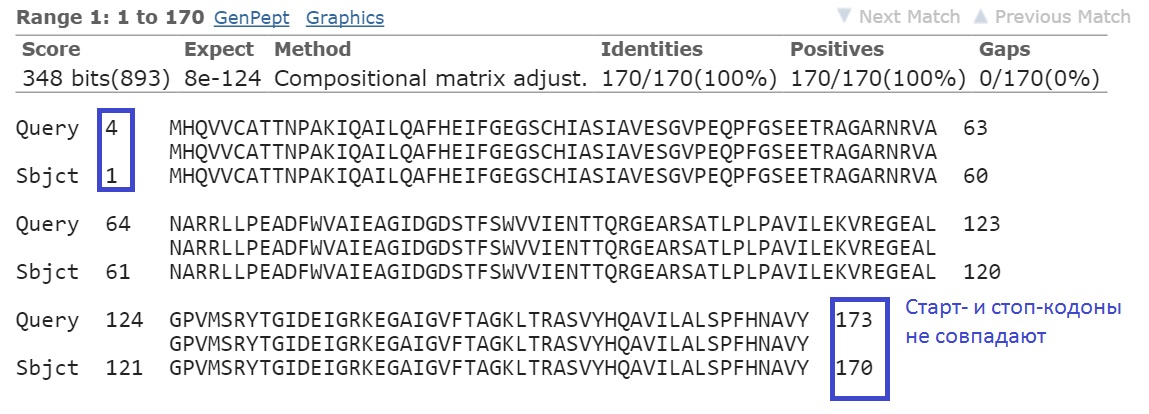
Лучшими в обоих случаях являются результаты для NTPase, следовательно, у Genbank правильное название продукта.
Так как для RAST не совпадают старт- и стоп-кодоны, предпочтительнее выбор в качестве старт-кодона, предложенного GenBank.