Anna Zheltova
A-, B-, Z- form DNA (A-, B-, Z-формы ДНК)
Complexes of DNA-protein (Комплексы ДНК-белок)
Reading Sanger sequencing (Прочтение последовательностей по Сэнгеру)
Nucleotide databanks (Нуклеотидные банки данных)
Aligning genomes (Выравнивание геномов)
The genes of prokaryotes (Гены прокариот)
The genes of eukaryotes (Гены эукариот)
Search for snp (Поиск полиморфизмов)
Задание 1.Предскажите гены X5 с помощью AUGUSTUS
Из сборки генома X5 был выбран один скэффолд (scaffold-698), длина которого составляет 23460 п.о.
infoseq с опциями -only -name -snucleotide1 –length
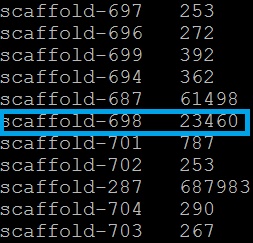
Последовательность этого скэффолда в формате fasta
Этап 1. Предсказание генов с помощью AUGUSTUS
Для начала необходимо выбрать организм, наиболее близкий к какому-либо организму из списка, для которого уже произведено обучение сервиса AUGUSTUS
Для этого был использован алгоритм blastx, транслирующий поданную ему неклеотидную последовательность в 6 рамках и ищет гомологичные белки в банке данных белков. Данный алгоритм ищет именно продукт гена.
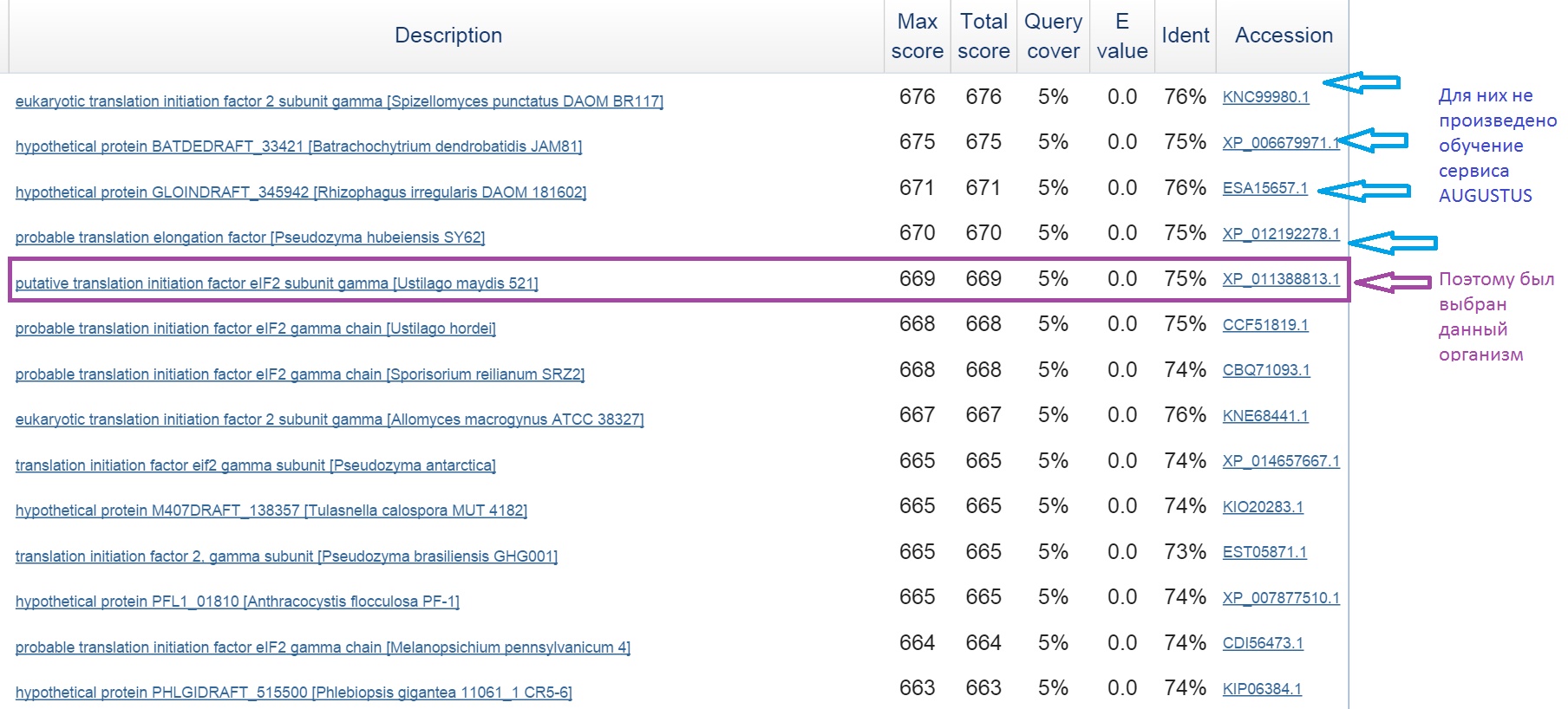
Самым близким организмом, для которого было произведено обучение сервиса AUGUSTUS - Ustilago maydis (fungus)
Страница с описанием статуса задания.
Ссылка от 22 ноября дана так как
Результат работы AUGUSTUS включает 6 файлов:
o augustus.aa содержит аминокислотные последовательности белков, транслированные с найденных генов (формат fasta).
o augustus.cdsexons содержит нуклеотидные последовательности экзонов найденных в предсказанных генах (формат fasta).
o augustus.codingseq содержит кодирующие нуклеотидные последовательности генов.
o augustus.gbrowse представляет информацию, обрабатываемую геномным браузером GBrowse;
o augustus.gff содержит подробную информацию о предсказанных генах. Указаны координаты: интронов, CDS, гена, его транскрипта, старт- и стоп-кодонов.
o augustus.gtf содержит ту же информацию, что и файл augustus.gff в виде единой таблицы.
Этап 2. Проверка предсказания с помощью BLAST
Было предсказано всего 2 гена, кодирующих белки. Файл augustus.aa, выданный AUGUSTUS, содержит последовательности предсказанных белков в fasta формате.
g1, g2 были выбраны для проверки с помощью BLAST. Предсказанная структура данных генов была взята из файла augustus.gff и обработана в Excel .
Далее для аминокислотных последовательностей предсказанных белков из файла augustus.aa проводился поиск гомологов с помощью blastp по БД Swissprot. Область поиска была ограничена таксоном Fungi.
g1
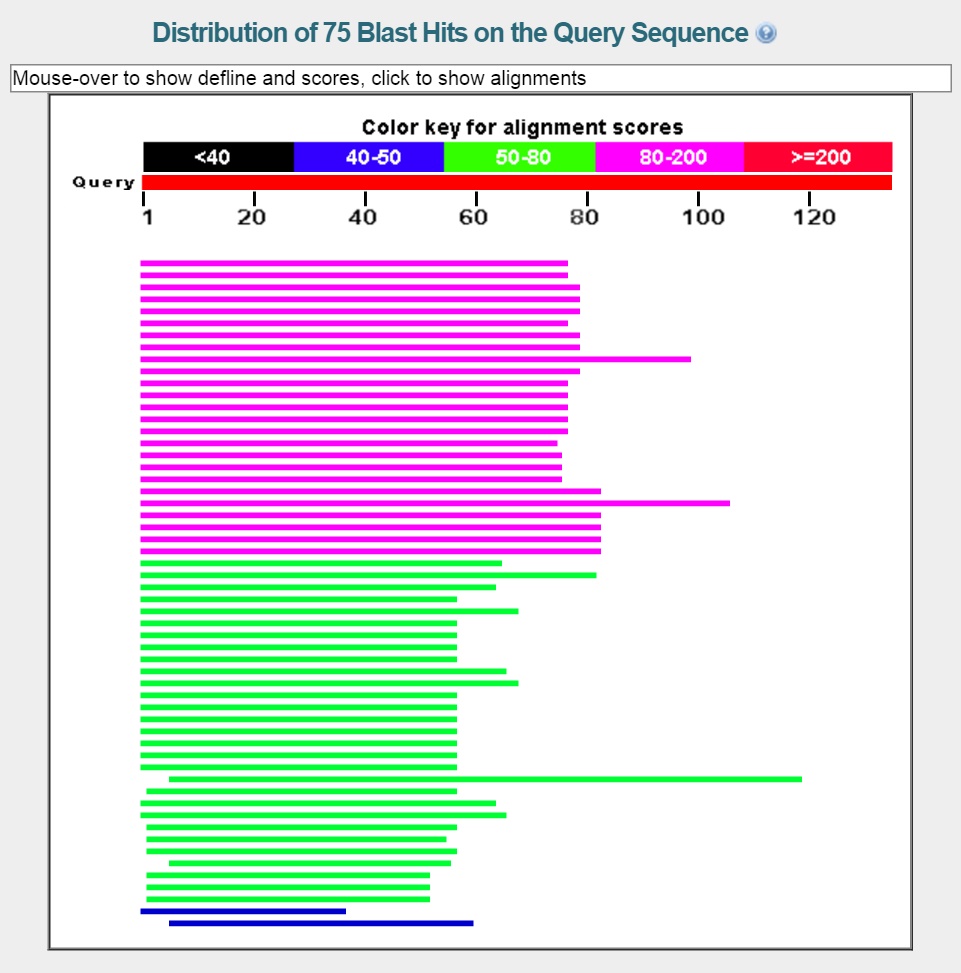
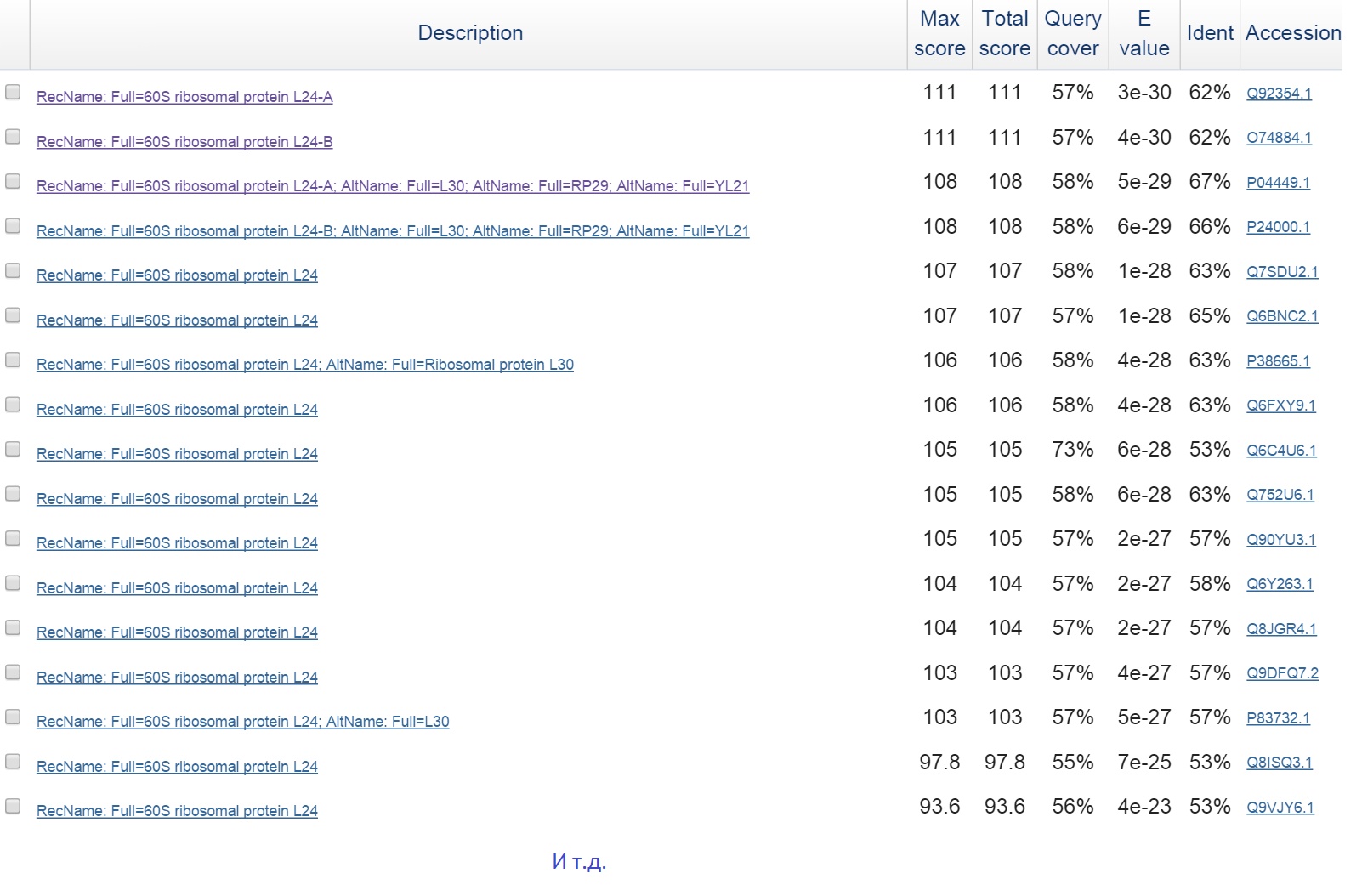
Первая находка имеет подтверждение только на уровне гомологии.
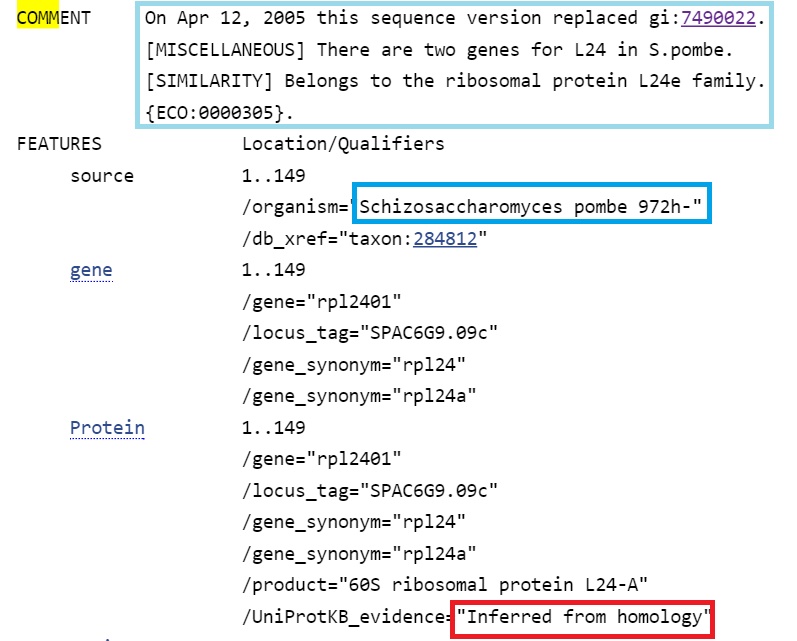
Вторая имеет также хороший score, но зато подтверждена экспериментально.
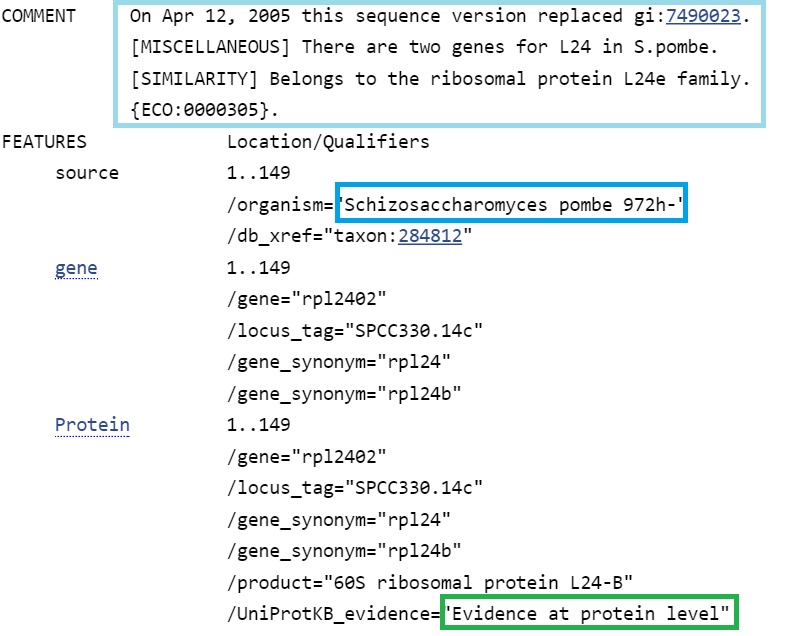
Третья находка имеет также хороший score и подтверждена экспериментально.
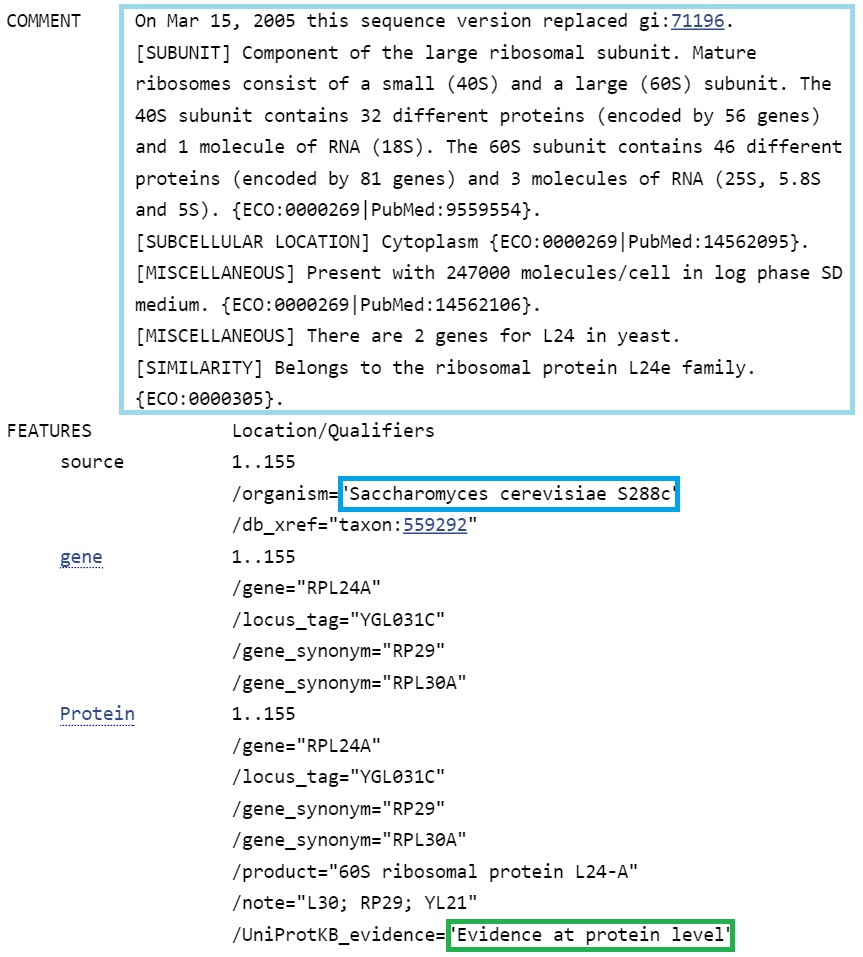
Конкретно функции указано не было. Но, можно предположить, что т.к. лучшие находки принадлежат Schizosaccharomyces pombe 972h-, возможно рассмотреть функции в данном организме:
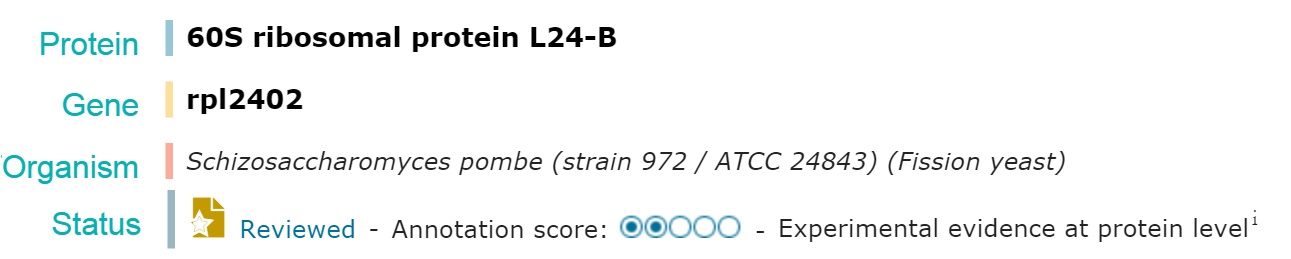

Функции были рассмотрены на примере 2 находки, так как первая подтверждена только на уровне гомологии.
Стоит отметить, что две лучшие находки принадлежат Schizosaccharomyces pombe 972h-. Не было находок с score более 200. Можно сделать вывод, что действительно предсказанный ген - ген, кодирующий рибосомальный белок L24, однако, вероятно, AUGUSTUS ошибся в определении границ гена и экзон-интронной структуры: на самом деле белок короче предсказанного ресурсом. Таким образом, достоверных гомологов нет.
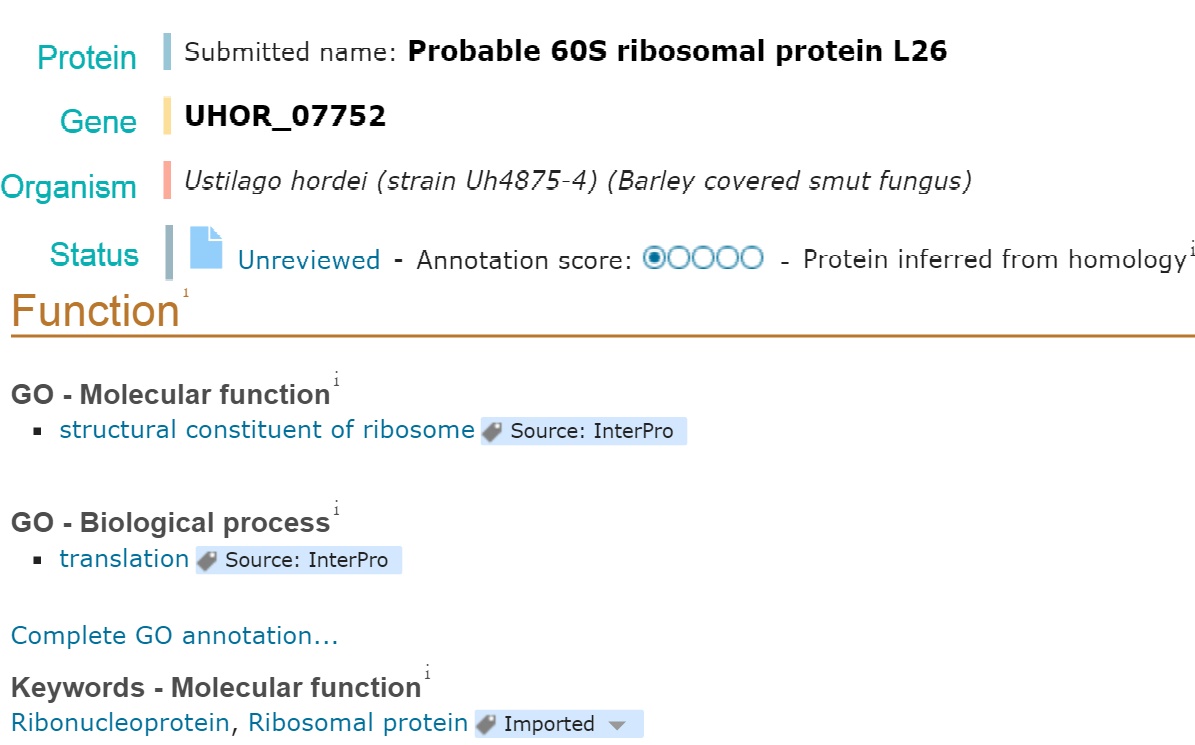
Однако, следует отметить, что существование 60s ribosomal protein l24 для Ustilago не подтверждено. Известно только о возможном существовании 60s ribosomal protein l26.
g2

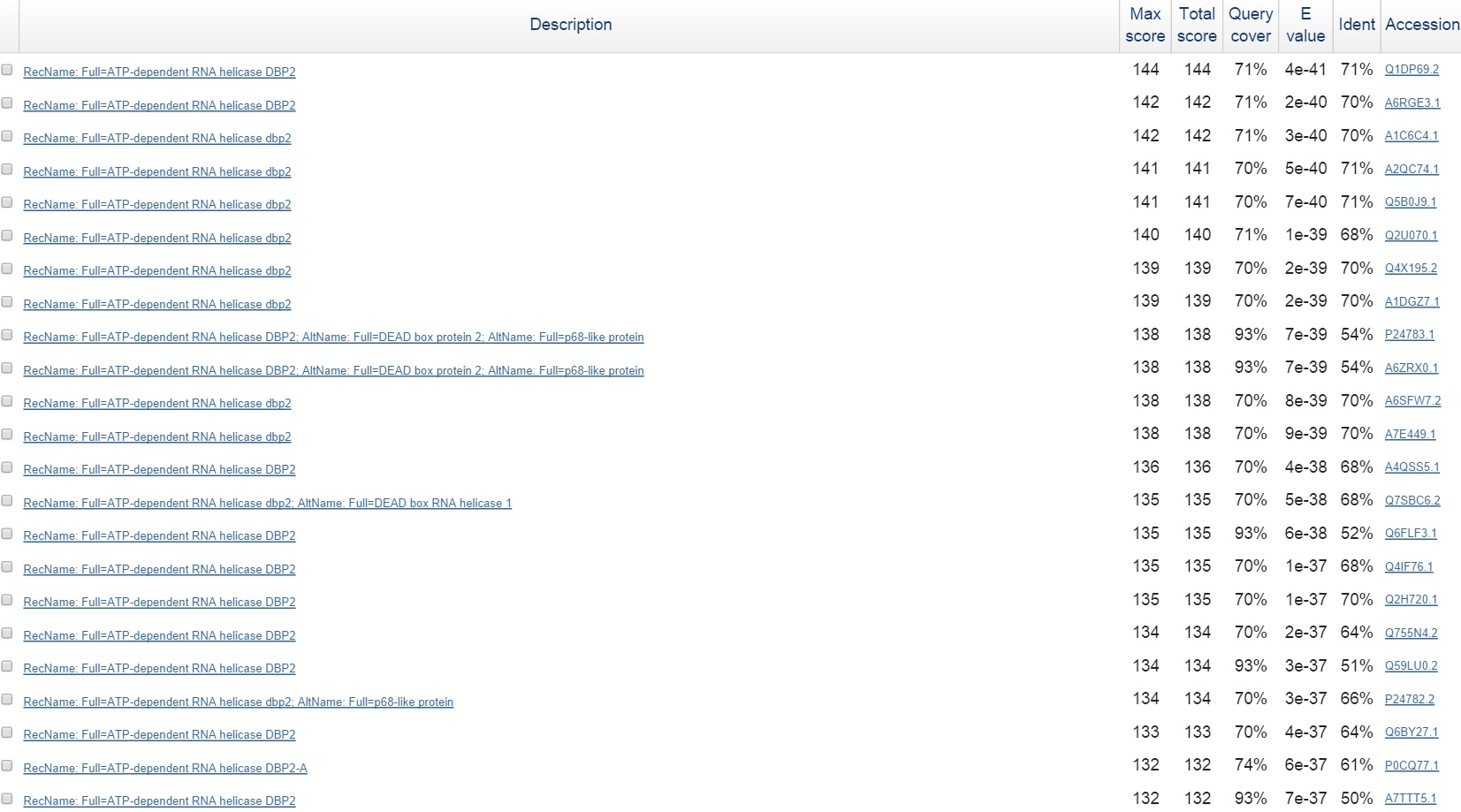
Первая находка имеет подтверждение только на уровне гомологии, как и большинство находок.
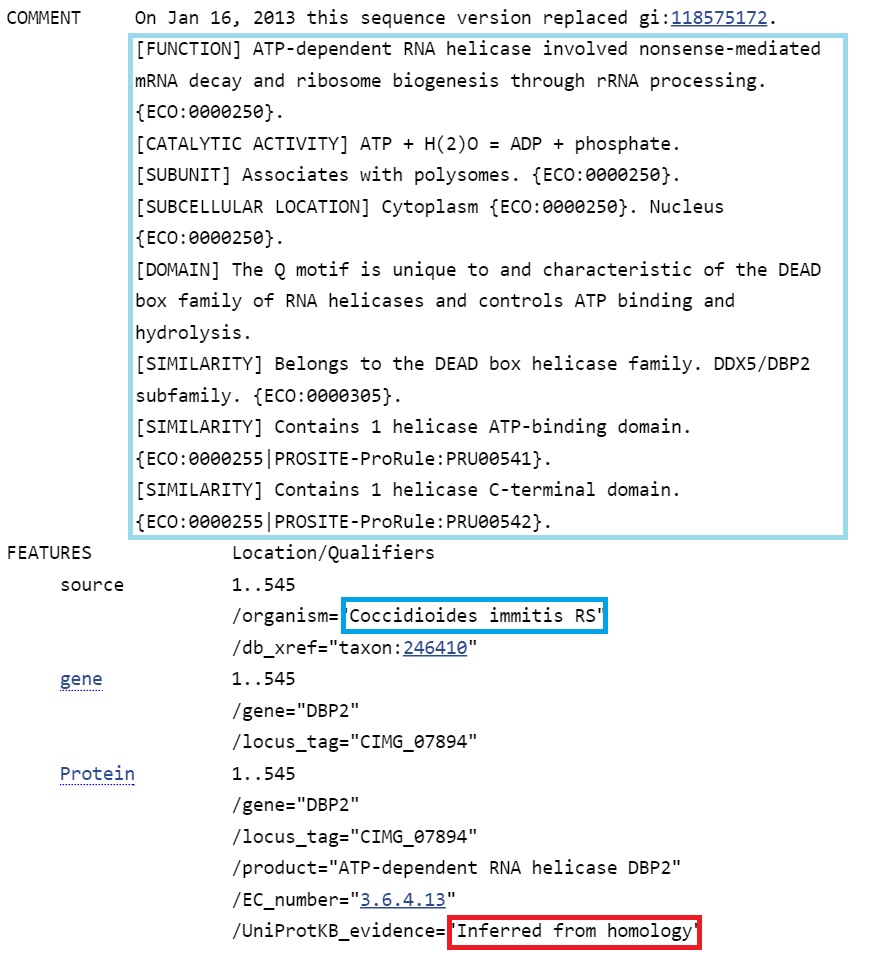
Из 20 первых находок только 20 и 9 подтверждены экспериментально.
9 имеет хороший score и подтверждена экспериментально.
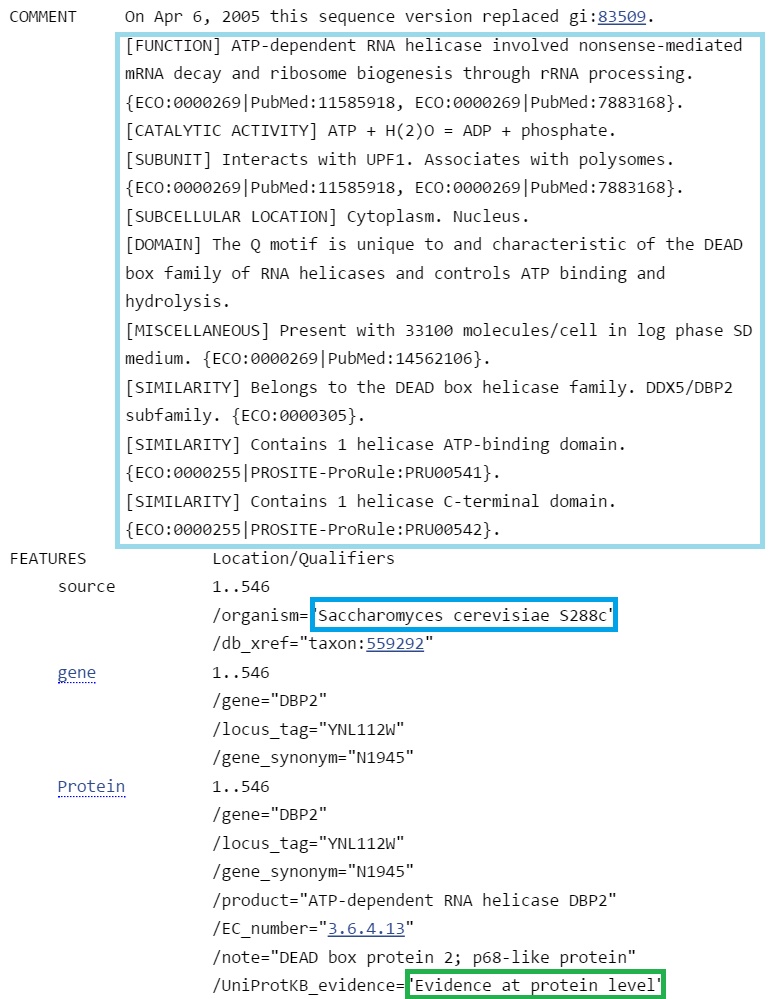
20 находка имеет не такой хороший score, но подтверждена экспериментально.
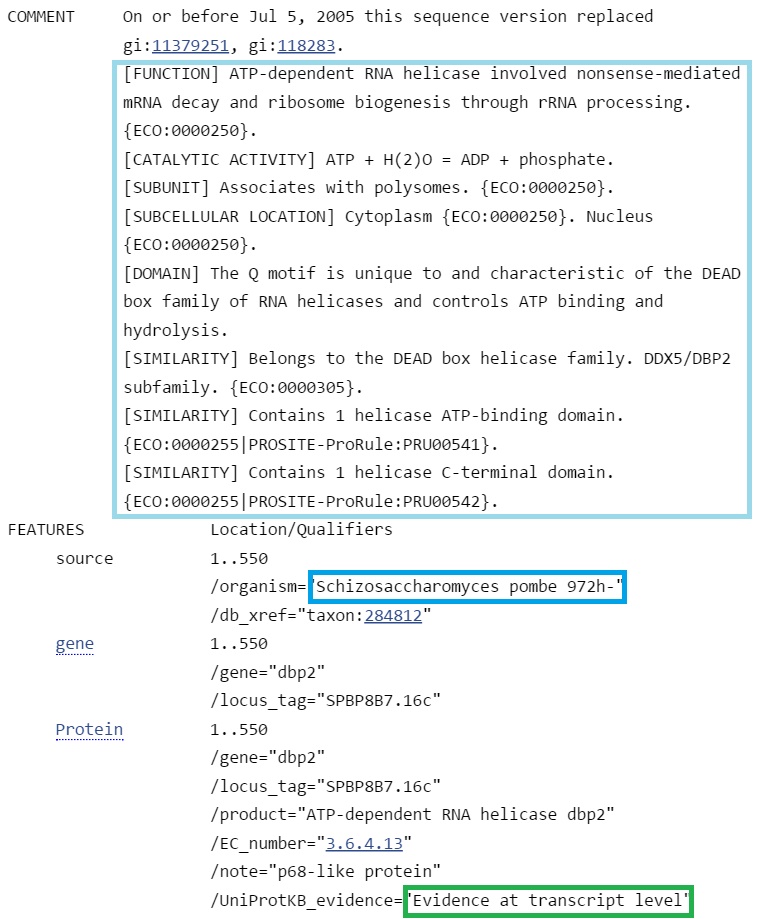
Не было находок с score более 200. Можно сделать вывод, что действительно предсказанный ген - ген, кодирующий ATP-dependent RNA helicase dbp2, однако, вероятно, AUGUSTUS ошибся в определении границ гена и экзон-интронной структуры: на самом деле белок короче предсказанного ресурсом. Таким образом, абсолютно достоверных гомологов нет.
В связи с тем, что из данного скэффолда удалось получить только 2 гена, было принято решение выбрать еще один скэффолд.
Дубль 2
Из сборки генома X5 был выбран один скэффолд (scaffold-266), длина которого составляет 47841 п.о.
infoseq с опциями -only -name -snucleotide1 –length
Последовательность этого скэффолда в формате fasta
Этап 1. Предсказание генов с помощью AUGUSTUS
Для начала необходимо выбрать организм, наиболее близкий к какому-либо организму из списка, для которого уже произведено обучение сервиса AUGUSTUS.
Для этого был использован алгоритм blastx, транслирующий поданную ему неклеотидную последовательность в 6 рамках и ищет гомологичные белки в банке данных белков. Данный алгоритм ищет именно продукт гена.
Самым близким организмом, для которого было произведено обучение сервиса AUGUSTUS - Candida tropicalis (fungus).
Страница с описанием статуса задания.
Результат работы AUGUSTUS включает 6 файлов:
o augustus.aa содержит аминокислотные последовательности белков, транслированные с найденных генов (формат fasta).
o augustus.cdsexons содержит нуклеотидные последовательности экзонов найденных в предсказанных генах (формат fasta).
o augustus.codingseq содержит кодирующие нуклеотидные последовательности генов.
o augustus.gbrowse представляет информацию, обрабатываемую геномным браузером GBrowse;
o augustus.gff содержит подробную информацию о предсказанных генах. Указаны координаты: интронов, CDS, гена, его транскрипта, старт- и стоп-кодонов.
o augustus.gtf содержит ту же информацию, что и файл augustus.gff в виде единой таблицы.
Этап 2. Проверка предсказания с помощью BLAST
Было предсказано всего 14 генов, кодирующих белки. Файл augustus.aa, выданный AUGUSTUS, содержит последовательности предсказанных белков в fasta формате.
g1, g2, g3, g4, g5 были выбраны для проверки с помощью BLAST. Предсказанная структура данных генов была взята из файла augustus.gff и обработана в Excel.
Далее для аминокислотных последовательностей предсказанных белков из файла augustus.aa проводился поиск гомологов с помощью blastp по БД Swissprot. Область поиска была ограничена таксоном Fungi.
g1
Не было найдено ни одной находки. Можно сделать вывод, что предсказание гена g1 - ошибка AUGUSTUS.
g2
Blast выдал 6 находок и все они плохие. Не было обнаружено консервативных доменов. Вывод: Предсказание ошибочно. Предсказание гена g2 - ошибка AUGUSTUS.
g3
Не было найдено ни одной находки. Таким образом, предсказание гена g3 - ошибка AUGUSTUS.
g4
Не было найдено ни одной находки. Наиболее вероятно, что предсказание гена g4 - ошибка AUGUSTUS.
g5
Не было найдено ни одной находки. Следовательно, предсказание гена g5 - ошибка AUGUSTUS.
Увы, с этими генами явно не сложилось…
Поэтому g6, g7, g8, g9, g10 были выбраны для проверки с помощью BLAST.
g6
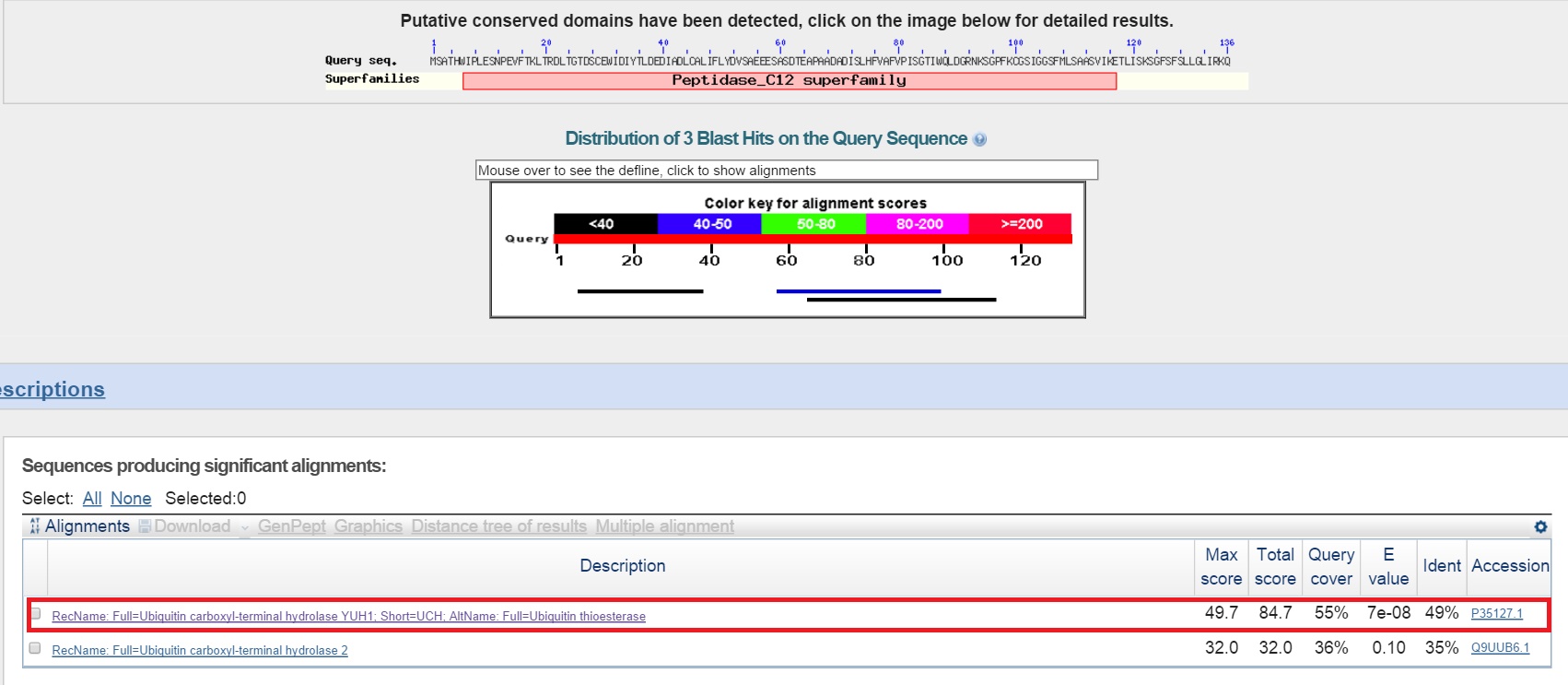
Находку с e-value 0,10 не рассматриваем. Можно сделать вывод, что ген g6 кодирует белок, который имеет слабое сходство с найденным гомологом.
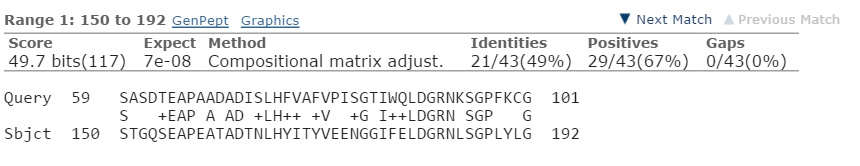
Наблюдаются отдельные участки с высоким сходством. Старт- и стоп-кодоны не совпадают.
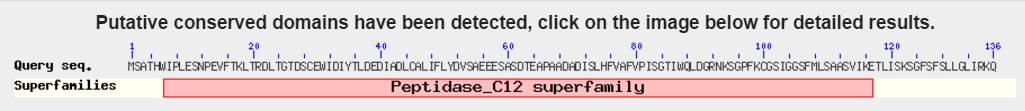
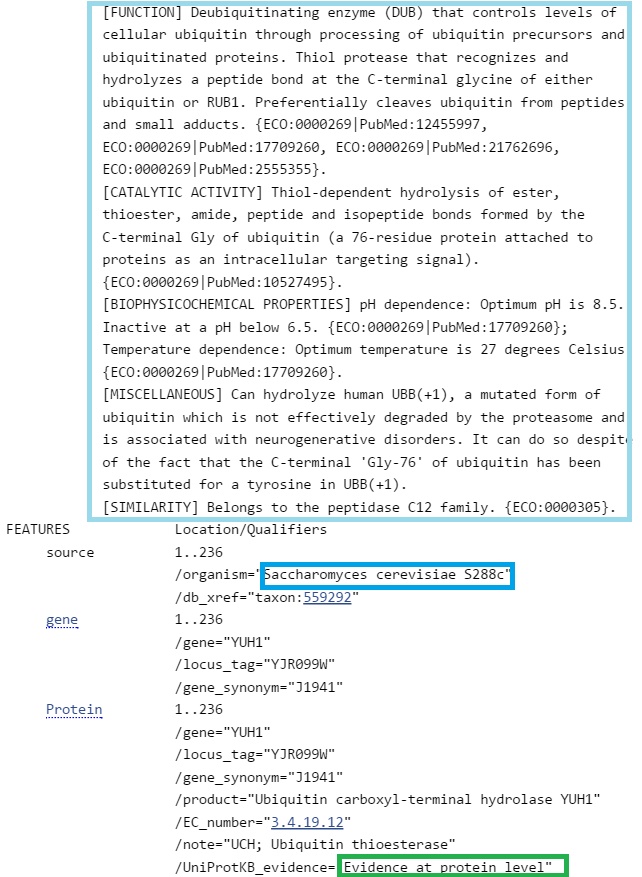
Можно сделать вывод, что предсказанный ген, вероятно, - ген, кодирующий Ubiquitin carboxyl-terminal hydrolase YUH1. Относится к семейству пептидаз C12.Наиболее вероятно, что AUGUSTUS ошибся в определении границ гена и экзон-интронной структуры.
g7
Не было найдено ни одной находки. Следовательно, предсказание гена g7 - ошибка AUGUSTUS.
g8
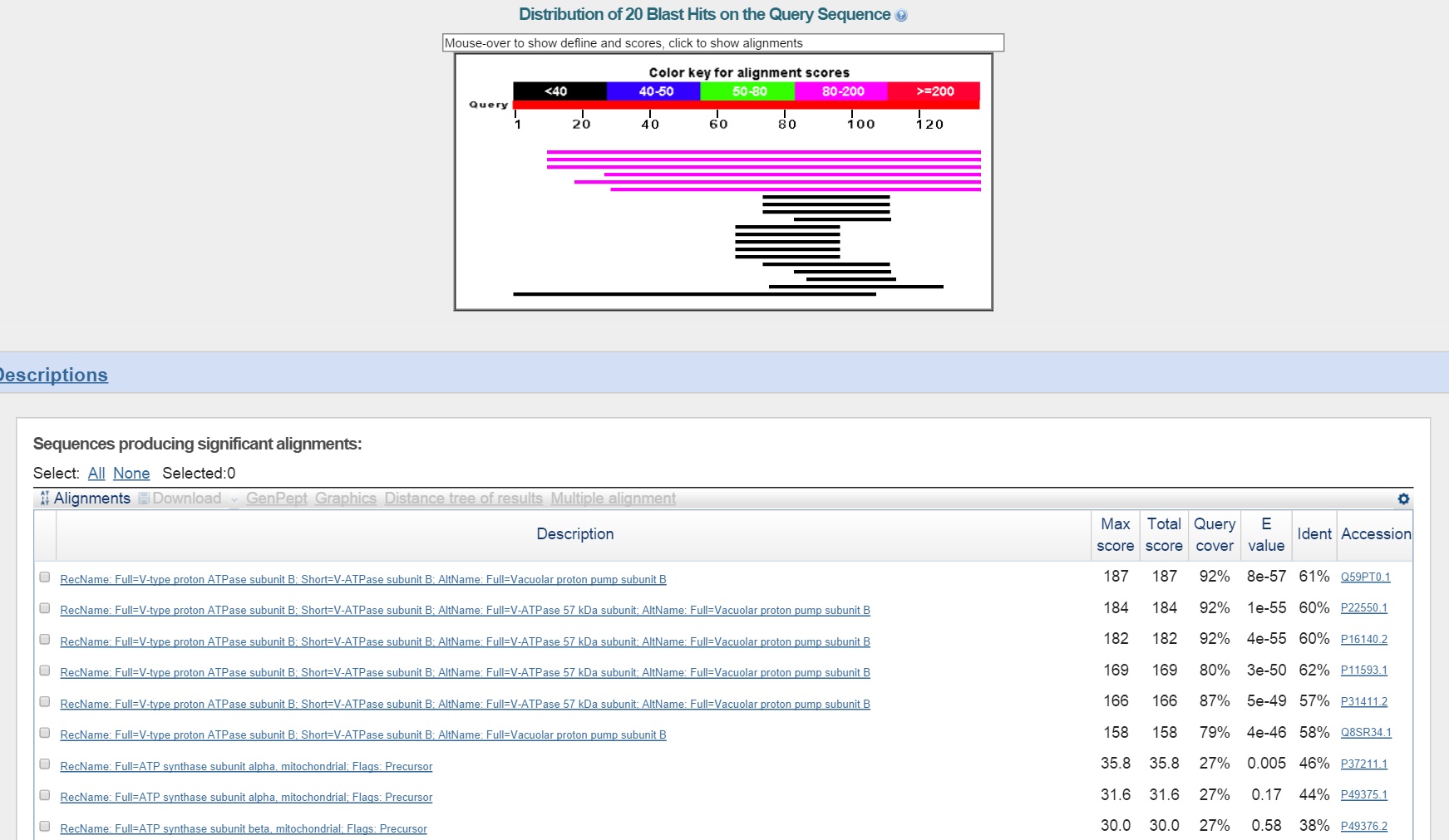
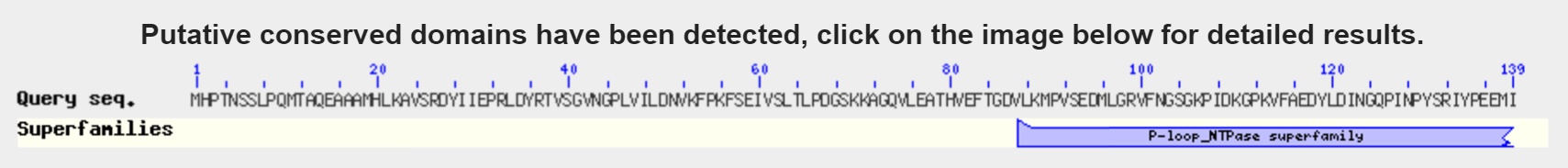
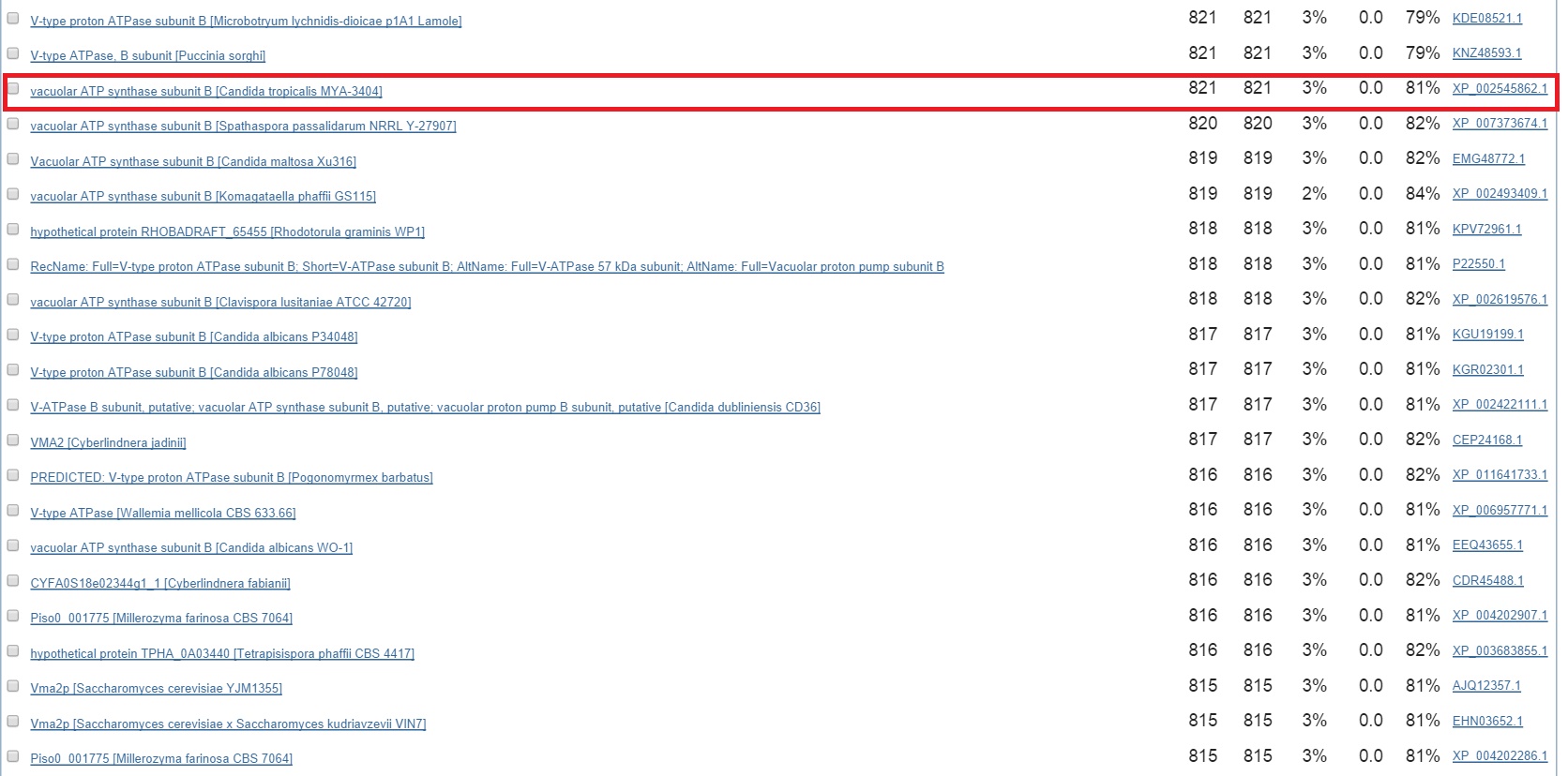
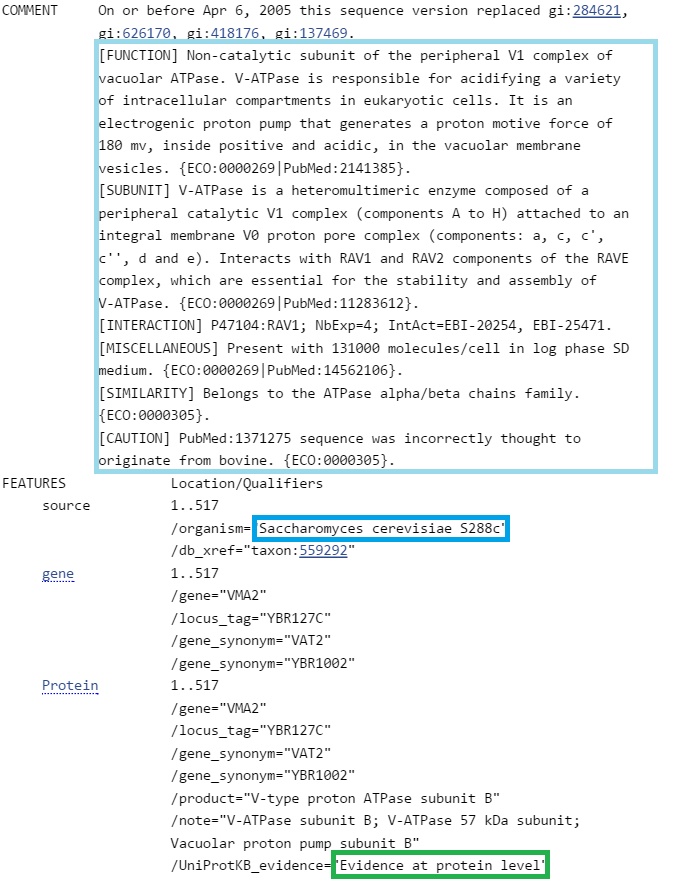
Наиболее вероятно, что g8 кодирует V-ATPase subunit B; Vacuolar proton pump subunit B (субъединицу белка АТФазы). Это каталитическая субъединица интегрального мембранного белка, осуществляющего перемещение протонов через мембрану.
Рассмотрим первые три находки.
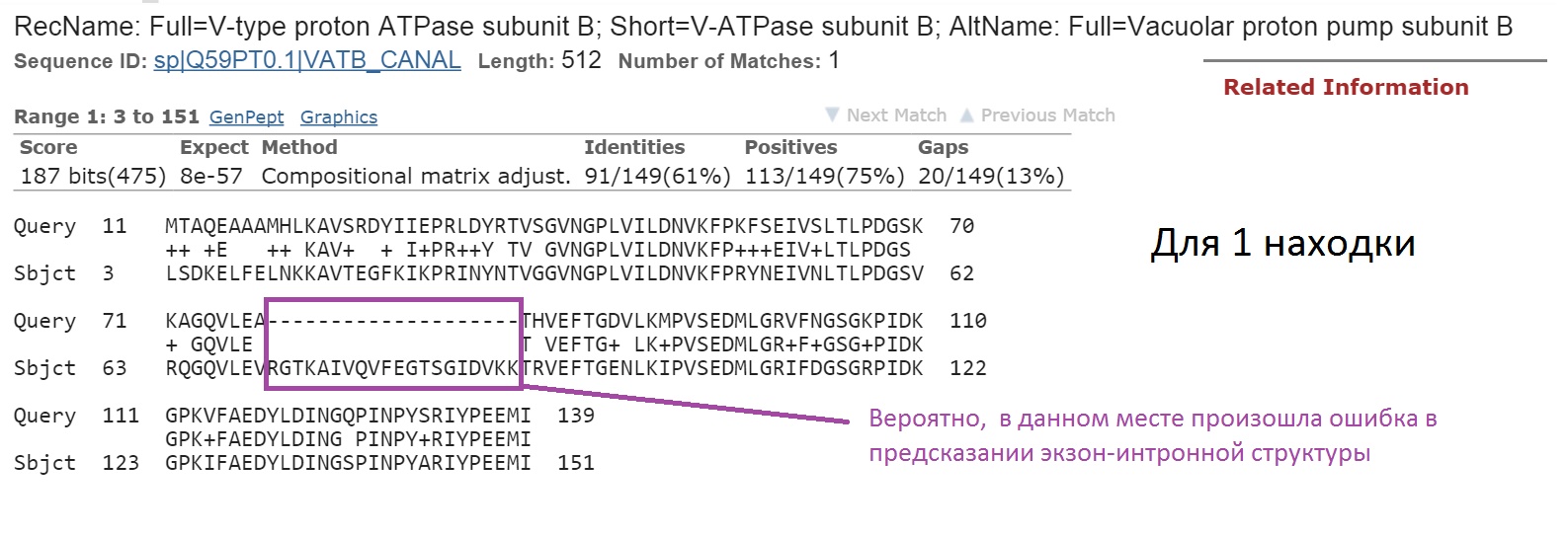

Первая находка:

Первая находка имеет подтверждение только на уровне гомологии.
Вторая:
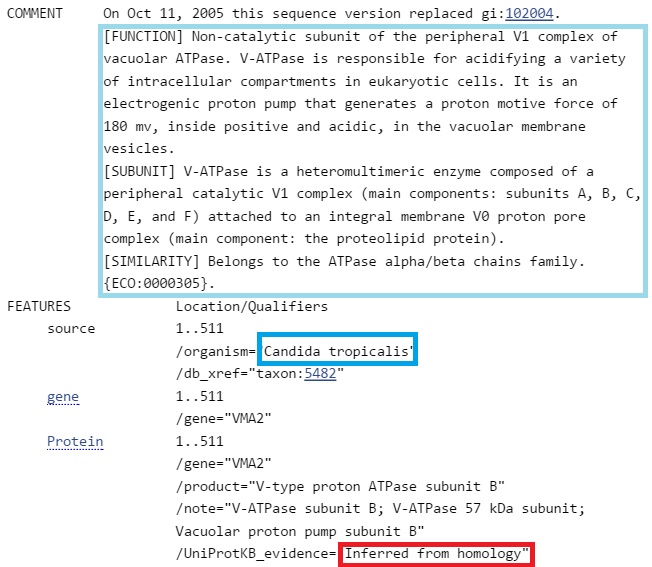
Вторая находка имеет подтверждение только на уровне гомологии.
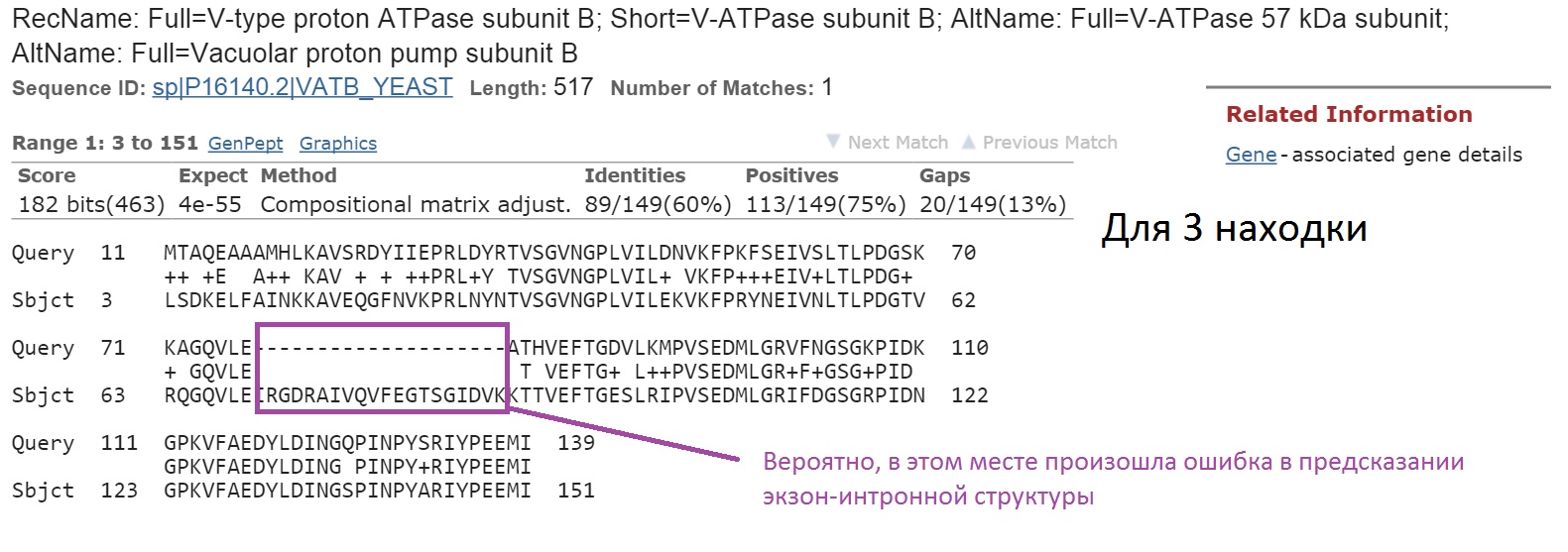
Третья:
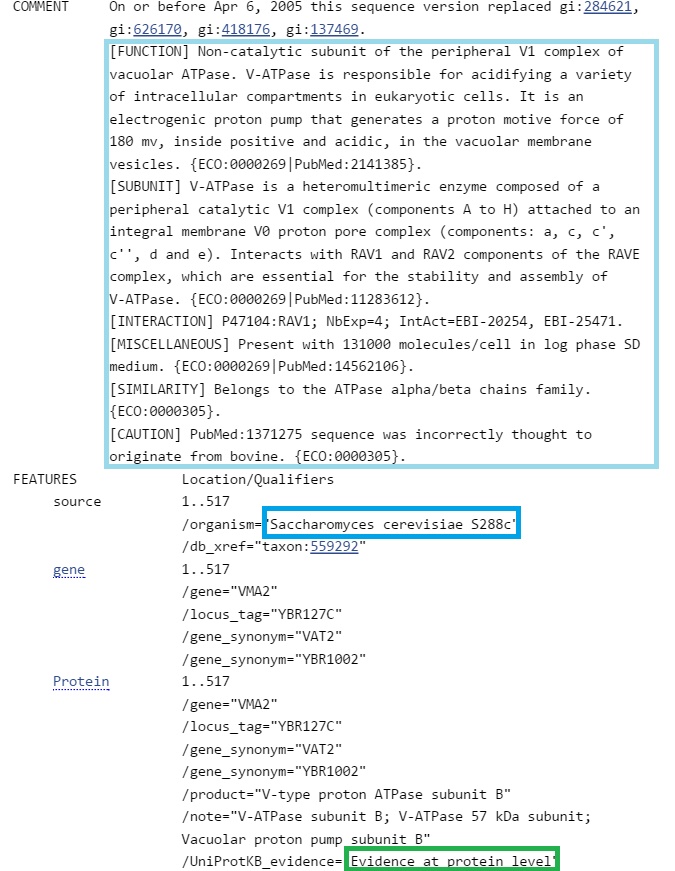
Третья находка имеет также хороший score, но зато подтверждена экспериментально.
Большое сходство последовательностей говорит о том, что найденные экзоны, скорее всего, правильные.
Гомологичные белки имеют гораздо большую длину, чем искомый. Наиболее вероятно, что AUGUSTUS ошибся в определении экзон-интронной структуры и нашел не все экзоны.
g9
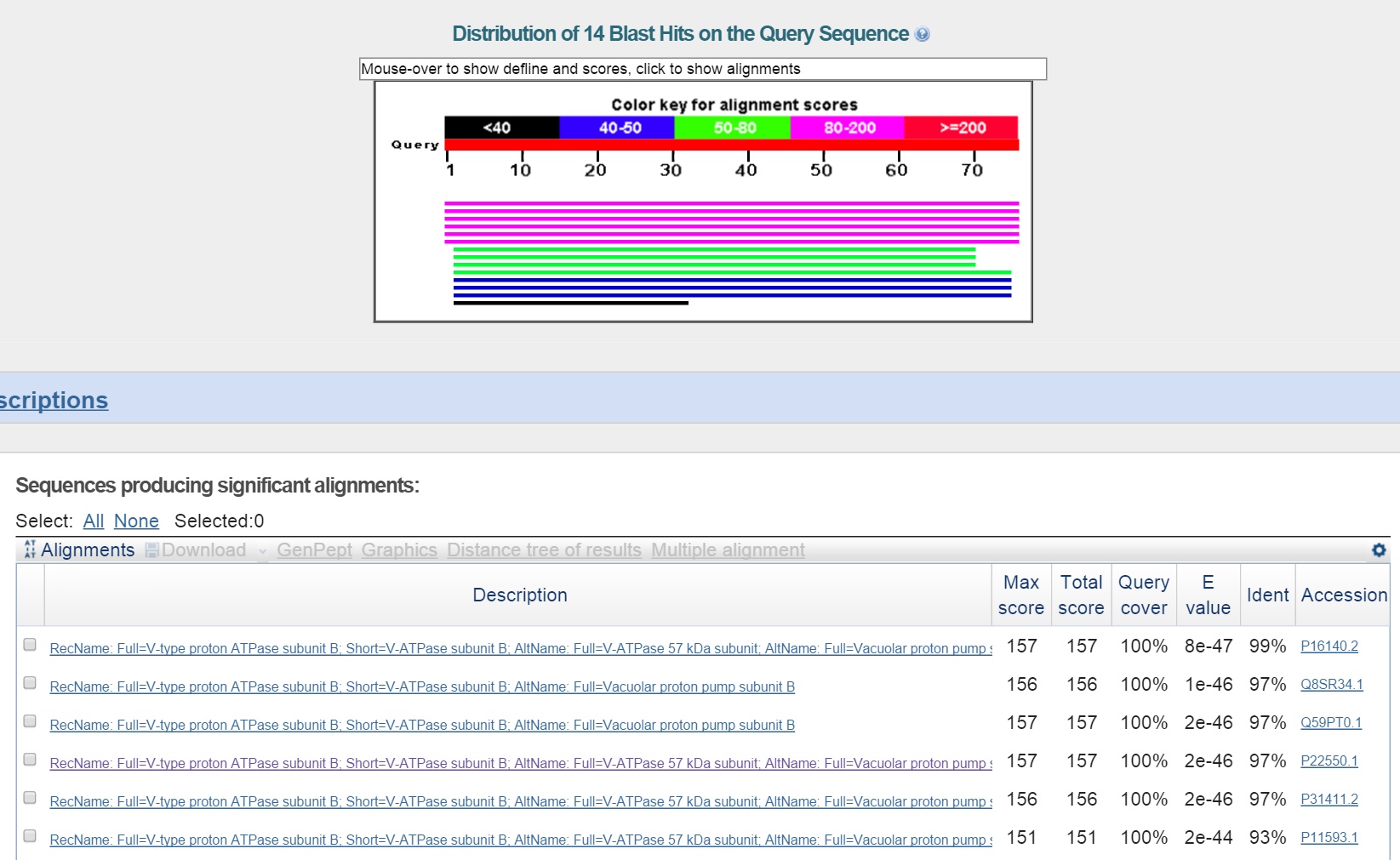
Наиболее вероятно, что g9 кодирует участок белка вакуолярной АТФазы, что подтверждается выравниванием с лучшей находкой.
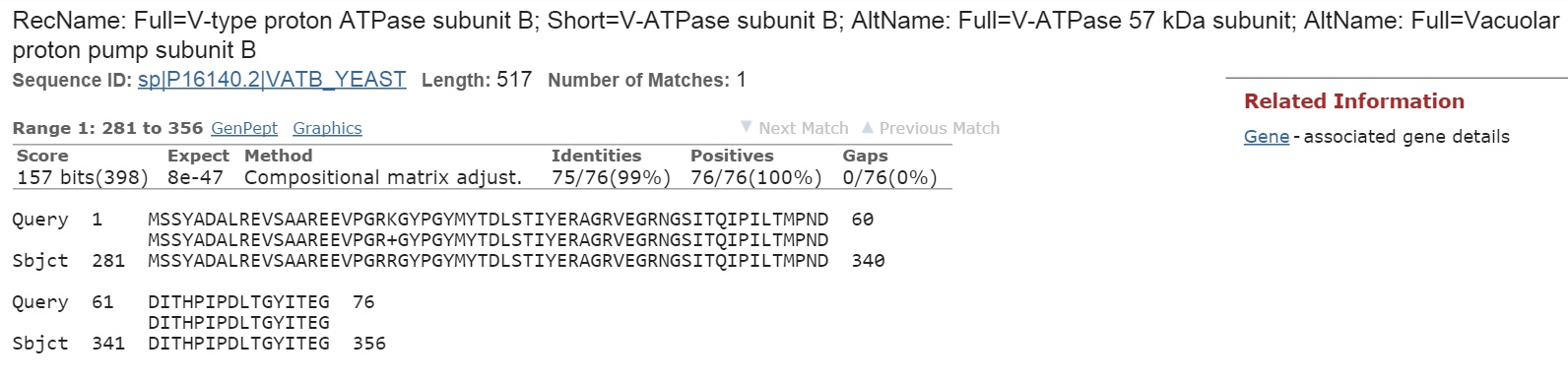
Однако, AUGUSTUS ошибся в определении границ гена.
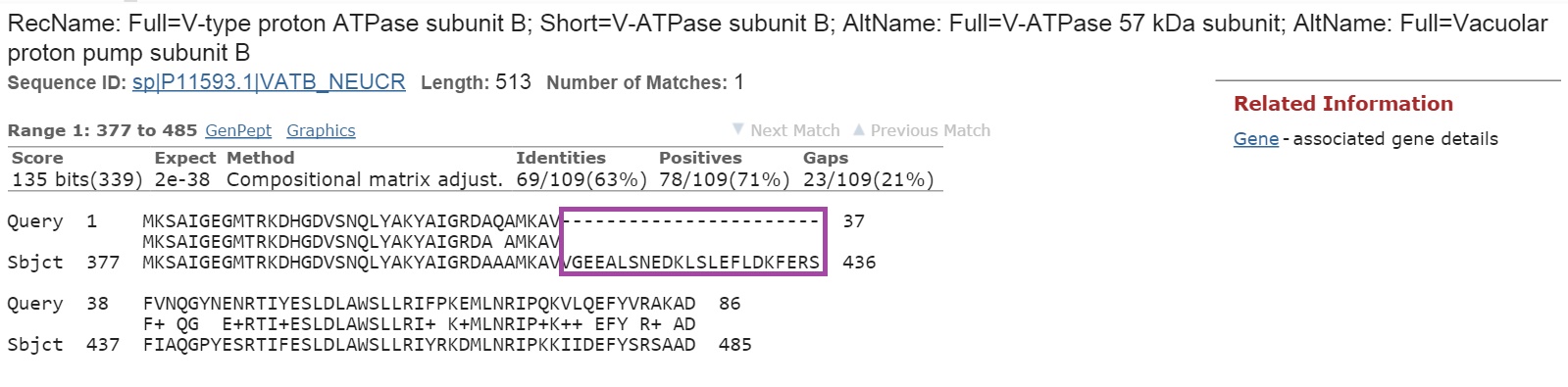
g10
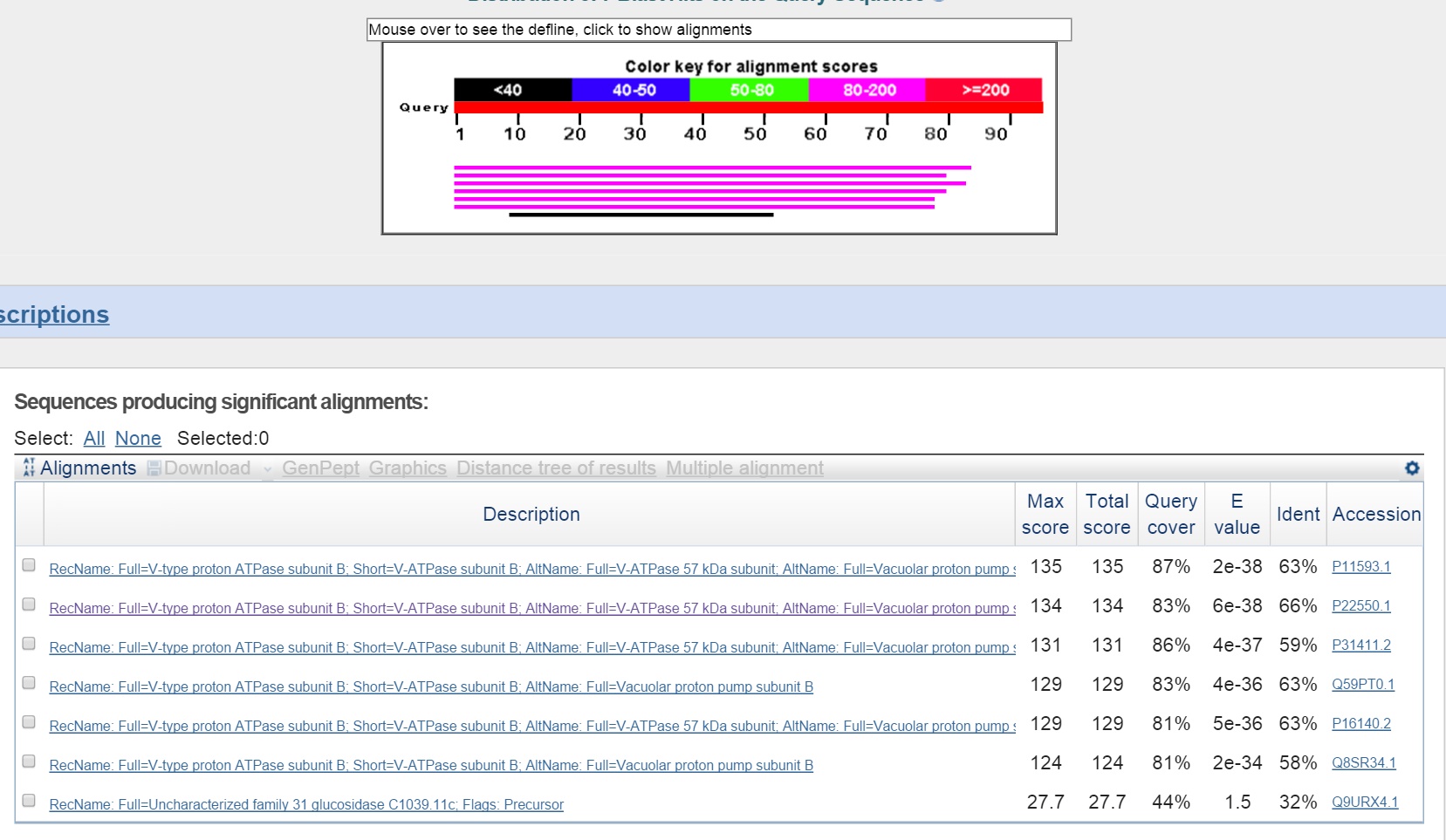

Наиболее вероятно, что g9 кодирует субъединицу B протонной вакуолярной АТФазы.
Первая находка:
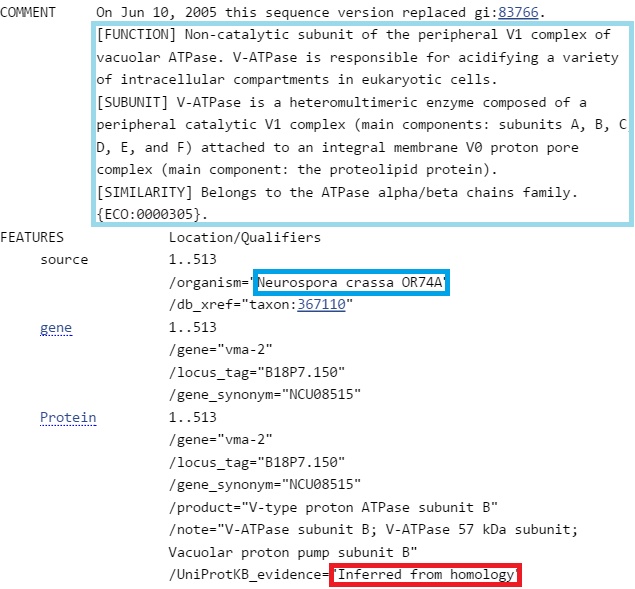
Первая находка имеет подтверждение только на уровне гомологии (как и вторая).
Третья находка:
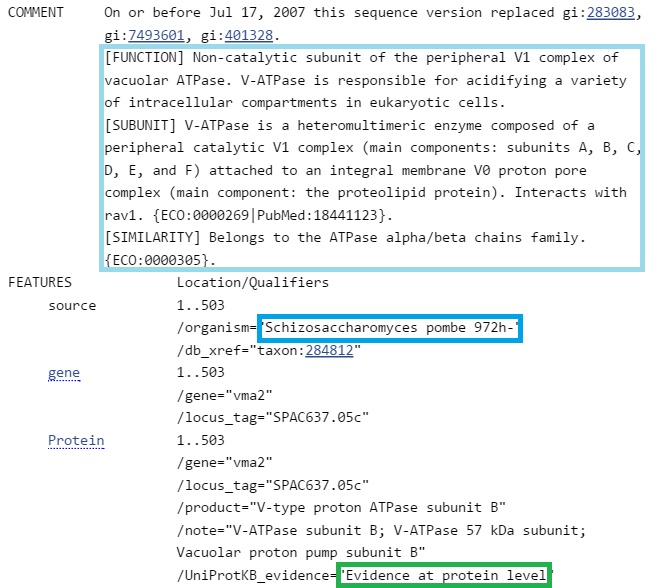
Рассмотрим выравнивания для первых трех находок:
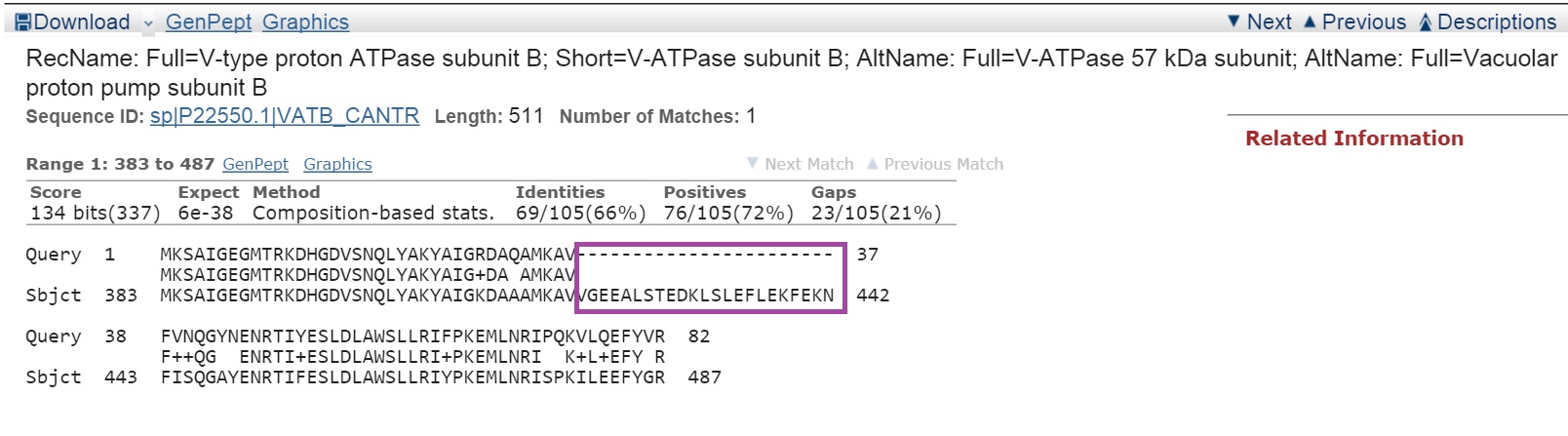
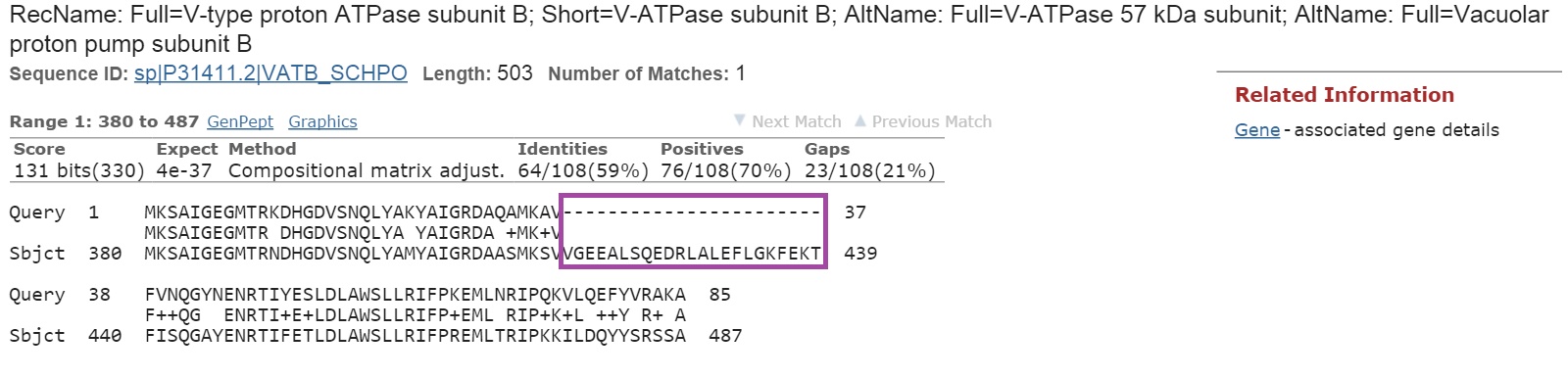
Наиболее вероятно, что AUGUSTUS ошибся в определении границ гена и экзон-интронной структуры.
Задание 2. Сравните аннотацию Refseq и AUGUSTUS одного гена человека
Был взят ген человека IRF6 (interferon regulatory factor 6).
Регулирующий интерферон фактор 6 , известный также под именем IRF6 — белок, который у человека, кодируется геном IRF6.
Регуляторные факторы интерферона содержат консервативную N-концевую область около 120 аминокислот, содержащиеся в структуре, которая специфически связывается с последовательностью интерферонного консенсуса (ICS), нкаходящегосясяперед генами интерферона. Остальные части последовательности регуляторного фактора интерферона изменяются в зависимости от точной функции белка.

По данным NCBI данный ген располагается на 1 хромосоме. Координаты: 209785623..209806175
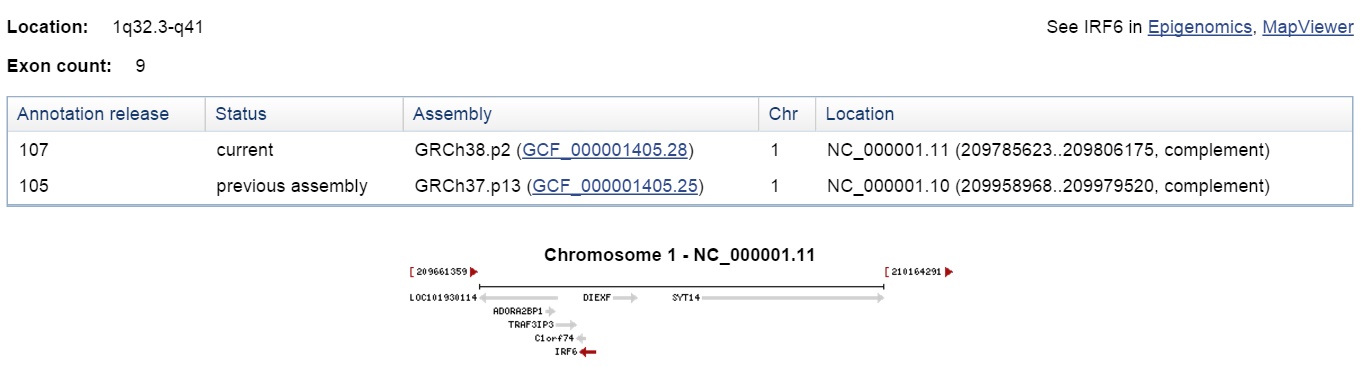
Скриншот окна браузера с двумя аннотациями гена.
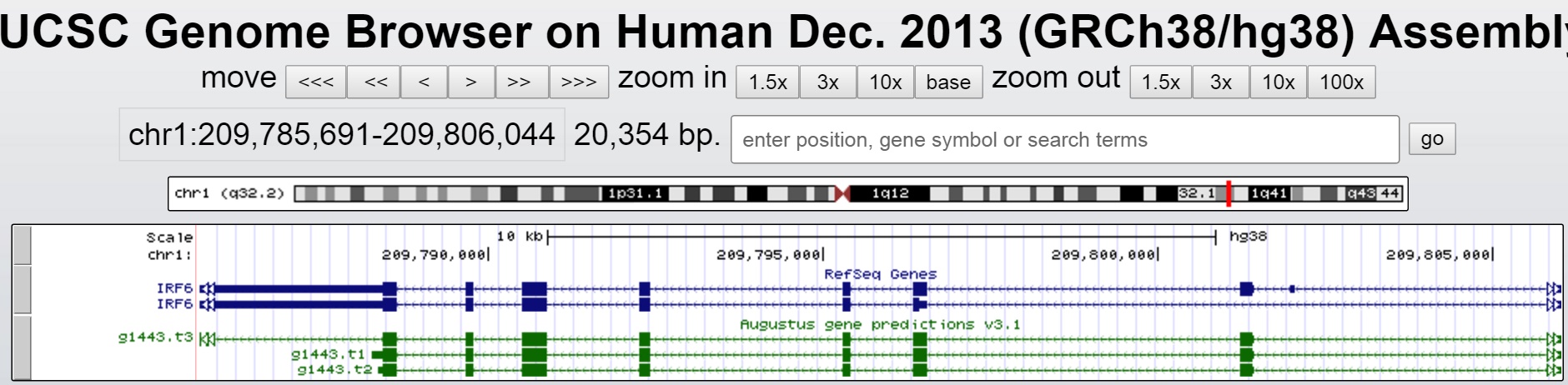
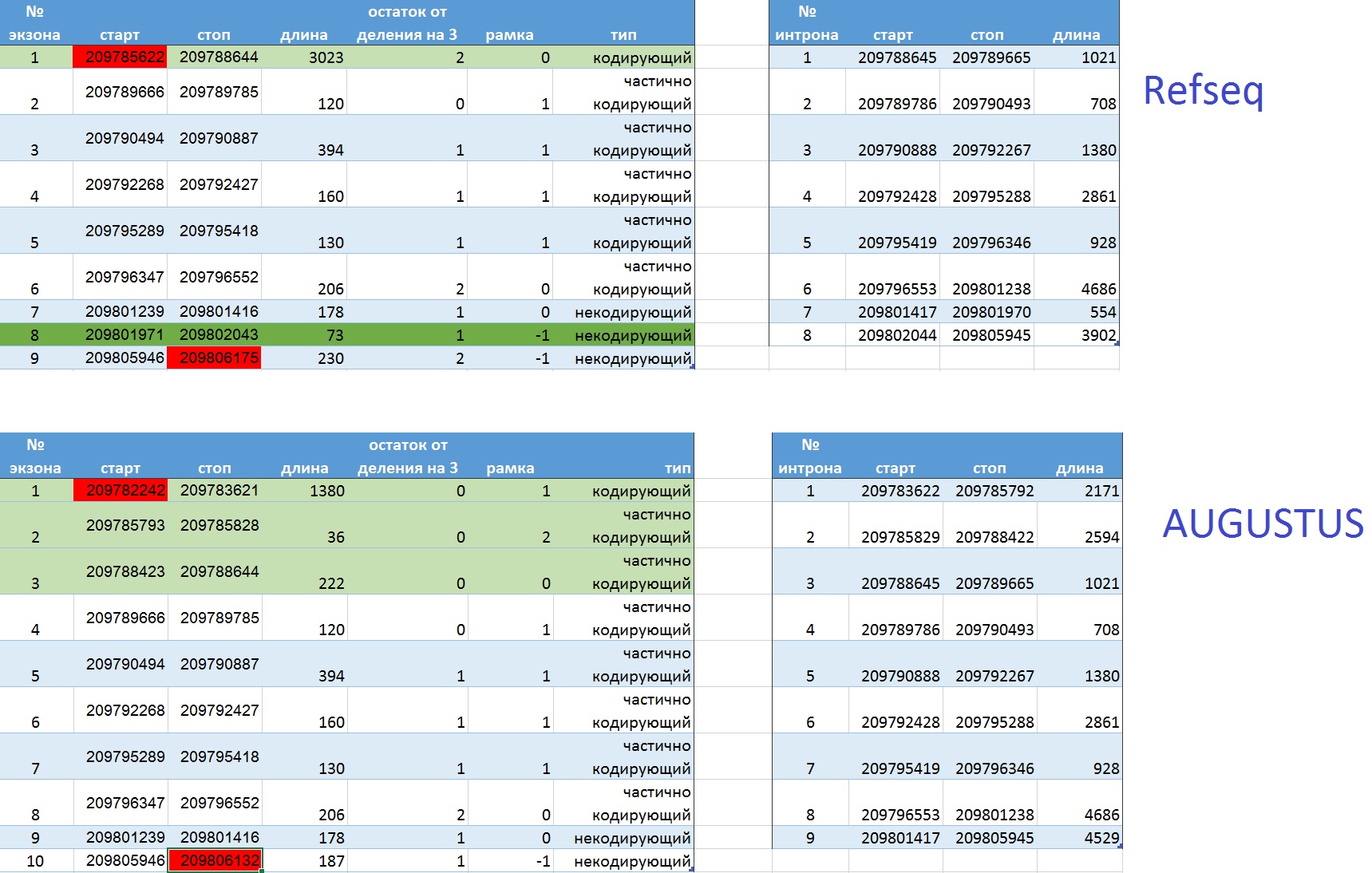
Таблица сравнения аннотаций Refseq и AUGUSTUS одного гена IRF6 (interferon regulatory factor 6).
Что хотелось бы отметить при сравнении аннотаций:
• В предсказаниях не совпадают координаты начала 1 экзона и координаты конца последнего (отличия выделены в таблице красным цветом).
• 1 экзон в аннотации Refseq разбит на три экзона в аннотации AUGUSTUS (выделены светло-зеленым)
• В аннотации Refseq указан 8 экзон, который не определил AUGUSTUS (выделен зеленым). Можно предположить, что AUGUSTUS не аннотировал этот экзон так как он, вероятно, вырезается или нет при альтернативном сплайсинге.
• В аннотации Refseq CDC не делится на 3 без остатка, так же, как и CDC для AUGUSTUS (что очень странно)
• Конец CDC у Refseq и AUGUSTUS совпадают, в отличии от начала.
• В целом аннотации сильно отличаются