Anna Zheltova
A-, B-, Z- form DNA (A-, B-, Z-формы ДНК)
Complexes of DNA-protein (Комплексы ДНК-белок)
Reading Sanger sequencing (Прочтение последовательностей по Сэнгеру)
Nucleotide databanks (Нуклеотидные банки данных)
EMBOSSAligning genomes (Выравнивание геномов)
The genes of prokaryotes (Гены прокариот)
The genes of eukaryotes (Гены эукариот)
Search for snp (Поиск полиморфизмов)
Задание 1. Определите таксономию и функцию данной нуклеотидной последовательности
Было выполнено в 7 практикуме .
Задание 2. Сравните списки находок нуклеотидной последовательности тремя разными алгоритмами
Затем был проведен поиск тремя алгоритмами blast: blastn, megablast, discontiguous megablast. Поиск был сначала ограничен Loxosomatidae, но так как при данном ограничении megablast и discontiguous megablast все еще находили по 100 находок, был изменен параметр Match/Mismatch:1,-1.
| Параметр | blastn | megablast | discontiguous megablast |
| Число находок | 18 | 16 | 16 |
| E-value худшей находки | 2.5 | 6e-179 | 0.0 |
| Query cover худшей находки | 3% | 51% | 54% |
| Сходство худшей находки | 94% | 92% | 90% |
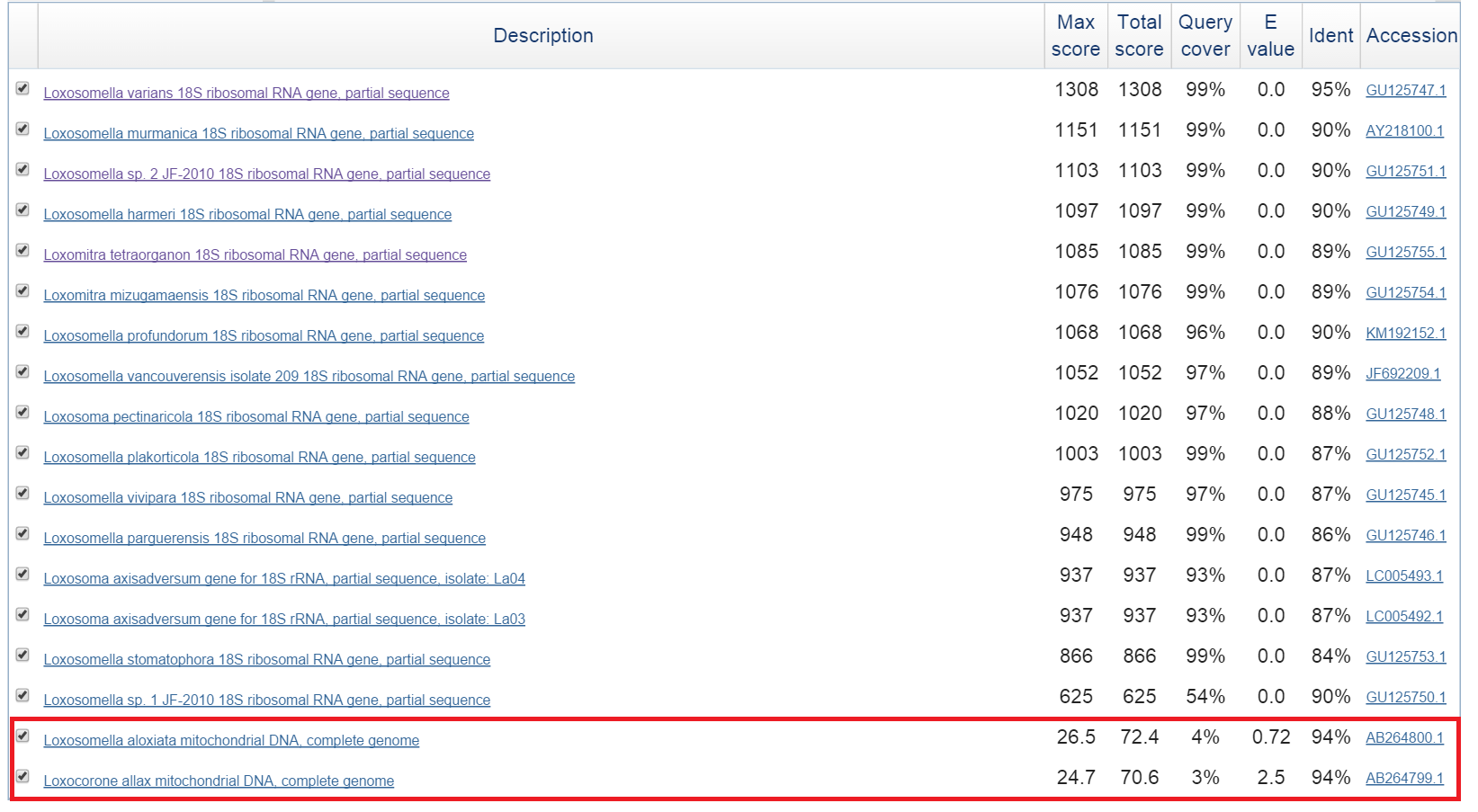
Blastn выдал на 2 находки больше, чем discontiguous megablast и megablast (данные находки выделены красной рамкой).
В частности, megablast нашел только 16 находок. discontiguous megablast нашел тоже 16 находок.
Blastn нашел на 2 находки больше, которые относятся к участкам митохондриальной: эти находки имеют худший E-value = 2.5.
Вероятно, что blastn ищет любые сходные участки, а discontiguous megablast - сходные последовательности, которые могут являться близкими гомологами. Данный пример как раз это иллюстрирует: blastn нашел сходные участки, но они совершенно точно не могут быть гомологами исходной последовательности.
3. Проверьте наличие гомологов пяти белков в геноме одного из организмов
Организм из задания 1 практикума 7 (Caenorhabditis elegans (C. elegans)); белки: HSP7C_HUMAN, TERT_HUMAN, CISY_HUMAN, RPB1_HUMAN, PABP2_HUMAN.
Был произведен поиск tblastn, база данных - refsec nucleotide. Все находки аннотированы, так как лучшая находка во всех случаях представляет собой целую хромосому.
• HSP7C_HUMAN - действует как репрессор транскрипцию активации. Подавляет транскрипцию коактиватора активности CITED1. Шаперон. Компонент комплекса PRP19-CDC5L, который образует неотъемлемую часть сплайсосоме и требуется для активации пре-мРНК сплайсинга.
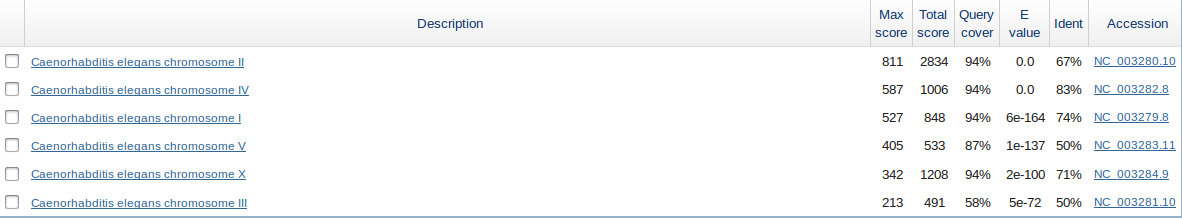
Было найдено 6 хороших находок в геноме (хромосомы I-V и Х). Все находки имеют хороший e-value.
Вывод: белки теплового шока имеют достаточно широкое распространение и являются необходимыми для жизнедеятельности клетки, что объясняет полученные результаты.
• TERT_HUMAN - теломераза - рибонуклеопротеиновый фермент, необходимый для репликации концов хромосомы у большинства эукариот. Каталитический компонент теломераз - холоферментный комплекс, чьей основной деятельностью является удлинение теломер, путем работы в качестве обратной транскриптазы. Он добавляет повторяющуюся простую последовательность на концы хромосом путем копирования последовательности шаблона внутри РНК-компонента фермента. Теломеразы играют важную роль в старении и антиапоптозе.
Находок не было найдено.
Вывод: отсутствие TERT_HUMAN в нашем организме может быть связано с наличием у C. Elegans иного сходного по выполняемым функциям белка.
• CISY_HUMAN - катализирует реакцию Acetyl-CoA + H2O + oxaloacetate = citrate + CoA в цикле трикарбоновых кислот. Этот белок участвует в 1 стадии цикла, синтезируя изоцитрат из оксалоацетата.
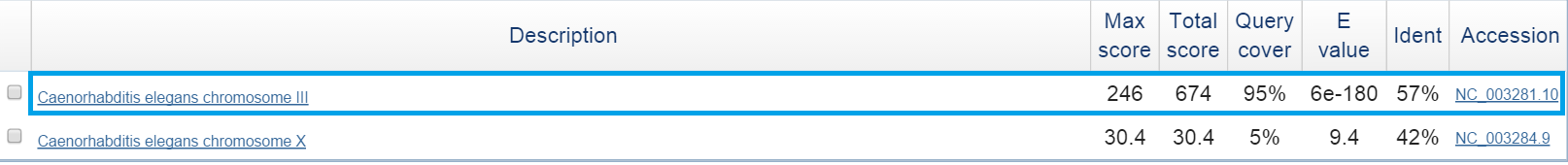
Было найдено две находки и только одна обладала достаточно хорошими характеристиками (в синей рамке).
• RPB1_HUMAN – субъединица ДНК-зависимой РНК-полимеразы, которая катализирует транскрипцию ДНК в РНК, используя четыре рибонуклеозидтрифосфата в качестве субстрата. Крупнейший и каталитический компонент РНК-полимеразы II, которая синтезирует предшественников мРНК и многие функциональные некодирующие РНК. Формирует полимеразный активный центр вместе с второй по величине субъединицей.
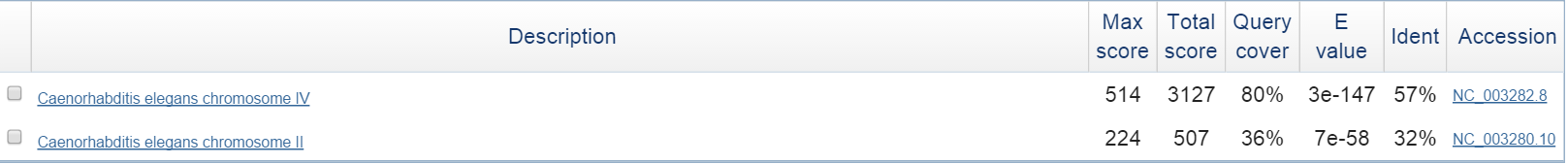
Было найдено 2 находки.
• PABP2_HUMAN - участвует в 3'-концевом формирования предшественников мРНК (пре-мРНК) путем добавления поли (А) хвоста 200-250 нуклеотидов к входному продукту расщепления. Стимулирует поли (А) полимеразы (Papola) пришивать поли(А) конец. Увеличивает сродство поли (А) полимеразы к РНК. Также присутствует на различных этапах метаболизма мРНК. Может защитить поли (А) хвост от деградации.

Обнаружено 3 находки с не очень хорошим Query cover.
4. Классифицируйте геномы родственных вирусов по сходству последовательностей
Были выбраны 5 геномов:
Potato virus Y strain nnp (AF237963.2)
Potato virus Y strain N-Egypt (AF522296.1)
Potato virus Y strain O (EF026074.1)
Potato virus Y strain N:O (EF026076.1)
Potato virus Y strain NTN (EF026075.1)
Была создана таблица с сортированными по «bit» значениями. Далее она была обработана.
Выводы: все сравниваемые геномы сходны в той или иной степени, что не удивительно, так как сравнение производилось между пятью штаммами одного и того же вируса. Наибольшим сходством обладают геномы Potato virus Y strain N-Egypt (AF522296.1) и Potato virus Y strain O (EF026074.1). Наименьшим сходством среди рассматриваемых геномов обладают геномы Potato virus Y strain nnp (AF237963.2) и Potato virus Y strain NTN (EF026075.1).