| Учебный сайт Саши Погорельской |

|
|
||||||
| Главная | Семестры | Скрипты | Обо мне | Ссылки | ||
|
|
Однонуклеотидные полиморфизмы, индели и сборка В этом практикуме анализировалось картирование чтений на геном митохондрий и хлоропласта резуховидки, выполненное в прошлом задании. Для поиска количества полиморфизмов и инделей нужен файл в формате bfc. Создаие файла в несжатом формате bfc выполняется с помощью опции -u, опция -f отвечает за референсную последовательность:
Подсчет количества полиморфизмов и инделей:
В итоге, инделей 283, а полиморфизмов 674. Полученный файл в формате .vcf можно скачать здесь. Для сборки генома использовался алгоритм velvet. В первую очередь, надо создать базу k-меров (команда velveth), а потом эти k-меры объединяются в контиги (команда velvetg):
Я попробовала сборку для разной длины k-меров, наилучший показатель N50 составил 246 нуклеотидов, то есть 50% генома покрыто контигами длиннее 246 нуклеотидов. Это значение для k-меров длины 23. Десять самых длинных контигов принадлежат митохондриальному геному, их порядковые номера и длина указаны в таблице 1. Для определения принадлежности использовался локальный blast. Таблица 1. Десять наиболее длинный контигов сборки 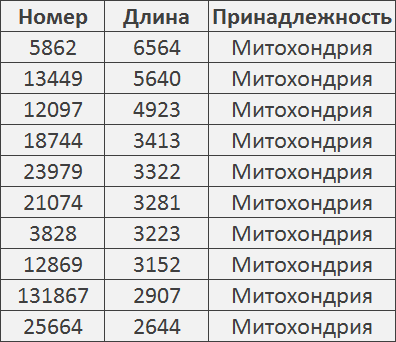
|
||||||||||||||||||||||
| © Pogorelskaya Sasha | Last modification date: 19.02.15 | |||||||||||||||||||||||