| Учебный сайт Саши Погорельской |

|
|
||||||
| Главная | Семестры | Скрипты | Обо мне | Ссылки | ||
|
|
Локальный BLAST С помощью FTP-сервера NCBI и программ на сервере kodomo, я получила последовательности некодирующих РНК (misc-RNA) бактерии Bacillus_subtilis_168_uid57675. Их можно посмотреть здесь. С того же сервера был взят полный геном штамма Bacillus_subtilis_spizizenii_TU_B_10_uid73967. Программой standalone megablast были найдены гомологи 62 misc-RNA в геноме второго штамма. В таблице приведены координаты начала и конца лучшего гомолога каждой misc-RNA и E-value выравниваний. Дальше я искала гомологи этих РНК в другой бактерии: Bacillus cereus, штамм Q1. Для сравнения работы нескольких алгоритмов, как это делалось для предыдущего практикума, я искала гомлоги с помощью megablast, blastn с параметрами по умолчанию и blastn с длиной слова 4, наградой за совпадение 1 и штрафом за несовпадение –1. Результаты приведены в этой таблице. Для каждой misc-RNA и каждого алгоритма подсчитано количесвто найденных гомологов с E-value меньше 0,001. Для последнего задания использовался вновь геном бактерии Bacillus_subtilis_spizizenii_TU_B_10_uid73967, а точнее последовательности предсказанных белков. С помощью программы blastx, которая транслирует нуклеотидную последовательность и ищет гомологи в белковой базе данных, я искала гомологи misc-RNA из первых заданий. Для выбора подходящего для задания порога E-value, я подсчитала количество находок. Эти результаты представлены в таблице 1. Таблица 1. Подбор порога E-value 
Значения E-value 1 и 0,5 на мой взгляд довольно большие для гоологичных последовательностей, их я проверила для сравнения. Результаты для более низких значений (0,1 и 0,01) отличаются незначительно, поэтому я решила проверить все 13 находок с порогом E-value 0,1. Для каждой находки в таблице 2 указано E-value, название белка и номер предположительно гомологичной misc-RNA. Таблица 2. Результаты blastx 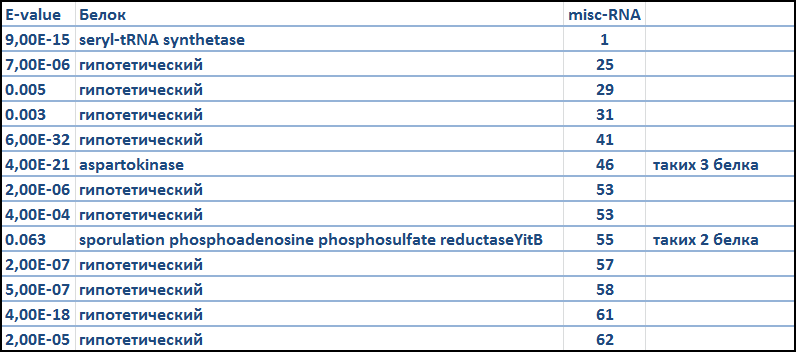
Из 13 находок аннотированно только три белка, при чем названия двух из них встречаются в полной коллекции белков бактерии не по одному разу. |
|||||||||||||||
| © Pogorelskaya Sasha | Last modification date: 19.02.15 | ||||||||||||||||