Занятие 7: BLAST
При выполнении заданий использовался web-интерфейс к BLASTp на сервере NCBI.
Поиск гипотетических гомологов белка CDD_BACSU в разных банках
C помощью программы BLASTp произведём поиск гомологов белка CDD_BACSU
(AC P19079) в банках
(Лучшая находка, как и ожидалось, совпала с заданным для поиска белком. Поэтому будем рассматривать поисковую выдачу сервиса, начиная со второй находки.
Более того, при поиске в банках PDB и nr последующие 3 находки в PDB и 5 находок в nr соответствуют искомому белку CDD_BACSU с точечной мутацией; их идентификаторы в базах данных: 1UX0, 1UWZ, 1UX1 в PDB и ZP_06875765, 1UX0, 1UWZ, BAI86051, 1UX1 в nr.)
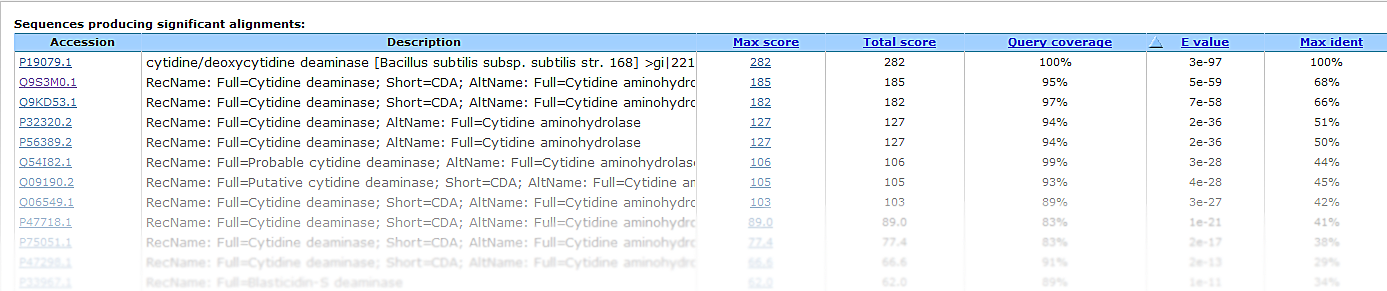
Таблица 1. Результаты поиска гипотетических гомологов белка CDD_BACSU
| Искомая информация | Поиск по |
Поиск по PDB | Поиск по nr |
|
|||
| Accession | Q9S3M0 | 2D30 | YP_001421951 |
| E-value | 5e-59 | 3e-66 | 2e-88 |
| Вес (в битах) | 185 | 201 | 264 |
| Процент идентичности | 68% | 70% | 91% |
|
12 | 12 | 100 |
|
|||
| Номер находки в списке описаний | 85 | 21 | 100 |
| Accession | Q6D3B4 | 3IIP | ZP_09679236 |
| E-value | 5e-04 | 0.98 | 1e-52 |
| Вес (в битах) | 41.6 | 28.9 | 173 |
| Процент идентичности | 30% | 52% | 59% |
| Процент сходства | 45% | 71% | 79% |
| Длина выравнивания | 111 | 21 | 131 |
| Координаты выравнивания | В запросе: 22 - 130. В находке: 69 - 170. |
В запросе: 25 - 45. В находке: 6 - 26. |
В запросе: 3 - 133. В находке: 21 - 150. |
| Число гэпов | 11 | 0 | 1 |
-
Следует также обратить внимание на следующие моменты:
- исходный белок CDD_BACSU удалось найти в
Swiss-Prot и nr, а его структуру - в PDB; - число явных гомологов (E-value < 1e-10) при поиске по базе данных nr заметно больше, чем при поиске в других базах данных
(если снять ограничение в 100 отображаемых результатов, то мы сможем найти 1720 явных гомологов); это вполне объяснимо, если учесть тот факт,
что nr включает в себя все белковые последовательности (17612906 последовательностей по состоянию
на 24 марта 2012 года; это на 2 порядка больше, чем 450602 последовательностей в
Swiss-Prot, и на 3 порядка больше, чем 57172 в PDB) из всевозможных источников (а именно: All non-redundant GenBank CDS translations+ PDB+SwissProt+PIR+PRF excluding environmental samples from WGS projects); - общее количество находок (при параметрах по умолчанию) - 91, 28 и 100 в
Swiss-Prot, PDB и nr соответственно; в первых двух случаях число находок было лимитировано значением E-value (равным соответственно 9.0 и 4.2 для последней находки), в последнем, как уже было отмечено, - заданным по умолчанию предельным размером выдачи (если в последнем случае повысить предельный размер выдачи, то мы получим 2426 находок).
Поиск гипотетических гомологов белка CDD_BACSU с фильтром по таксонам
Таксономия Bacillus subtilis:
› Bacteria
› Firmicutes
› Bacilli
› Bacillales
› Bacillaceae
› Bacillus
› Bacillus subtilis
Для поиска лучшего гомолога в организмах таксона, филогенетически как можно более далекого, предлагаются следующие таксоны:
› Eukaryota - другое царство
› Actinobacteria - другой отдел царства Bacteria
› Clostridia - другой класс отдела Firmicutes
› Lactobacillales - другой порядок класса Bacilli
› Listeriaceae - другое семейство порядка Bacillales
› Geobacillus - другой род семейства Bacillaceae
› Bacillus anthracis - другой вид рода Bacillus
Установленный критерий наличия гипотетического гомолога - E-value < 0,001.
При выполнении поиска в порядке приближения к Bacillus subtilis первый гомолог найден в царстве Eukaryota. Это оказалась цитидин-дезаминаза человека.
| Искомая информация | Значение |
| Номер находки в списке описаний | 1 |
| Accession | P32320 |
| E-value | 3e-40 |
| Вес (в битах) | 127 |
| Процент идентичности | 51% |
| Процент сходства | 70% |
| Длина выравнивания | 129 |
| Координаты выравнивания | В запросе: 4 - 131. В находке: 16 - 143. |
| Число гэпов | 2 |
Сравнение выравниваний, выданных программой BLASTp, с оптимальными глобальным и локальным выравниваниями
Для сравнения используем выравнивание BLASTp последовательностей белка CDD_BACSU (AC P19079) и белка CDD_HUMAN (AC P32320), которое было получено при выполнении предыдущего задания.
>sp|P32320.2|CDD_HUMAN RecName: Full=Cytidine deaminase; AltName: Full=Cytidine aminohydrolase Length=146 GENE ID: 978 CDA | cytidine deaminase [Homo sapiens] (Over 10 PubMed links) Score = 127 bits (320), Expect = 3e-40, Method: Compositional matrix adjust. Identities = 66/129 (51%), Positives = 90/129 (70%), Gaps = 2/129 (2%) Query 4 QELITEALKARDMAYAPYSKFQVGAALLTKDGKVYRGCNIENAAYSMCNCAERTALFKAV 63 Q+L+ + +A+ AY PYS F VGAALLT++G++++GCNIENA Y + CAERTA+ KAV Sbjct 16 QQLLVCSQEAKKSAYCPYSHFPVGAALLTQEGRIFKGCNIENACYPLGICAERTAIQKAV 75 Query 64 SEGDTEFQMLAVAADTPGP-VSPCGACRQVISELCTKDVIVVLTNLQGQIKEMTVEELLP 122 SEG +F+ +A+A+D +SPCGACRQV+ E T + V +T G MTV+ELLP Sbjct 76 SEGYKDFRAIAIASDMQDDFISPCGACRQVMREFGT-NWPVYMTKPDGTYIVMTVQELLP 134 Query 123 GAFSSEDLH 131 +F EDL Sbjct 135 SSFGPEDLQ 143
- Функция веса, использованная при построении:
- матрица весов замен - BLOSUM62;
- штраф за создание гэпа - 11;
- штаф за удлинение гэпа - 1.
Программа needle пакета EMBOSS выдаёт оптимальное полное выравнивание.
Для построения оптимального полного выравнивания последовательностей белков CDD_BACSU (AC P19079) и CDD_HUMAN (AC P32320) при той же функции веса выполним следующую команду:
needle sw:p19079 sw:p32320 cdd_bacsu-cdd_human.needle -gapopen 11 -gapextend 1
В результате получим файл cdd_bacsu-cdd_human.needle.
Полученное программой needle оптимальное полное выравнивание:
CDD_BACSU 1 ------------MNRQELITEALKARDMAYAPYSKFQVGAALLTKDGKVY 38 ...|:|:..:.:|:..||.|||.|.|||||||::|::: CDD_HUMAN 1 MAQKRPACTLKPECVQQLLVCSQEAKKSAYCPYSHFPVGAALLTQEGRIF 50 CDD_BACSU 39 RGCNIENAAYSMCNCAERTALFKAVSEGDTEFQMLAVAADTPGP-VSPCG 87 :|||||||.|.:..||||||:.||||||..:|:.:|:|:|.... :|||| CDD_HUMAN 51 KGCNIENACYPLGICAERTAIQKAVSEGYKDFRAIAIASDMQDDFISPCG 100 CDD_BACSU 88 ACRQVISELCTKDVIVVLTNLQGQIKEMTVEELLPGAFSSEDLHDERKL 136 |||||:.|..| :..|.:|...|....|||:||||.:|..|||...: CDD_HUMAN 101 ACRQVMREFGT-NWPVYMTKPDGTYIVMTVQELLPSSFGPEDLQKTQ-- 146
Для построения оптимального частичного выравнивания используем программу water пакета EMBOSS. Выполним команду:
water sw:p19079 sw:p32320 cdd_bacsu-cdd_human.water -gapopen 11 -gapextend 1
В результате получим файл cdd_bacsu-cdd_human.water.
Полученное программой water частичное выравнивание:
CDD_BACSU 4 QELITEALKARDMAYAPYSKFQVGAALLTKDGKVYRGCNIENAAYSMCNC 53 |:|:..:.:|:..||.|||.|.|||||||::|::::|||||||.|.:..| CDD_HUMAN 16 QQLLVCSQEAKKSAYCPYSHFPVGAALLTQEGRIFKGCNIENACYPLGIC 65 CDD_BACSU 54 AERTALFKAVSEGDTEFQMLAVAADTPGP-VSPCGACRQVISELCTKDVI 102 |||||:.||||||..:|:.:|:|:|.... :|||||||||:.|..| :.. CDD_HUMAN 66 AERTAIQKAVSEGYKDFRAIAIASDMQDDFISPCGACRQVMREFGT-NWP 114 CDD_BACSU 103 VVLTNLQGQIKEMTVEELLPGAFSSEDL 130 |.:|...|....|||:||||.:|..||| CDD_HUMAN 115 VYMTKPDGTYIVMTVQELLPSSFGPEDL 142
Таблица сравнения полученных выравниваний
| Признак сравнения | BLASTp | needle (оптимальное полное) | water (оптимальное локальное) |
| Вес | 320 | 307 | 316 |
| Процент идентичности | 51% | 44.3% | 51.6% |
| Процент сходства | 70% | 61.1% | 70.3% |
| Число гэпов | 2 | 16 | 2 |
| Длина выравнивания | 129 | 149 | 128 |
| Координаты выравнивания | В запросе: 4 - 131. В находке: 16 - 143. |
В запросе: 1 - 136. В находке: 1 - 146. |
В запросе: 4 - 130. В находке: 16 - 142. |
Мерой совпадения двух выравниваний служит процент согласованных колонок первого (аналогично, второго) выравнивания относительно общего числа колонок.
Рассчитаем меру совпадения локального выравнивания, полученного программой BLAST, и оптимального глобального выравнивания последовательностей тех же белков.
При этом ограничимся выравниванием фрагментов, входящих в оба выравнивания (в запросе: 4 - 131; в находке: 16 - 143).
Число согласовынных колонок для выбранных фрагментов - 129. Общее число колонок первого выравнивания - 129, второго выравнивания (для указанного фрагмента) - 129.
Мера согласованности равна 100% для первого выравнивания и 100% для второго выравнивания.
Таким образом, все сопоставления в двух выравниваниях одинаковы.
Выбранный фрагмент оптимального глобального выравнивания составляет 86,6% от общей длины этого глобального выравнивания.
Аналогично рассчитаем меру совпадения локального выравнивания, полученного программой BLAST, и оптимального локального выравнивания последовательностей тех же белков.
При этом ограничимся выравниванием фрагментов, входящих в оба выравнивания (в запросе: 4 - 130; в находке: 16 - 142).
Число согласовынных колонок - 128. Общее число колонок первого выравнивания (для выбранного фрагмента) - 128, второго выравнивания - 128.
Мера согласованности равна 100% для первого выравнивания и 100% для второго выравнивания.
Таким образом, все сопоставления в двух выравниваниях одинаковы.
Выбранный фрагмент локального выравнивания, полученного программой BLAST, составляет 99,2% от общей длины этого локального выравнивания.
Дополнение страницы описаний программ
Раздел пакет EMBOSS страницы с описаниями программ дополнен описаниями следующих программ:
seqret matcher stretcher needle waterПоиск с альтернативными параметрами BLAST
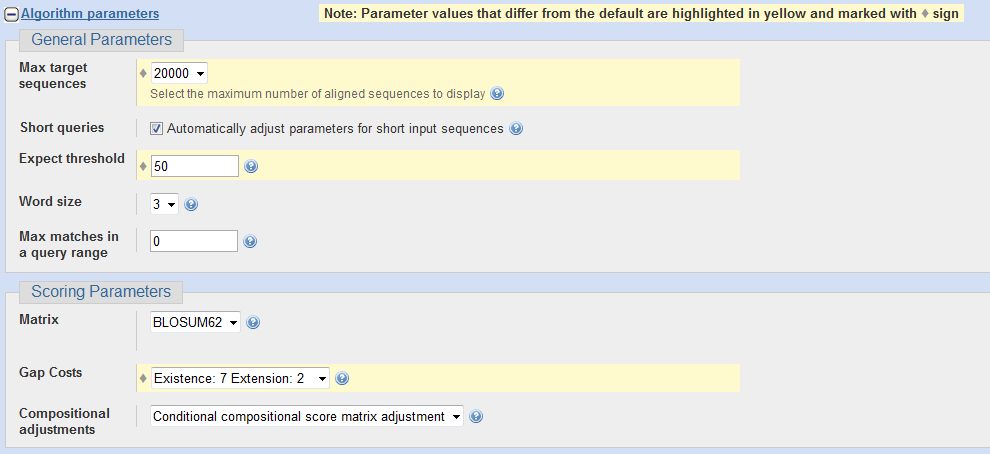
При поиске, например, в базе данных nr изменим некоторые параметры (лимит количества находок, максимальное значение E-value, а также штрафы за гэпы). В результате получим 2487 находок с наибольшим значением E-value 49. Таким образом, в данном случае число находок будет лимитировано значением E-value. Первая находка по-прежнему будет совпадать с искомым белком.
Рассуждения о гомологии находки с E-value > 1
E-value - это ожидаемое количество случайных находок с таким же и лучшим весом в той же базе данных и при тех же параметрах.
E-value = 1 означает, что в базе данных определённого размера возможна 1 совершенно случайная находка с таким же (или лучшим) весом.
Соответственно, значение E-value больше 1 означает увеличение числа таких случаных находок.
При поиске изучаемого белка в базе данных Swiss-Prot (при параметрах поиска по умолчанию) мы обнаруживаем
одну находку с E-value = 1.8, две находки с E-value = 3.4 и т.д.
Таким образом, данные находки попадают под критерий "случайности", следующий из определения E-value, и гомологами исходного белка, на мой взгляд,
являться не будут.
Альтернативные интерфейсы к программе BLAST
Интерфейс к программе BLAST на сайте EBI:
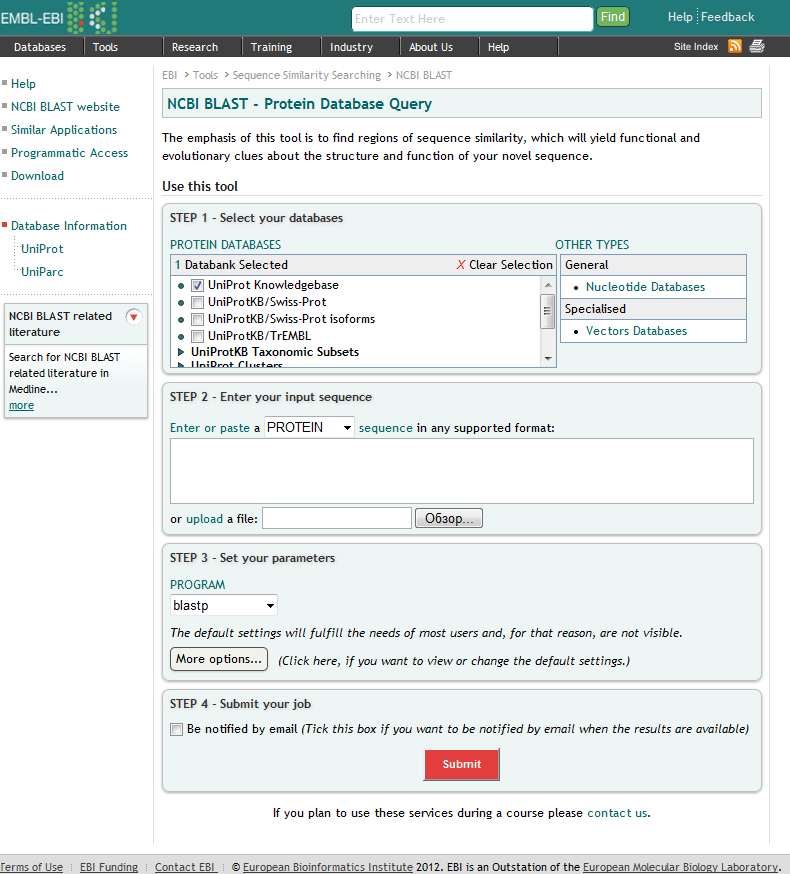
Особенности интерфейса, которые обращают на себя внимание.
- Возможность одновременного выбора нескольких банков данных для поиска. Более удобный и структурированный список банков данных, который "испорчен" небольшим окошком списка доступных для поиска банков.
- Более удобный инструмент поиска с разделением на шаги ввода данных. Более "дружелюбное" представление дополнительных параметров.
- Возможность оповещения по электронной почте о доступности результатов запроса.
- Доступность результатов поиска в течение 7 дней по ссылке.
- Как правило, более долгий процесс поиска.
- Удобное представление результатов, с указанием областей поиска, нумерацией результатов, с начилием возможности управления находками, например, для загрузки всех результатов поиска или их части в виде файла.
- Отсутствие наглядного представления "покрытия" последовательностей при построении выравниваний для результатов поиска.
- "Спрятанные" выравнивания, которые при необходимости можно отобразить только для нужных находок или для всех сразу.
- "Нежелание" сервиса находить какие-либо результаты по указанному AC (при этом поиск по непосредственно введённой последовательности работает).
- Визуально более приятный и более удобный для чтения раздел помощи (Help).
Интерфейс к программе BLAST на сайте Expasy:
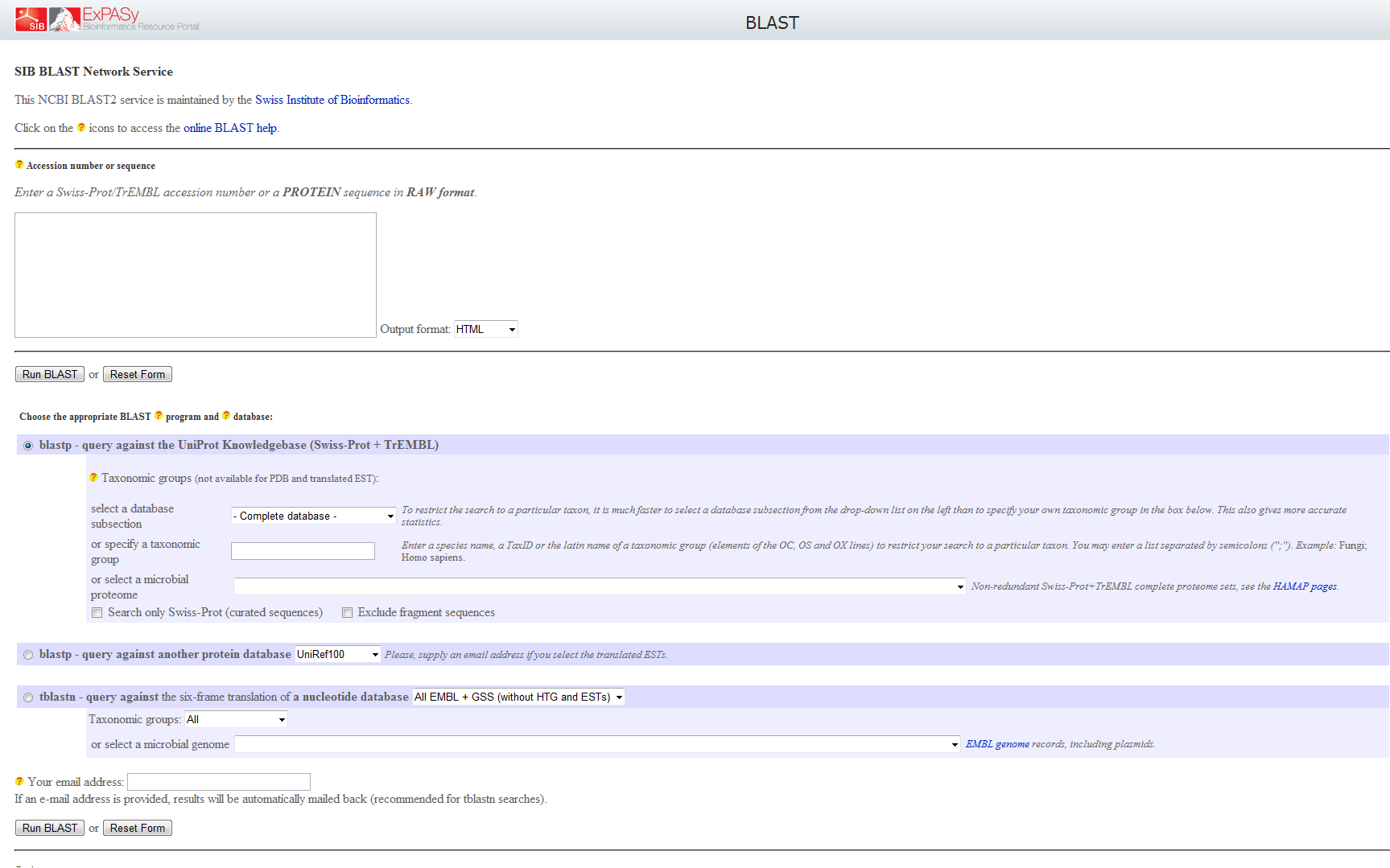
Особенности интерфейса, которые обращают на себя внимание.
- Неразумное распределение свободного места на экране, что, впрочем, не мешает использованию инструментов поиска.
- Пояснения к отдельным пунктам формы поиска вынесены на отделбную страницу, что неудобно.
- Возможность выбора представления результатов поиска уже на стадии заполнения формы поиска.
- Возможность оповещения по электронной почте о доступности результатов запроса.
- Субъективно более долгий процесс поиска.
- Возможность отправления выбранных последовательностей по электронной почте (с дополнительными опциями).
- Возможность представления результатов с группировкой по таксономическим признакам.
- Небольшой (но вполне достаточный) раздел помощи (Help) с неработающими ссылками на разделы помощи по BLAST на сайте NCBI.
Ссылки
- Файл cdd_bacsu-cdd_human.needle - выравнивание, полученное программой needle.
- Файл cdd_bacsu-cdd_human.water - выравнивание, полученное программой water.
