Занятие 2: Банк UniProt. Белок в UniProt.
Извлечь из банка UniProt документ, содержащий информацию о белке CDD_BASCU
Прорамма entret на сервере kodomo умеет извлекать записи из локальных банков данных. Чтобы получить документ для белка CDD_BACSU, запишем команду:
entret sw:cdd_bacsu
Нам будет предложено ввести имя файла. Можно указать имя файла сразу (по умолчанию формат файла - .entret):
entret sw:cdd_bacsu cdd_bacsu
Таким образом, в файле cdd_bacsu.entret записан документ для белка CDD_BACSU.
Заполнение таблицы по данным белка
| Искомая информация | Метка поля | Содержание |
| Код(ы) доступа (Accession number) | AC | P19079 |
| Идентификатор записи в БД | ID | CDD_BACSU |
| Название (краткое описание) белка | DE | Полное название - Cytidine deaminase Короткое название - CDA Номер EC (Enzyme Commission) - 3.5.4.5 Альтернативное название (полное) - Cytidine aminohydrolase |
| Дата создания документа | DT | 01 ноября 1990 |
| Дата последнего исправления аннотации | DT | 25 января 2012 (версия записи - 104) |
| Число публикаций, использованных при создании документа | RN | 6 (поле с меткой RN содержит номер ссылки на публикацию; всего ссылок на публикации - шесть) |
| Журнал и год самой поздней публикации | RL | Журнал Biochemistry, год 2004 |
| Ключевые слова | KW | 3D-structure; Complete proteome; Hydrolase; Metal-binding; Reference proteome; Zinc (3D-структура, полный протеом, гидролаза, металл-связывающий, референсный протеом, цинк) |
| Что содержит поле комментариев? | CC | Поле комментариев содержит следующую информацию: функции, каталитическая активность, кофактор, субъединицы, сходство (принадлежность к семейству), а также указания на авторское право и тип лицензии. |
| Идентификаторы записей PDB | DR | 1JTK 1UWZ 1UX0 1UX1 |
Ответы на вопросы о белке CDD_BACSU
Основываясь на записи UniProt, содержащей информацию о белке CDD_BACSU, а также на публикациях о белке в базе данных PubMed, можно ответить на следующие вопросы.
| Вопрос | Ответ |
| Какие аминокислотные остатки Вы бы стали модифицировать, чтобы изменить характер связывания металла с белком? | 56-й аминокислотный остаток R (аргинин Arg); замена его на D (аспарагиновую кислоту Asp) уменьшает связывание цинка на 80%. Также модификации можно подвергнуть остатки, связанные с металлом (см. ниже). |
| Какие аминокислотные остатки участвуют в образовании активного центра? | Это 55-й аминокислотный остаток E (глутаминовая кислота Glu). |
| Какие ионы связываются с белком? | Это ионы цинка, связанные с 53, 86 и 89 аминокислотными остатками (C - цистеином Cys). |
| Какие мутации белка исследованы? Какие аминокислотные остатки мутировали и к чему это приводило? | 1. Мутация C->H (53-й остаток цистеин Cys мутирует в гистидин His) приводит к
потере активности (уменьшает активность в 500 раз, не оказывая влияния на связывание цинка). 2. Мутация R->A (56-й остаток аргинин Arg мутирует в аланин Ala) значительно уменьшает максимальную скорость фермента (Vmax), не воздействуя на связывание цинка. 3. Мутация R->D (56-й остаток аргинин Arg мутирует в аспарагиновую кислоту Asp) приводит к потере активности и уменьшению связывания цинка на 80%. 4. Мутация R->Q (56-й остаток аргинин Arg мутирует в глутамин Gln) значительно уменьшает максимальную скорость фермента, не оказывает влияния на связывание цинка и приводит к уменьшению активности в 500 раз. |
| Какие участки белка участвуют в связывании лиганда? Какого? | В связывании цинка (Zinc) участвуют аминокислотные остатки №№ 53, 86 и 89. |
| Мутация по какому аминокислотному остатку нарушит связывание белка с каким-либо субстратом? | По остаткам 42-44, которые отвечают за связывание с субстратом (это N - аспарагин Asn, I - изолейцин Ile и E - глутаминовая кислота Glu). |
| Получите последовательность 2-й альфа-спирали, используя команду seqret пакета EMBOSS. | YSM Результат получен с помощью следующей команды: seqret sw:cdd_bacsu second_helix.fasta -sbegin 48 -send 50 См. файл. |
| Получите последовательность 3-го бета-тяжа, используя команду seqret пакета EMBOSS. | FQMLAVAAD Результат получен с помощью следующей команды: seqret sw:cdd_bacsu third_strand.fasta -sbegin 70 -send 78 См. файл. |
| Последовательности большинства белков начинаются с метионина. Почему? После биосинтеза в процессе созревания белка метионин может быть удален. Указан ли метионин в начальной позиции заданного белка? А удаляется ли он потом? | Синтез белка в большинстве случаев начинается с AUG-кодона, кодирующего метионин.
(Этот кодон обычно называют стартовым.) В начальной позиции заданного белка метионин указан. Информация последующем удалении метионина отсутствует. |
| Предложите мутацию, которая, на Ваш взгляд, сильно повлияет на активность белка. Ответ требует краткого обоснования. | На активность белка сильно повлияет замена 53-го остатка цистеина Cys на гистидин His; замена 56-го остатка аргинина Arg на аланин Ala, аспарагиновую кислоту Asp или глутамин Gln (см. выше вопрос об исследованных мутациях белка). |
Поиск белков с кодом CDD или со сходным описанием
Для поиска в SwissProt можно воспользоваться командой infoseq. Выполним следующую команду:
infoseq sw:cdd_* -only -description -noheading | wc --line
Мы сделали запрос на выдачу только описаний белков (-noheading убирает заголовок колонки), а затем с помощью конвейера перенаправили команде wc данные для подсчёта количества строк. В результате получим число 121. Итак, количество записей в SwissProt с кодом белка CDD равно 121.
На сайте UniProt с помощью поискового запроса мы можем узнать число записей со сходным описанием в SwissProt и TrEMBLE:
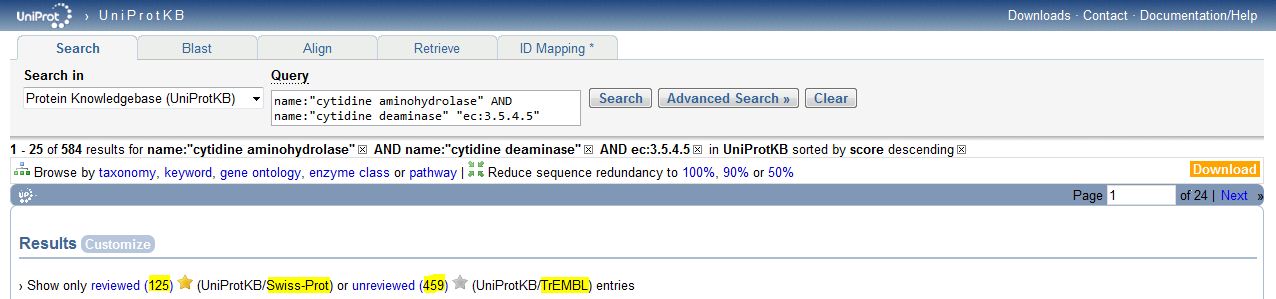
Заполним таблицу полученными результатами.
| Команда/Запрос | Число записей в SwissProt | Число записей в TrEMBL |
| infoseq sw:cdd_* -only -description -noheading | wc --line | 121 | - |
| name:"cytidine aminohydrolase" AND name:"cytidine deaminase" "ec:3.5.4.5" | 125 | 459 |
| name:"cytidine aminohydrolase" AND name:"cytidine deaminase" | 125 | 474 |
| "Cytidine aminohydrolase" "Cytidine deaminase" "3.5.4.5" | 125 | 460 |
| "Cytidine deaminase" | 759 | 17828 |
| "Cytidine aminohydrolase" | 125 | 476 |
Сравнение записи белка CDD_BACSU с записью белка с похожим описанием
Для выполнения задания подойдёт, например, белок CDD_HUMAN. На странице этого белка в UniProt предоставлена обширная информация о свойствах, функциях, строении, биологической роли белка; в наглядном виде приведена вторичная структура с выделением спиралей, тяжей и реверсивных поворотов; приведены последовательность белка и ссылки на статьи и другие базы данных, содержащие информацию о данном белке.
Получить файл с информацией о белке можно, выполнив следующую команду:
entret sw:cdd_human cdd_human
Файл cdd_human.entret содержит необходимую нам информацию.
Теперь произведём сравнение записей белков CDD_BACSU и CDD_HUMAN.
| Информация\Источник | Метка поля | Белок 1 | Белок 2 |
| Первый код доступа | AC | P19079 | P32320 |
| Идентификатор последовательности в БД | ID | CDD_BACSU | CDD_HUMAN |
| Название (краткое описание) белка | DE | Полное название - Cytidine deaminase Короткое название - CDA Номер EC (Enzyme Commission) - 3.5.4.5 Альтернативное название (полное) - Cytidine aminohydrolase |
Полное название - Cytidine deaminase Номер EC (Enzyme Commission) - 3.5.4.5 Альтернативное название (полное) - Cytidine aminohydrolase |
| Дата создания документа | DT | 01 ноября 1990 | 01 октября 1993 |
| Дата последнего исправления аннотации | DT | 25 января 2012 (версия записи - 104) | 25 января 2012 (версия записи - 111) |
| Название организма | OS | Bacillus subtilis | Homo sapiens (Human) |
| Классификация организма (список таксонов) | OC | Bacteria; Firmicutes; Bacillales; Bacillaceae; Bacillus | Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini; Catarrhini; Hominidae; Homo |
| Длина последовательности | SQ | 136 AA | 146 AA |
| Молекулярная масса белка | SQ | 14854 MW | 16185 MW |
| Число публикаций, использованных при создании документа | RN | 6 (шесть) | 7 (семь) |
| Журнал и год самой поздней публикации | RL | Журнал Biochemistry, год 2004 | Журнал Nature, год 2006 |
| Описание вторичной структуры | FT | 6 альфа-спиралей, 7 бета-тяжей, 1 реверсивный поворот:HELIX 3 14 TURN 20 22 STRAND 26 32 STRAND 37 41 HELIX 48 50 HELIX 54 64 STRAND 70 78 STRAND 80 82 HELIX 87 96 STRAND 102 106 STRAND 108 110 STRAND 112 116 HELIX 117 120 HELIX 127 130 |
7 альфа-спиралей, 5 бета-тяжей, 1 реверсивный поворот:HELIX 15 25 HELIX 26 28 TURN 32 34 STRAND 38 43 STRAND 49 53 HELIX 60 62 HELIX 66 76 STRAND 83 90 HELIX 100 107 STRAND 111 118 STRAND 124 128 HELIX 129 132 HELIX 139 141 |
| Ключевые слова | KW | 3D-structure; Complete proteome; Hydrolase; Metal-binding; Reference proteome; Zinc (3D-структура, полный протеом, гидролаза, металл-связывающий, референсный протеом, цинк) | 3D-structure; Complete proteome; Hydrolase; Metal-binding; Polymorphism; Reference proteome; Zinc (3D-структура, полный протеом, гидролаза, металл-связывающий, полиморфизм, референсный протеом, цинк) |
| Темы, освещённые в комментариях | CC | Функции, каталитическая активность, кофактор, субъединицы, сходство (принадлежность к семейству), а также указания на авторское право и тип лицензии. | Функции, каталитическая активность, кофактор, субъединицы, специфика ткани, сходство (принадлежность к семейству), а также указания на авторское право и тип лицензии. |
| Особенности последовательности | FT | За связывание с металлом отвечают 3 аминокислотных остатка; исследовано 4 мутации; 14 элементов вторичной структуры. | За связывание с металлом отвечают 3 аминокислотных остатка; имеется вариативность в одном аминокислотном остатке; 13 элементов вторичной структуры. |
| Идентификаторы записей PDB | DR | 1JTK 1UWZ 1UX0 1UX1 |
1MQ0 |
Мы видим, что документ с информацией о белке CDD_HUMAN был создан несколько позже, однако аннотация исправлялась немного чаще. Даты последнего исправления аннотации совпадают. Длина последовательности в белке CDD_HUMAN немного длиннее (на 10 оснований). Число публикаций, используемых для составления файла о белке CDD_HUMAN, незначительно больше. При этом информация более актуальна, впрочем, назвать существенной разницу в датах последних публикаций нельзя. Среди ключевых слов в записи о CDD_HUMAN замечаем полиморфизм. Белок CDD_BACSU имеет 4 идентификатора записей PDB, в то время как CDD_HUMAN - всего одну.
Подводя итог сравнения, можно сказать, что, несмотря на множество отличий, значительной разницы по рассмотренным критериям белков CDD_BACSU и CDD_HUMAN обнаружить не удаётся. Таким образом, сходное описание белков говорит о сходных значениях рассмотренных критериев в описаниях белков.
Ссылки
- Документ из банка UniProt для белка CDD_BACSU.
- Документ из банка UniProt для белка CDD_HUMAN.
- Результат поиска белков с описанием, сходным с описанием рассматриваевого белка CDD_BACSU, на сайте UniProt.
