Занятие 12: PSI-BLAST
Итеративный поиск программой PSI-BLAST
Для начала работы с программой PSI-BLAST необходимо перейти на домашнюю страницу BLAST, перейдя затем по ссылке protein blast. Поиск аминокислотных последовательностей с помощью программы PSI-BLAST требуется произвести по банку Swiss-Prot, используя значение 5000 для максимального количества отображаемых в результате последовательностей и стандартное значение для порога на E-value, равное 0,005.
Поиск MINC_ECOLI (P18196)
- Число находок от итерации к итерации увеличивается. На второй итерации число находок увеличилось на 25, на третьей - на 14, на четвёртой - на 249, на пятой - на 542 и стало равным 995.
- Разрыв между значениями E-value худшей находки выше порога и лучшей находки ниже порога от итерации к итерации сначала увеличивается (от 0.005 — 0.004 до 0.066 — 0.005), затем, на третьей итерации, уменьшается до 0.006 — 0.004, на четвёртой итерации лучшая находка ниже порога и худшая находка выше порога не имеют отличий в значении E-value с точностью до третьего знака после запятой (0.005), затем на пятой итерации разрыв снова возрастает и равен 0.023 — 7e-04.
- E-value лучшей находки (белка MINC_ECOLI) увеличивает от 8e-171на первой итерации (значения на 2, 3 и 4 итерациях соответственно равны 1e-117, 1e-100, 6e-97) до 1e-83 на пятой итерации.
- Если проследить за E-value "средней" находки, например, белка MINC_SYNY3 (AC Q55901), то мы увидим, что значение E-value уменьшается от 7e-04 на первой итерации до 1e-29 на пятой итерации (при этом значения на 2, 3 и 4 итерациях соответственно равны 7e-28, 6e-34, 5e-35).
- В данном случае можно наблюдать появление всё более разных новых последовательностей от итерации к итерации. После выполнения пяти итераций список не "стабилизировался", однако для последующих итераций появление новых находок не наблюдается.
- В данном случае можно говорить о расходимости результатов поиска программой PSI-BLAST.
Поиск SSRP_ECOLI (P0A832)
- Число находок от итерации к итерации не изменяется.
- Разрыв между значениями E-value лучшей находки ниже порога и худшей находки выше порога от итерации к итерации изменяется от 4.9 — 3e-12 до 0.42 — 3e-38.
- E-value лучшей находки (белка SSRP_ECOLI) равно 3e-116 на первой итерации. Затем белок перемещается на вторую позицию в выдаче с E-value, равным 2e-94; первую же позицию занимает белок SSRP_YERE8 (AC A1JKI0.1) со значением 1e-94.
- Если проследить за E-value "средней" находки, например, белка SSRP_MYCS5 (AC Q4A5T2), то мы увидим, что значение E-value уменьшается от 3e-12 на первой итерации до 3e-38 на второй итерации.
- В данном случае можно наблюдать отсутствие появления новых последовательностей от итерации к итерации. После выполнения двух итераций список "стабилизировался". Уже на второй итерации не было найдено новых последовательностей выше заданного порога.
- В данном случае можно говорить о сходимости результатов поиска программой PSI-BLAST.
- Произведем поиск последовательности белка SSRP_ECOLI (AC P0A832) в BLAST. Заметим, что результаты выдачи совпадают с полученными ранее с помощью программы PSI-BLAST.
Поиск Y380_RHIME (P17265)
- Число находок от итерации к итерации увеличивается. На второй итерации число находок увеличилось на 9, на третьей - на 1. На четвёртой и последующих итерациях число находок не изменяется.
- Разрыв между значениями E-value лучшей находки ниже порога и худшей находки выше порога от итерации к итерации увеличивается от 0.027 — 7e-04 до 0.024 — 3e-18 на четвёртой итерации (на второй итерации разрыв был следующим: 0.011 — 5e-09; на третьей - 0.012 — 0.002).
- E-value лучшей находки (белка Y380_RHIME) увеличивает от 7e-141 на первой итерации (значения на 2 и 3 итерациях соответственно равны 1e-77 и 2e-75) до 1e-71 на четвертой итерации.
- Если проследить за E-value "средней" находки, например, белка HPF_PSEPK (AC P0A147), то мы увидим, что значение E-value уменьшается от 7e-04 на первой итерации до 8e-30 на четвёртой итерации (при этом значения на 2 и 3 итерациях соответственно равны 2e-24 и 2e-29).
- После выполнения четырёх итераций список "стабилизировался". На четвёртой итерации не было найдено новых последовательностей выше заданного порога.
- В данном случае можно говорить о сходимости результатов поиска программой PSI-BLAST.
Поиск CDD_BACSU (P19079)
- Число находок от итерации к итерации не изменяется.
- Разрыв между значениями E-value лучшей находки ниже порога и худшей находки выше порога от итерации к итерации изменяется от 1.8 — 5e-04 до 0.23 — 2e-22.
- E-value лучшей находки (белка CDD_BACSU) увеличивается от 3e-97 на первой итерации до 3e-61 на четвертой итерации.
- Если проследить за E-value "средней" находки, например, белка CDD_ECO57 (AC Q8X648), то мы увидим, что значение E-value уменьшается от 4e-04 на первой итерации до 1e-43 на второй итерации.
- В данном случае можно наблюдать отсутствие появления новых последовательностей от итерации к итерации. После выполнения двух итераций список "стабилизировался". Уже на второй итерации не было найдено новых последовательностей выше заданного порога.
- В данном случае можно говорить о сходимости результатов поиска программой PSI-BLAST.
- Если произвести поиск последовательности белка CDD_BACSU (AC P19079) в BLAST и сравнить результаты поиска результатами, с полученными ранее с помощью программы PSI-BLAST, то можно сделать вывод о совпадении результатов.
Заполним следующую таблицу результатами произведённого поиска:
| ID белка | AC белка | Число итераций | Для первой итерации | Для последней итерации | ||||
| Число находок выше порога (0,005) | Худшее E-value выше порога | Лучшее E-value ниже порога | Число находок выше порога (0,005) | Худшее E-value выше порога | Лучшее E-value ниже порога | |||
| MINC_ECOLI | P18196 | 5 | 165 | 0.004 | 0.005 | 995 | 7e-04 | 0.023 |
| SSRP_ECOLI | P0A832 | 2 | 514 | 3e-12 | 4.9 | 514 | 3e-38 | 0.42 |
| Y380_RHIME | P17265 | 4 | 15 | 7e-04 | 0.027 | 25 | 3e-18 | 0.024 |
| CDD_BACSU | P19079 | 2 | 85 | 5e-04 | 1.8 | 85 | 2e-22 | 0.23 |
Повторный поиск с изменённым значением порога на E-value
Проведём повторный поиск для последовательности белка MINC_ECOLI (AC P18196), изменив порог на E-value с 0.005 на 0.001.
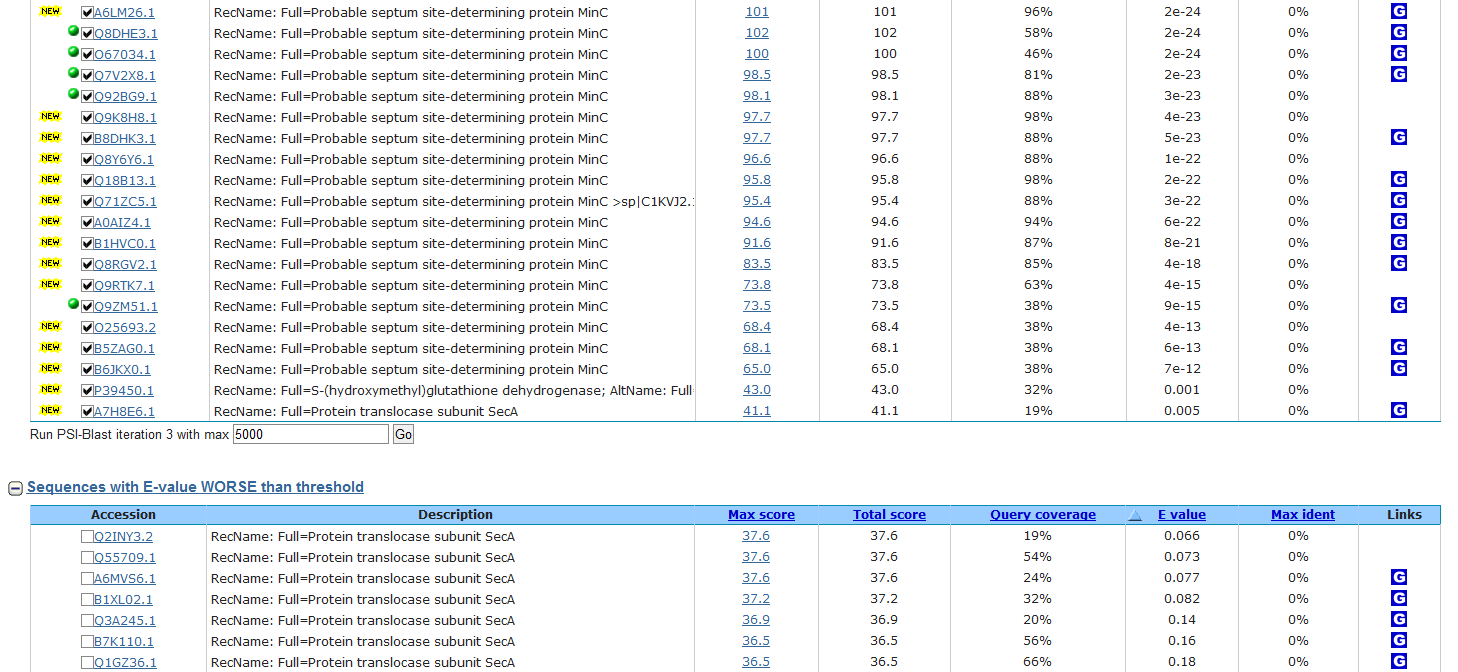
На первой итерации мы видим 157 находок выше порога, худшая из которых имеет значение E-value, равное 0.001. Лучшая находка ниже порога имеет значение 0.001. Все находки выше порога содержат в описании характеристику, как сайт-определяющего белка MinC.
На второй итерации мы видим 188 находок выше порога, худшая из которых имеет значение E-value, равное 8e-08. Лучшая находка ниже порога имеет значение 0.003. Разрыв между указанными значениями заметно увеличился. Все находки выше порога по-прежнему соответствуют описанию septum site-determining protein MinC.
На третьей итерации число находок выше порога не изменилось. Новых последовательностей выше заданного порога найдено не было. Худшая находка ниже порога имеет значение E-value, равное 4e-12, лучшая находка ниже порога - 0.001.
Таким образом, список "стабилизировался" ещё на второй итерации. Подобное отличие от результатов поиска с порогом E-value, равным 0.005, можно объяснить, сравнивая положение находок выше и ниже установленного порога. Заметим, что на второй итерации ниже порога в данном случае оказались последовательности белков FRMA_PASPI (AC P39450) и SECA_ANADF (AC A7H8E6), которые оказались выше порога, равного 0.005, при первом поиске. Описание этих белков не соответствует описанию большинства находок выше порога, приведённому выше. Если при первом поиске (порог на E-value равен 0.005) на второй итерации исключить две указанные последовательности из списка находок, по выравниванию которых составляется PSSM (позиционно специфическая матрица весов), убрав галочки из соответствующих чекбоксов, то на следующей итерации мы получим результат, аналогичный тому, который мы получили при повторном поиске (порог на E-value равен 0.001). Таким образом, при повторном поиске мы сделали фильтрацию результатов более "строгой", уменьшив значение E-value для порога, что привело к сходимости результатов поиска программой PSI-BLAST.
Максимальное значение порога в данном конкретном случае равно 0.0010 с точностью до четвёртого знака (уже при пороге 0.0011 выше порога оказывается последовательность белка FRMA_PASPI (AC P39450), что затем приводит к расходимости результатов).
< На страницу семестра ∧ Наверх