Занятие 3: Поисковые системы
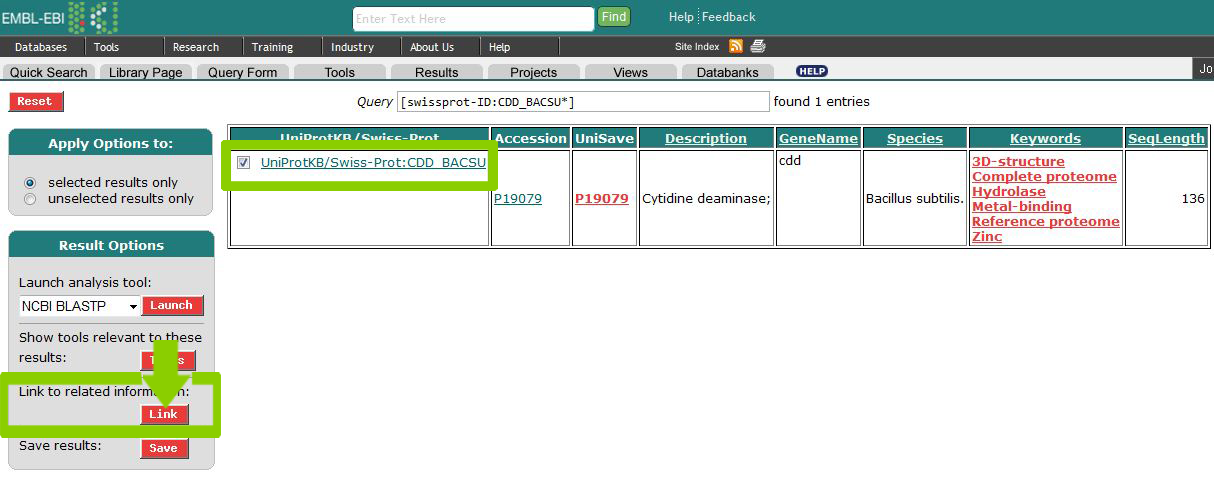
Найти белок CDD_BACSU в банке SwissProt с помощью SRS и описать не менее трех особенностей в его аминокислотной последовательности
Поиск осуществляется с помощью SRS - Sequence Retrieval System. Ход выполнения задания кратко представлен в виде скриншотов.
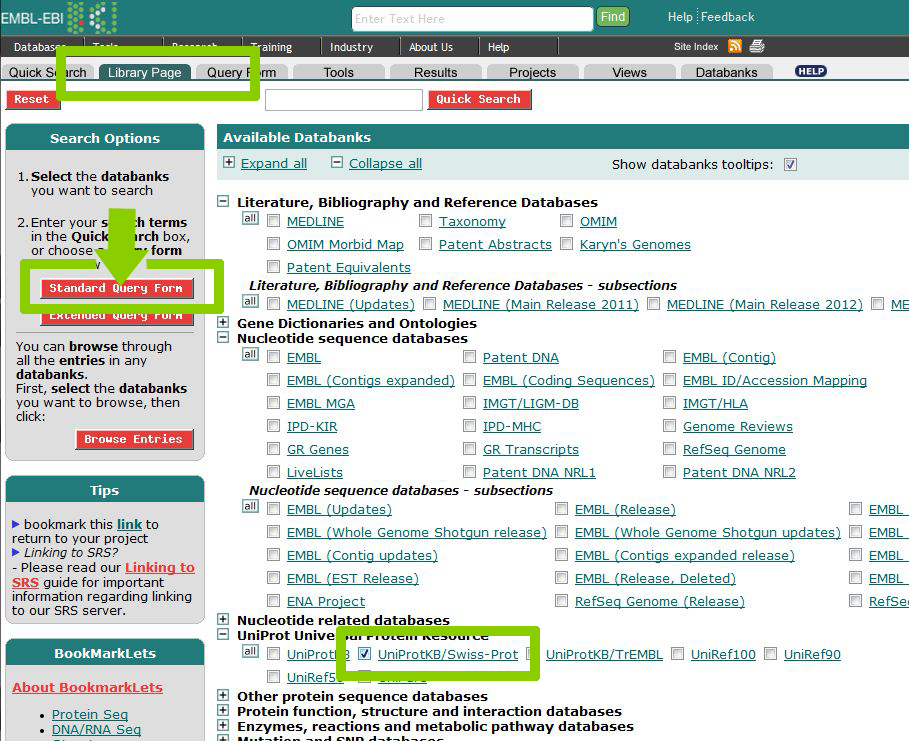
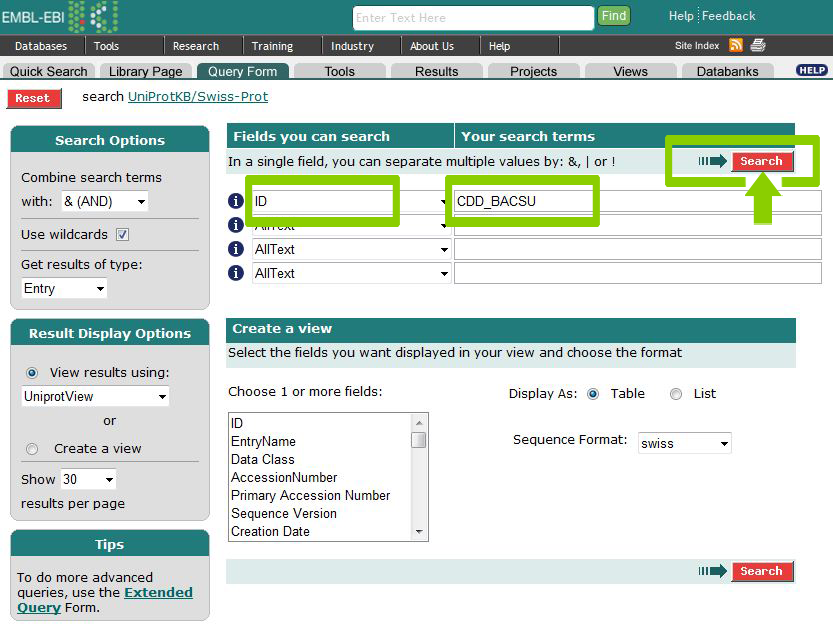
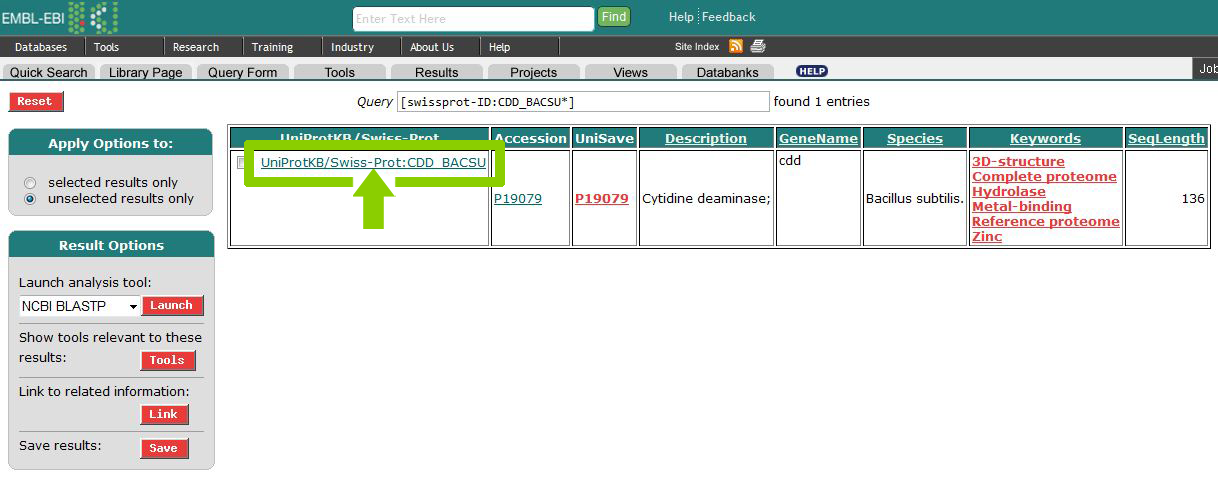
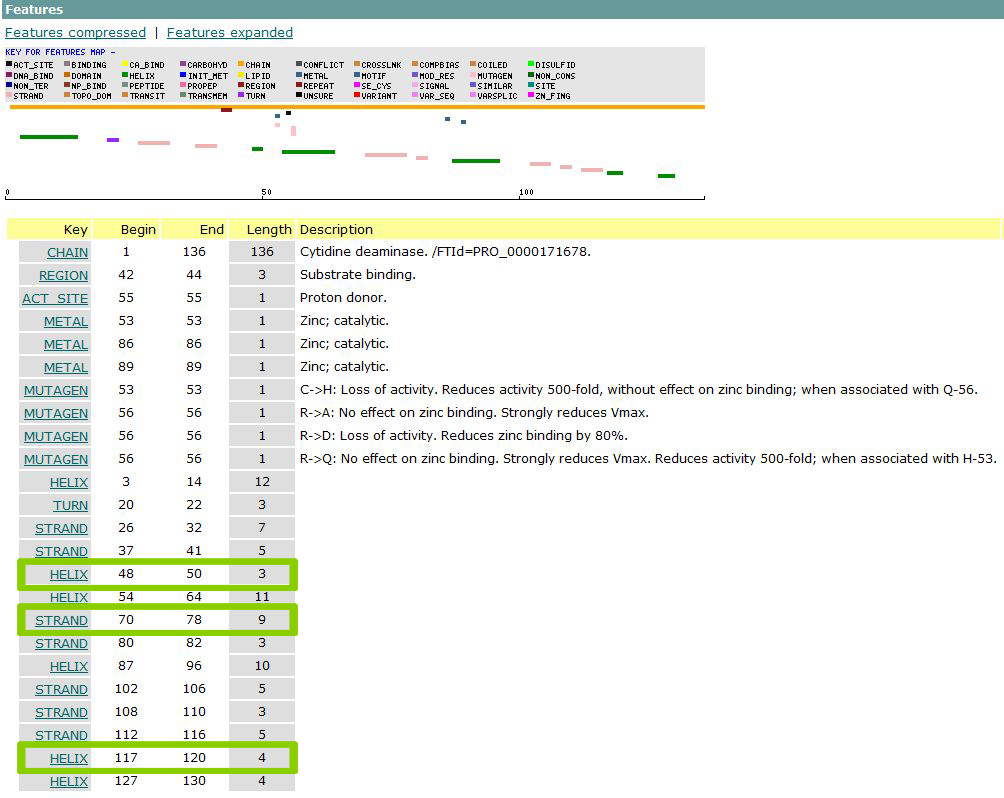
Выберем несколько особенностей, например, вторую альфа-спираль, третий бета-тяж и пятую альфа-спираль.
Вторая альфа-спираль образована тремя аминокислотными остатками (с 48 до 50): тирозином, серином и метионином (YSM):
ID CDD_BACSU_15; parent: CDD_BACSU FT HELIX 48 50 SQ Sequence 3 AA; YSM //
Третий бета-тяж образован девятью аминокислотными остатками (с 70 до 78): фенилаланином, глутамином, метионином, лейцином, аланином, валином, аланиом, аланином и аспарагиновой кислотой (FQMLAVAAD):
ID CDD_BACSU_17; parent: CDD_BACSU FT STRAND 70 78 SQ Sequence 9 AA; FQMLAVAAD //
Пятая альфа-спираль образована четырьмя аминокислотными остатками (с 117 до 120): валином, глутаминовой кислотой, глутаминовой кислотой и лейцином (VEEL):
ID CDD_BACSU_23; parent: CDD_BACSU FT HELIX 117 120 SQ Sequence 4 AA; VEEL //
Получить и сохранить описание всех записей банка трехмерных структур PDB, относящихся к белку CDD_BACSU
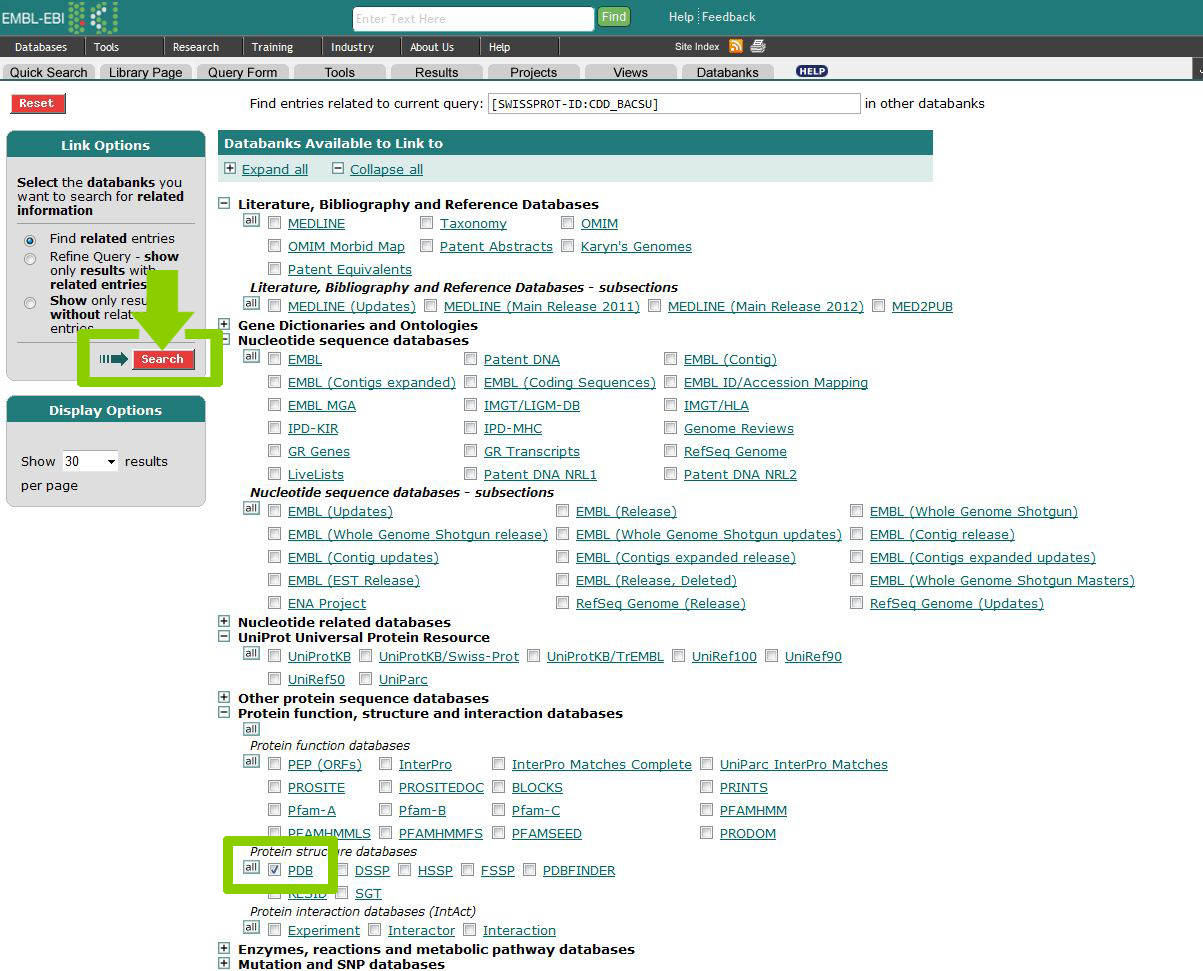
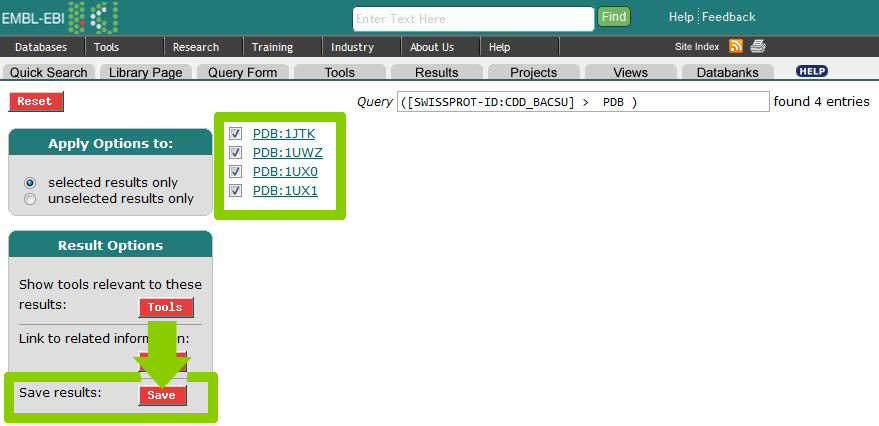
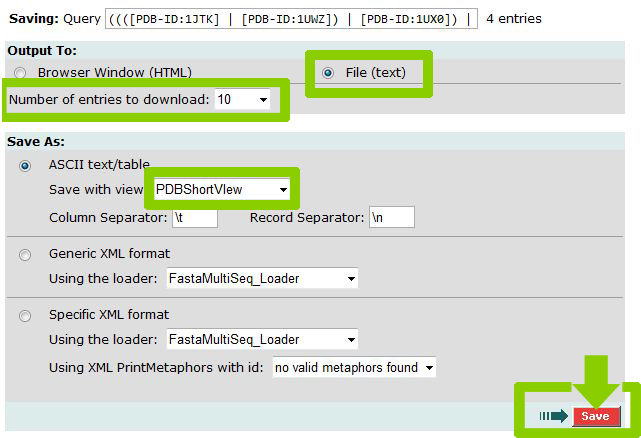
В результате сохранения полученной информации мы получим файл all_pdb_files_cdd_bacsu.txt, содержащий в себе информацию четырёх записей pdb (PDB:1JTK, PDB:1UWZ, PDB:1UX0, PDB:1UX1), представленную в виде PDBShortView.
Найти полноразмерные белки из Firmicutes, выполняющие функцию, сходную с функцией белка CDD_BACSU
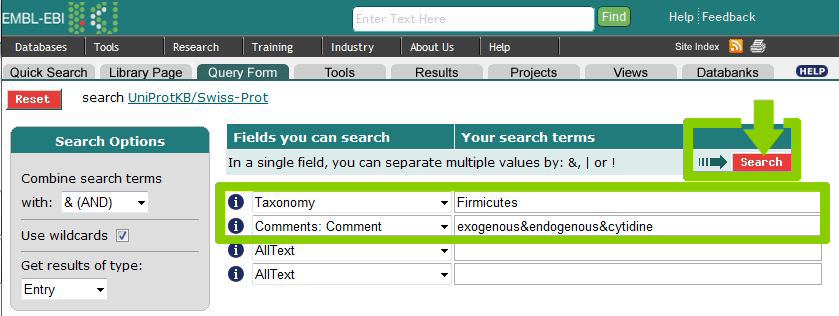
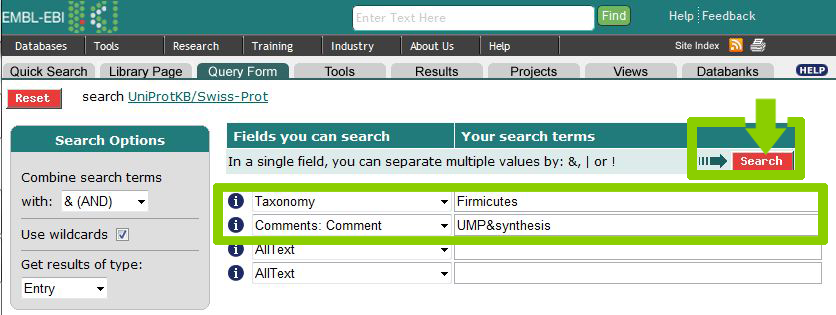
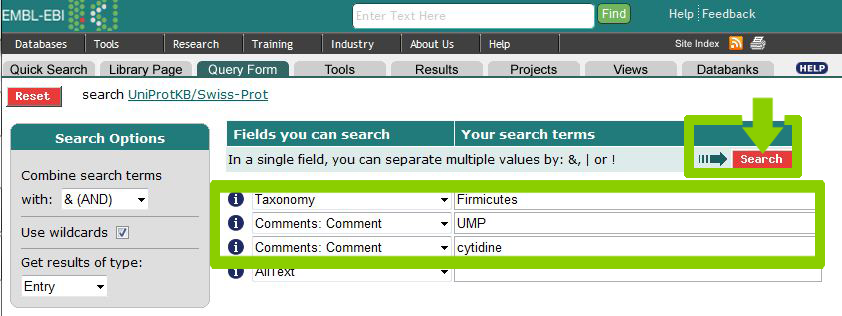
Сначала составим несколько запросов на все последовательности.
Затем составим запрос только на полноразмерные последовательности.
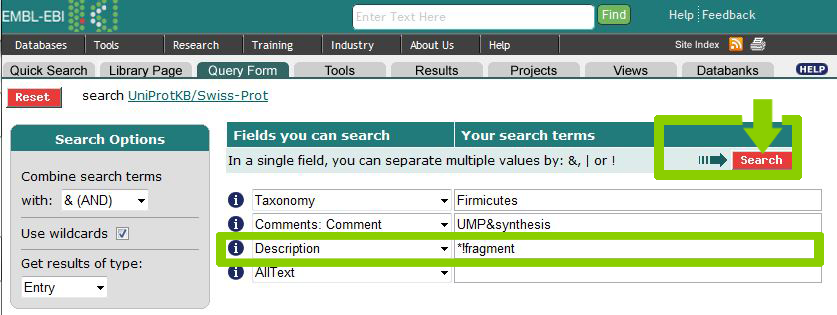
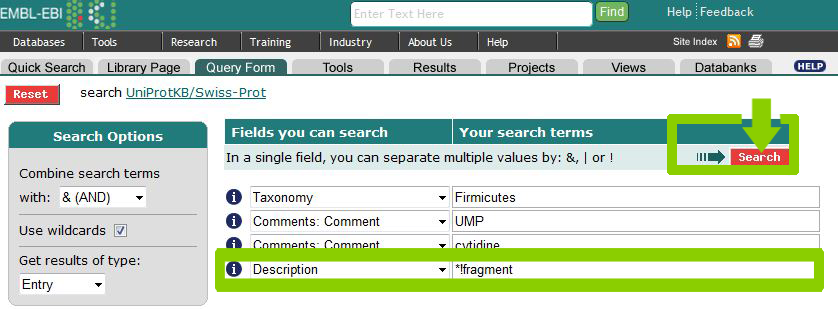
Таблица встеречаемости в SwissProt белков из Firmicutes, имеющих функцию, сходную с функцией белка CDD_BACSU из Bacillus subtilis
| Формулировка функции белка | Строка запроса | Количество найденных документов |
|---|---|---|
| Сборка экзогенного и эндогенного цитидина (exogenous&endogenous&cytidine) | ([swissprot-Taxonomy:Firmicutes*] & ((([swissprot-Comment:exogenous*] & [swissprot-Comment:endogenous*]) & [swissprot-Comment:cytidine*]) > parent )) | 3 |
| Синтез UMP-киназы UMP&synthesis | ([swissprot-Taxonomy:Firmicutes*] & (([swissprot-Comment:UMP*] & [swissprot-Comment:synthesis*]) > parent )) | 3 |
| Упоминание в описании функции белка UMP-киназы и цитидина (UMP; cytidine) | (([swissprot-Taxonomy:Firmicutes*] & ([swissprot-Comment:UMP*] > parent )) & ([swissprot-Comment:cytidine*] > parent )) | 108 |
| Поиск только полноразмерных последовательностей | ||
| Полноразмерная последовательность: синтез UMP-киназы UMP&synthesis | (([swissprot-Taxonomy:Firmicutes*] & (([swissprot-Comment:UMP*] & [swissprot-Comment:synthesis*]) > parent )) & ([swissprot-Description:*] ! [swissprot-Description:fragment*])) | 3 |
| Полноразмерная последовательность: упоминание в описании функции белка UMP-киназы и цитидина (UMP; cytidine) | ((([swissprot-Taxonomy:Firmicutes*] & ([swissprot-Comment:UMP*] > parent )) & ([swissprot-Comment:cytidine*] > parent )) & ([swissprot-Description:*] ! [swissprot-Description:fragment*])) | 108 |
Сохранить полноразмерные последовательности найденных белков в fasta-формате
Выберем первые десять результатов в выдаче по последнему указанному в таблице поисковому запросу.
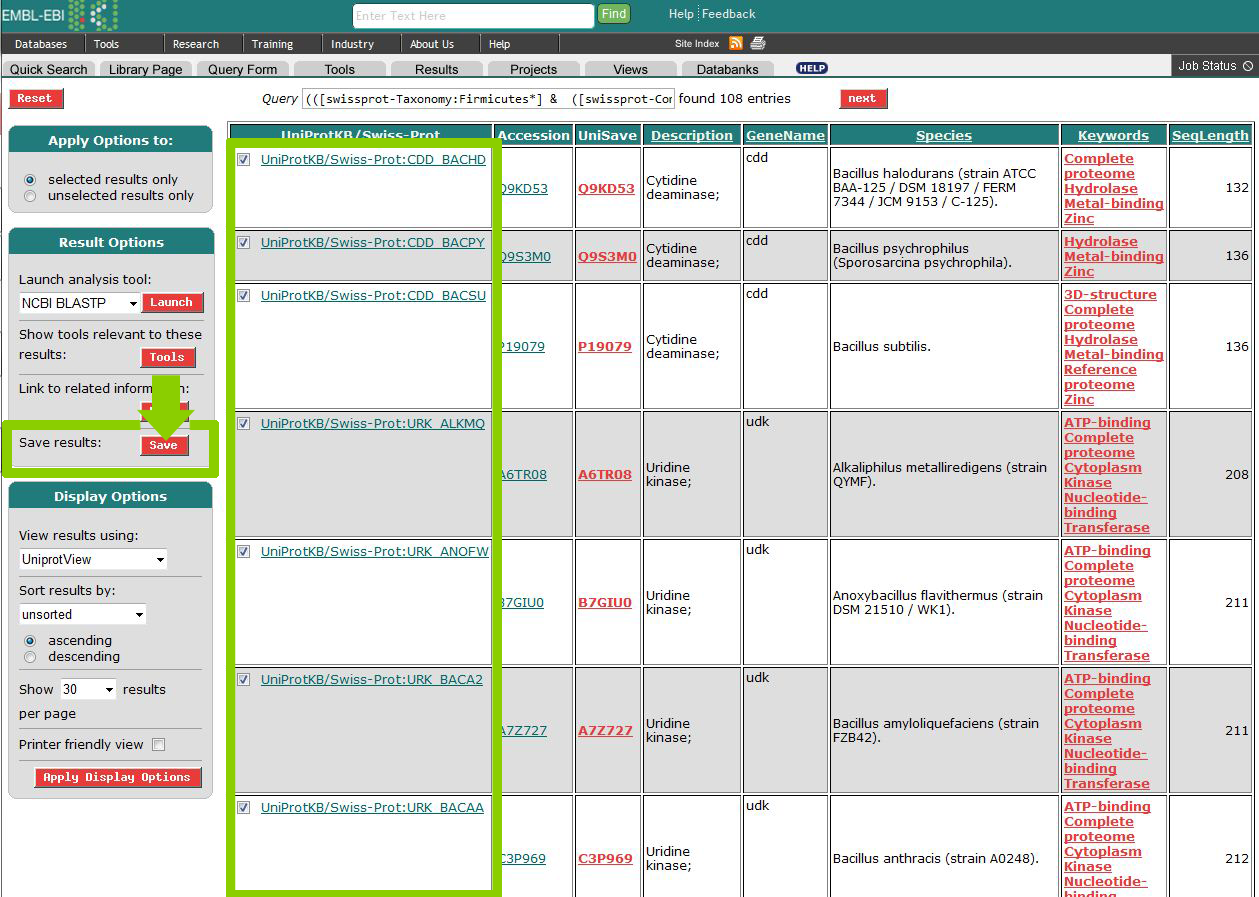
Затем сохраним выбранные полноразмерные последовательности найденных белков в формате .fasta.
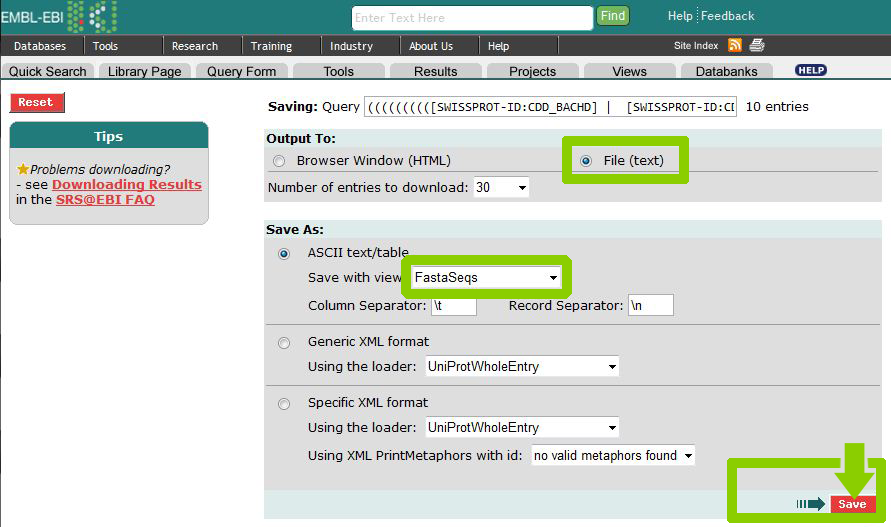
В результате получаем следующий файл с последовательностями: all_fasta_files_similar_cdd_bacsu.fasta.
Сохранить список последовательностей из предыдущего задания, в котором указаны ID, AC, Species, Description и длина последовательности
В разделе Create a view выберем следующие поля: ID, AccessionNumber, Description, Species, Sequence Length.
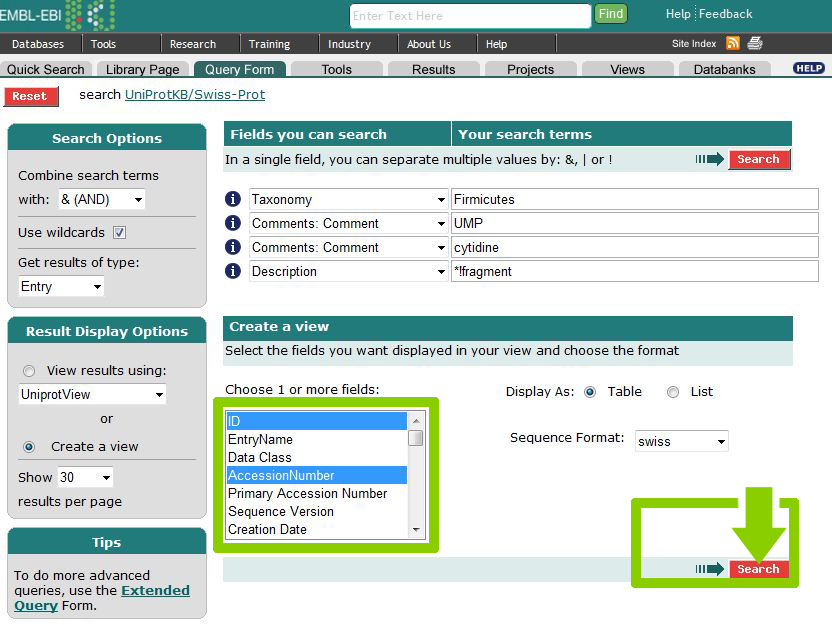
Отметим 10 последовательностей, которые были сохранены в формате .fasta.
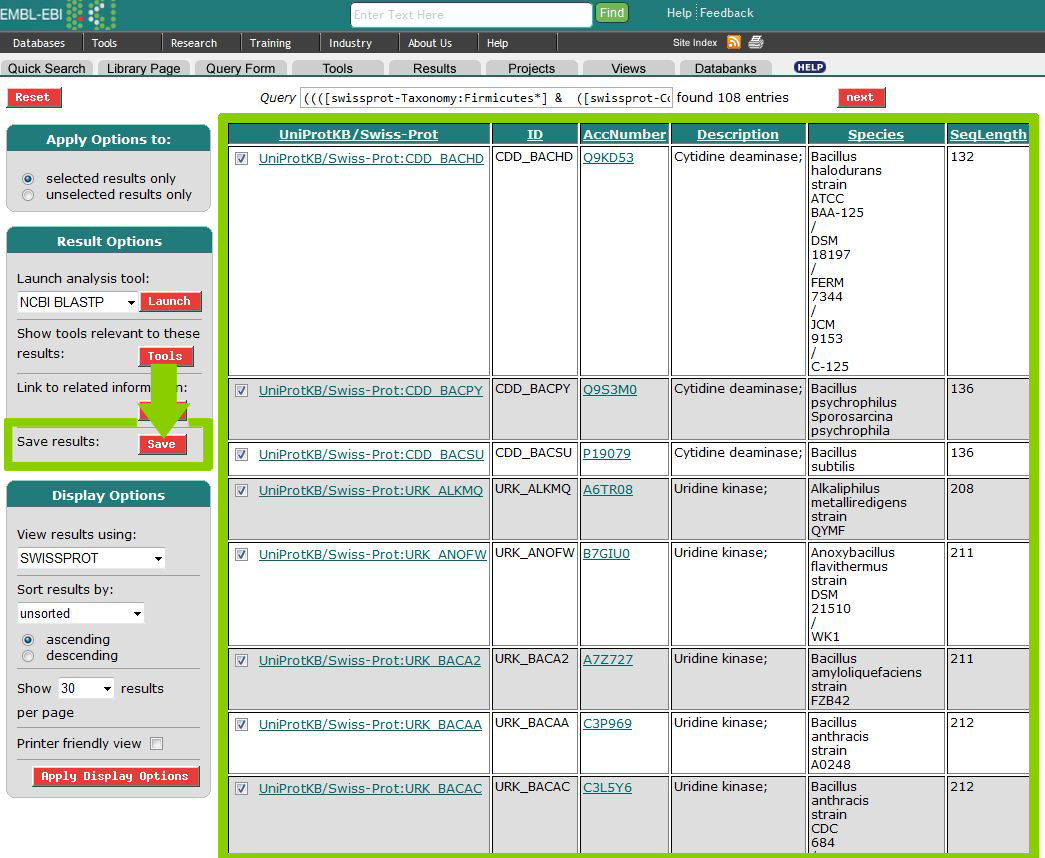
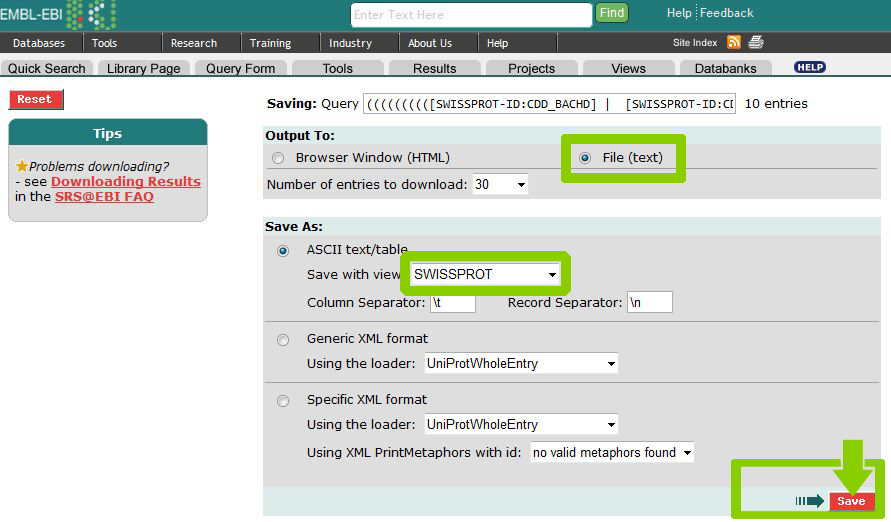
В результате получаем файл ten_similar_cdd_bacsu.xls , представляющий собой плоскую таблицу.
Описать изменения, произошедшие с записью белка CDD_BACSU от первого ее появления в банке Uniprot до последней версии
Воспользуемся сервисом ExPASy.
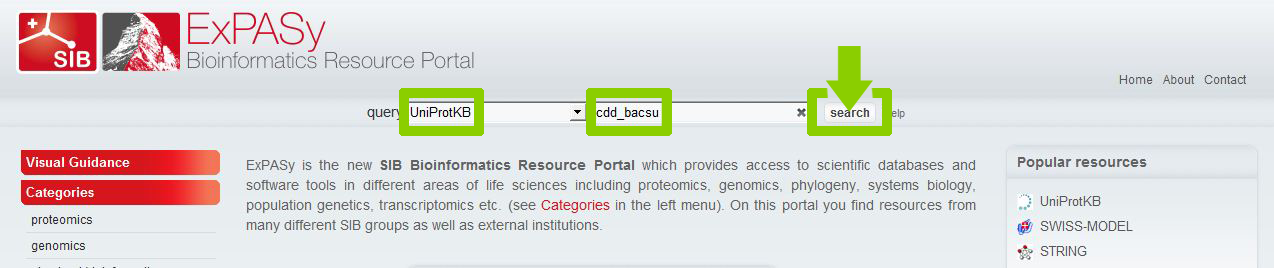
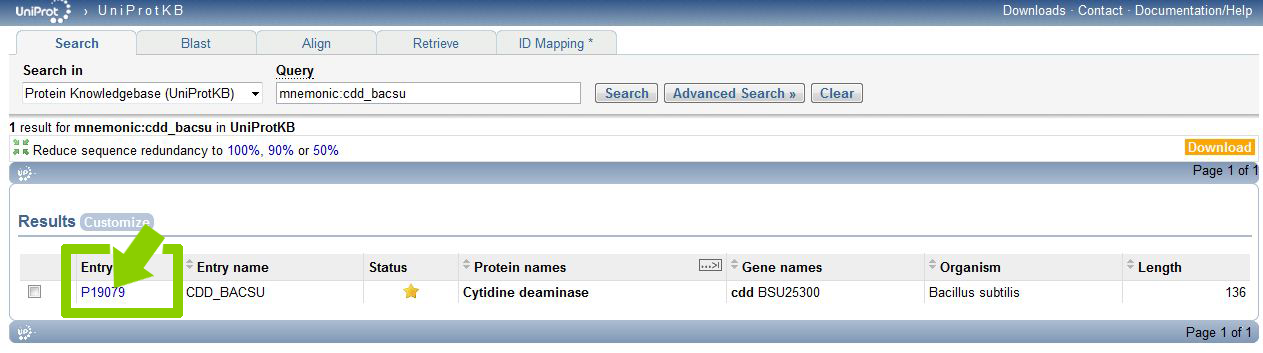
Изучим историю изменения записи белка CDD_BACSU. Для этого перейдём по ссылке History или Complete history.
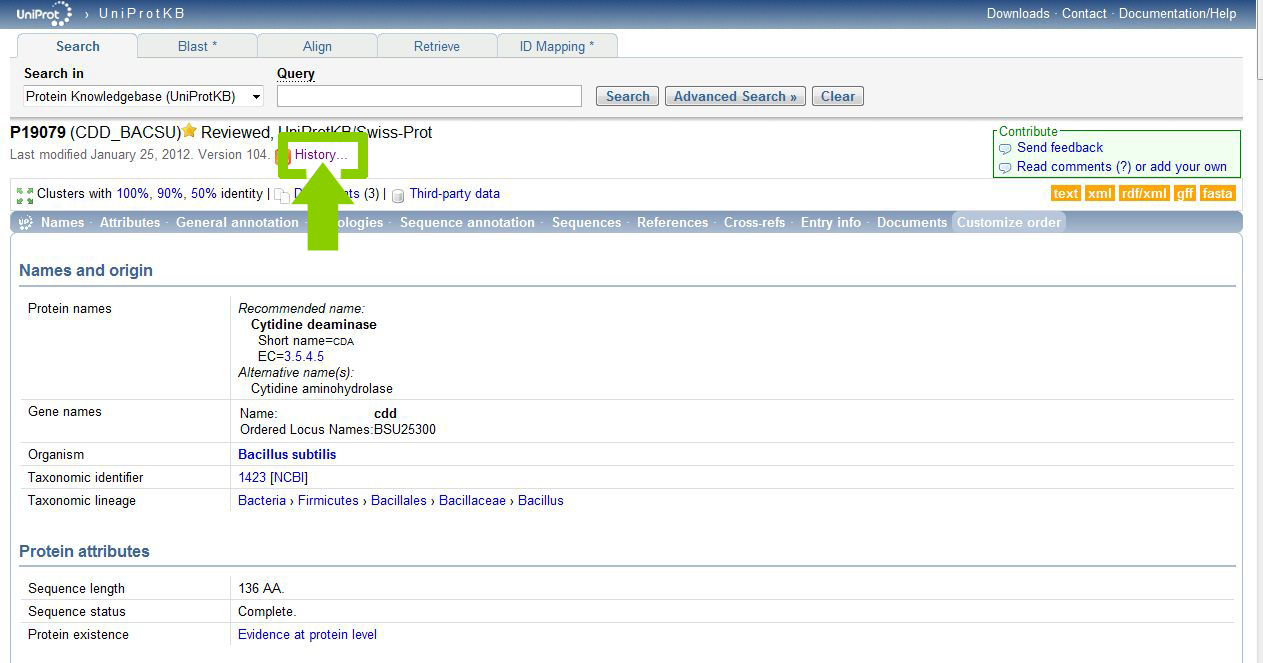
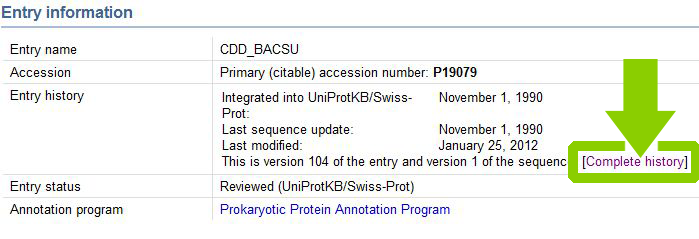
Выберем для сравнения первую и последнюю версии записи.
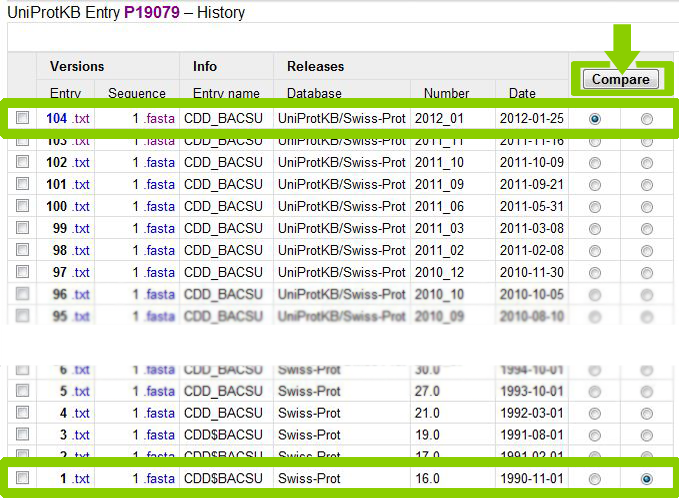
Информация об измененях в записи представлена в наглядной форме.
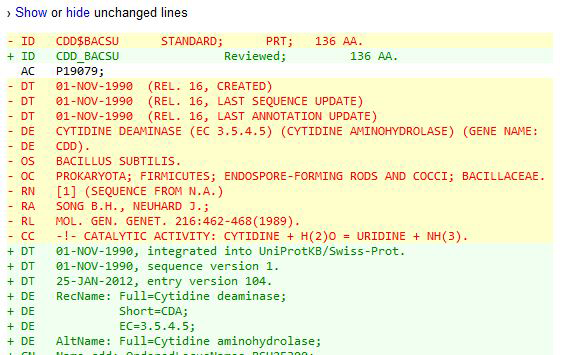
После изучения страницы сравнения версий записей можно сделать следующие выводы:
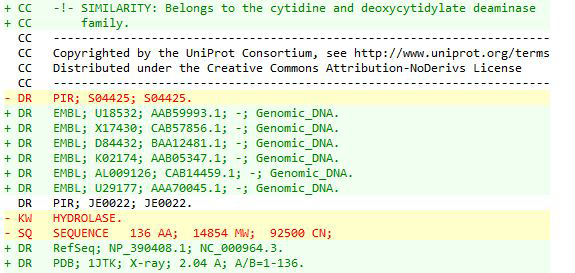
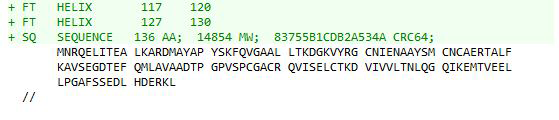
- Первая запись датируется 1 ноября 1990 года (01-NOV-1990), последняя - 25 января 2012 года (25-JAN-2012).
- Идентификатор AC остался неизменным - P19079. Идентификатор ID изменился с CDD$BACSU на CDD_BACSU (по сути, изменилась форма записи идентификатора ID).
- Изменения в последовательности отсутствуют.
- Последняя версия записи несёт заметно больший объём информации. Например, появились описание функции белка, ссылки на различные материалы с указанием авторов и другой информации, изменилась форма записи некоторых полей (так, в новой версии присутствует отдельное указание AltName). Отдельно стоит отметить отсутствие информации об особенностях белка (Features) в первой записи.
Таким образом, новая версия записи, состоящая из 182 строк (против 25 строк первой записи), содержит в себе намного больше информации, что, впрочем, естественно. Мы видим, что единственной информацией, сохранившейся на протяжении более чем 20 лет, стали идентификатор AC, последовательность, информация об авторских правах (Copyright) и лицензии, а также идентификатор записи в базе данных PIR (Protein Information Resource).
Найти и изучить страницу с описанием поля Taxonomy банка Swissprot
В SRS зайдём на страницу Library Page, откуда проследуем на страницу с описанием банка SwissProt.
Затем перейдём на страницу описания поля Taxonomy.
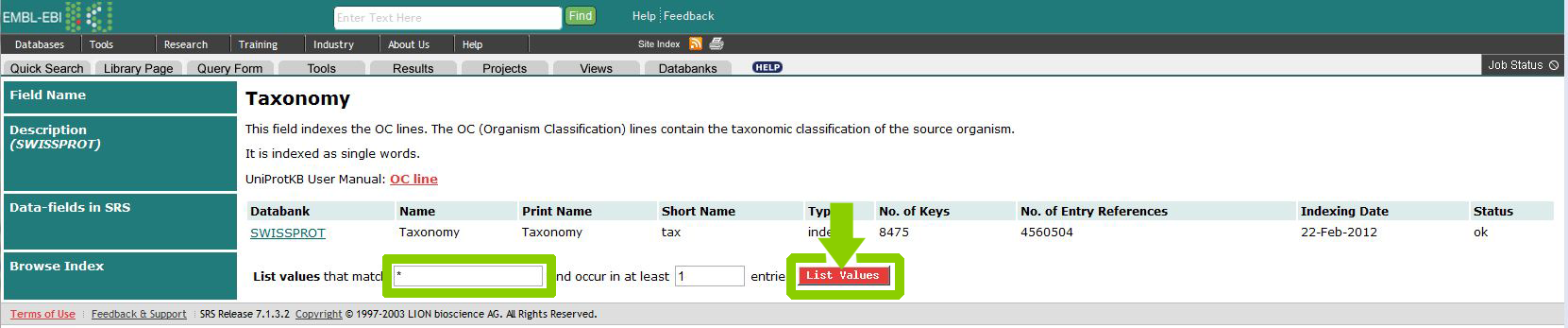
Узнать, названия каких таксонов начинаются на bacil, можно с помощью следующего поскового запроса:
bacil*
В результате получаем:
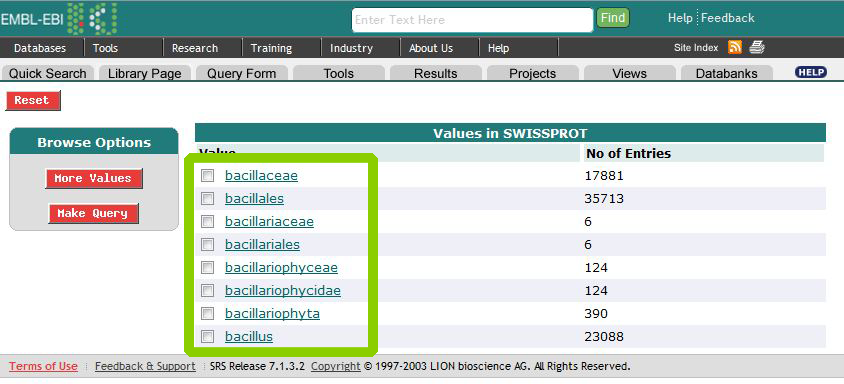
Итак, на bacil начинаются названия следующих таксонов:
bacillaceae
bacillales
bacillariaceae
bacillariales
bacillariophyceae
bacillariophycidae
bacillariophyta
bacillus
Чтобы выяснить, сколько в банке SwissProt записей, описывающих белки из рода Bacillus, составим следующий запрос:
bacillus
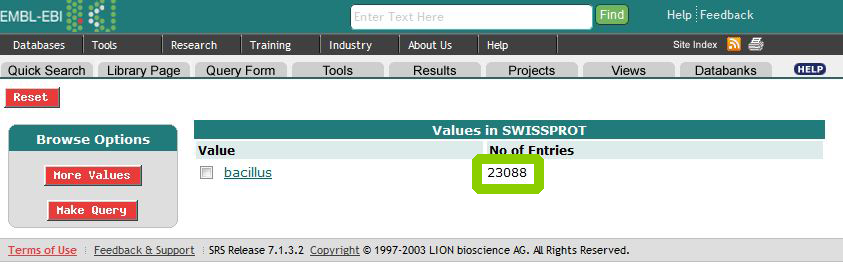
Итак, в банке SwissProt 23088 записей, описывающих белки из рода Bacillus.
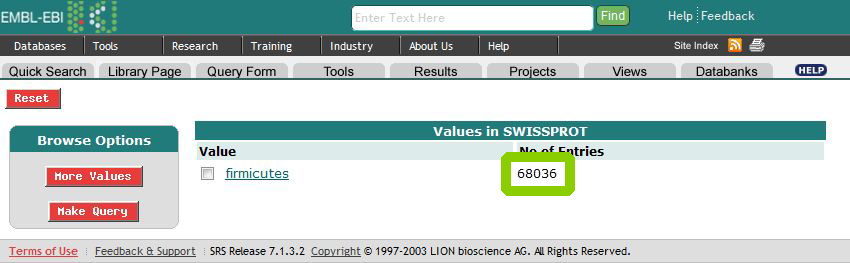
Аналогично выясняем, что в банке SwissProt 68036 записей, описывающих белки из рода Firmicutes:
Описать особенности сервиса MRS по сравнению с SRS
Используя MRS, находим запись белка CDD_BACSU в SwissProt:
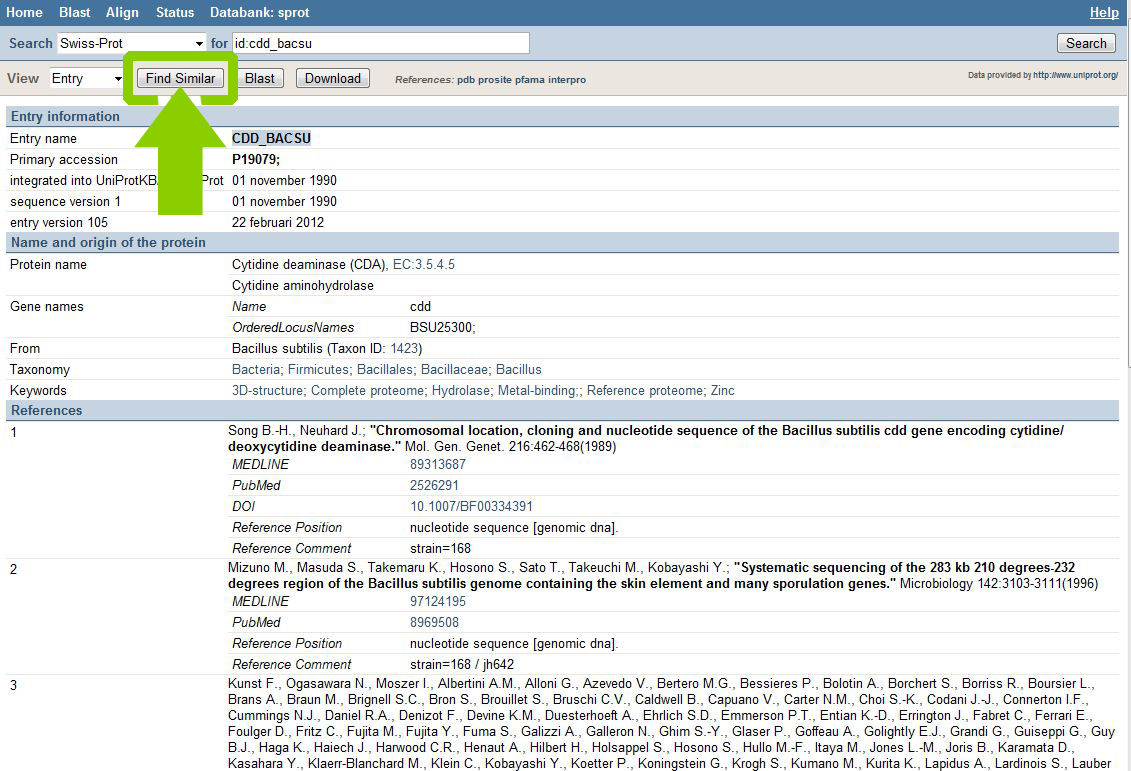
Нас интересует функция Find similar (найти похожие записи).
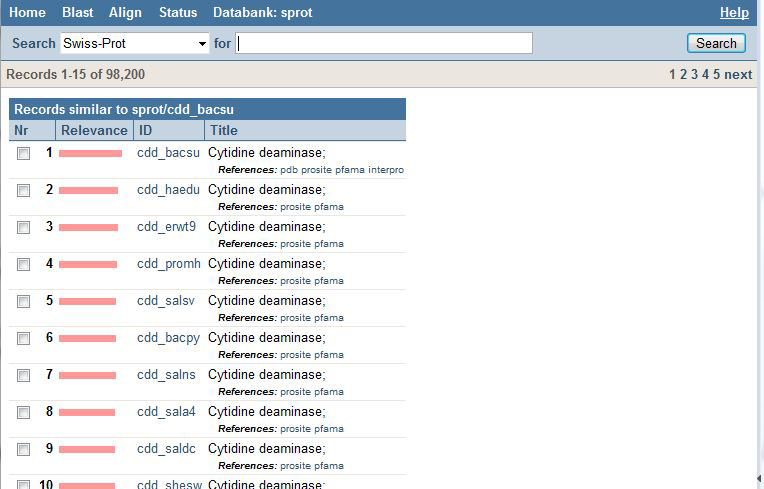
Если проанализировать полученные с помощью неё результаты, то можно сделать предположение, что сначала выдаются записи со таким же кодом белка (в даном случае - cdd), затем - записи с таким же кодом организма и со сходной информацией о белке (описание и функции).
Можно отметить необходимые записи среди найденных, например, для того, чтобы загрузить их. Однако здесь мы сталкиваемся с ограничением: отмеченные результаты актуальны только для текущей страницы и, соответственно, загрузить можно только видимые на странице результаты; при переходе на другую страницу загрузить записи, отмеченные на предыдущей странице, нельзя; однако если вернуться обратно на страницу, на которой уже были отмечены некоторые результаты, мы увидим, что наши отметки «сохранились».
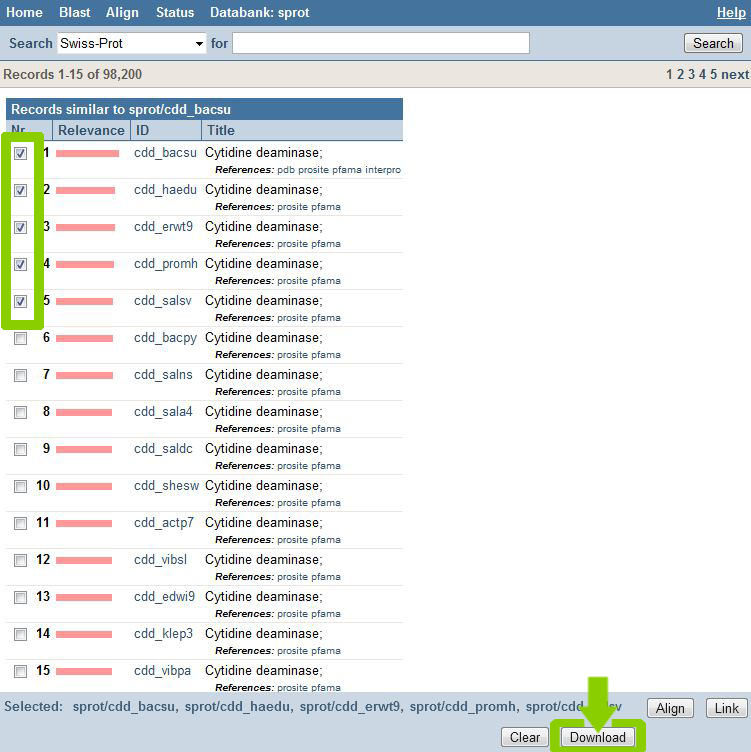
В MRS также присутствует возможность поиска найденной информации в других банках данных.
Представленный объём информации заметно меньше, нежели в SRS. Однако MRS является достаточно «быстрой» и удобной поисковой системой для
работы с биологическими и медицинскими банками данных.
Кроме того, MRS имеет
открытый исходный код, что может быть немаловажным аргументом за использование MRS.
Найти записи SwissProt, связанные с научными интересами известного российского биолога А.А.Нейфаха
Для начала узнаем, как правильно записать фамилию учёного.

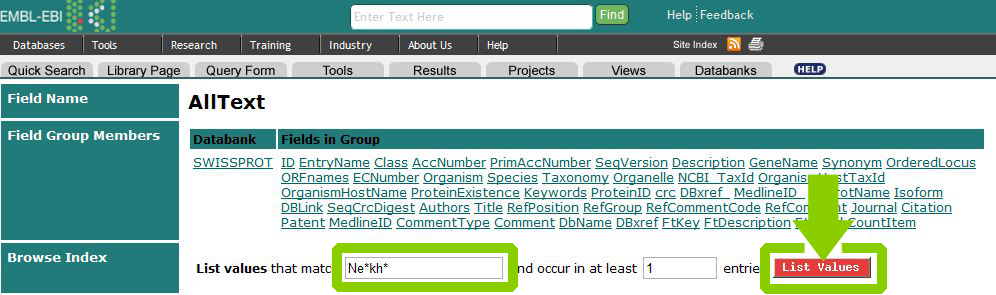
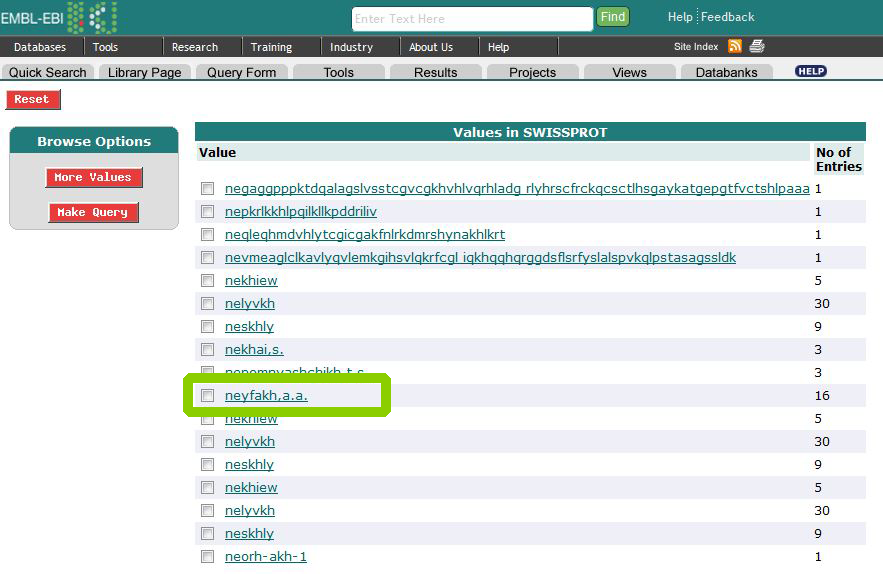
Теперь необходимо выполнить поиск по запросу neyfakh в поле Reference: Authors. При этом можно выбрать поля, которые мы желаем увидеть в выдаче, а именно Description, References: Authors, References: Title и References: Year:
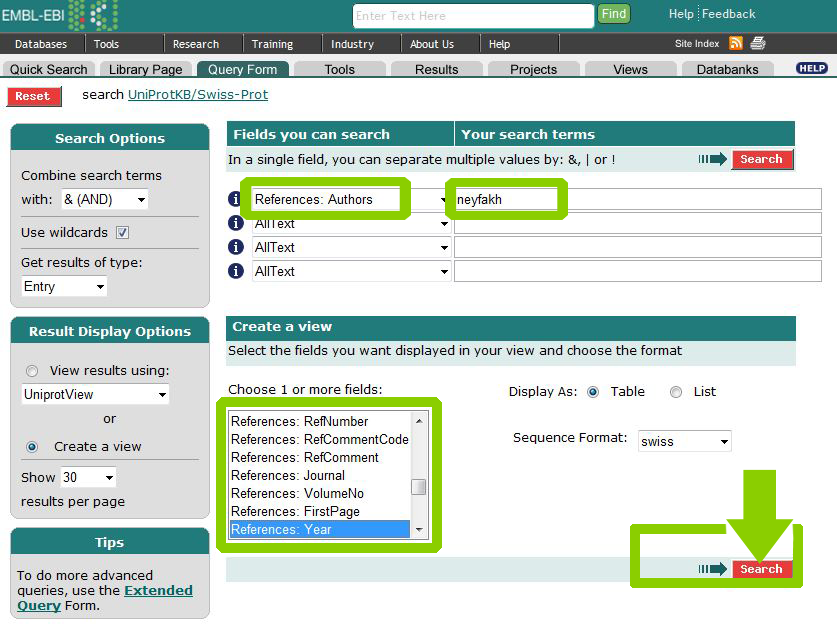
Затем, отметив необходимые записи, мы можем загрузить результаты в виде таблицы Excel, содержащей необходимые нам поля.
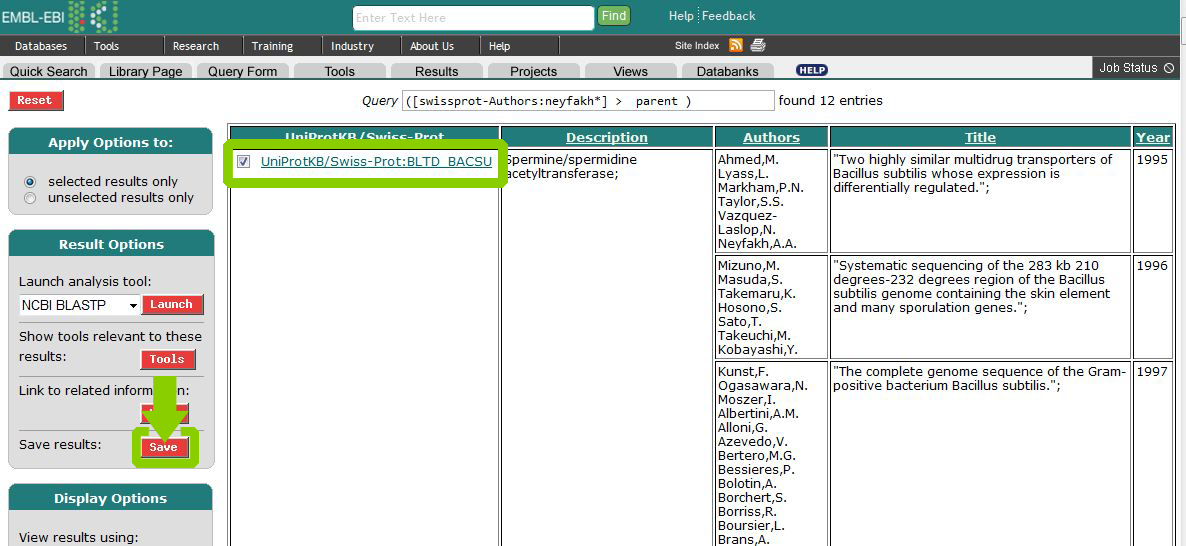
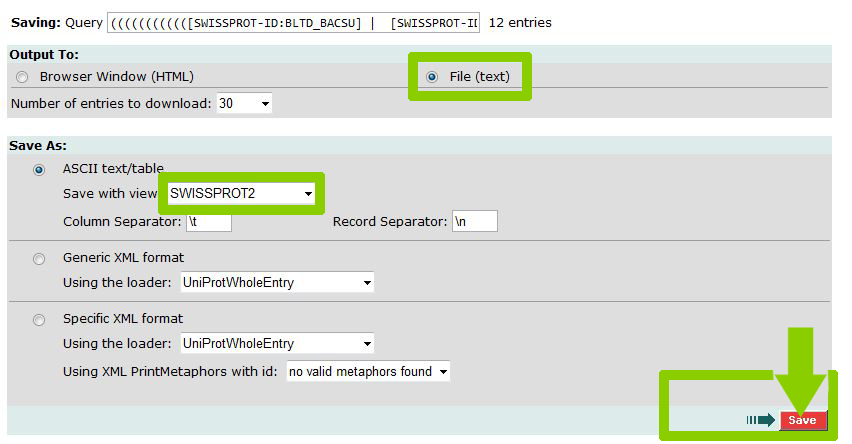
В итоге получаем файл neyfakh.xls.
Если в задании подразумевалось сохранение только тех записей, в которых Нейфах - автор именно первой публикации, то, исключив неподходящие под этот критерий записи, получим следующий файл: neyfakh_onlyfirst.xls.
Как видно из результатов, Нейфах занимался изучением бактерии Bacillus subtilis, является одним из авторов статей на тему генетического контроля синтеза белков.
Описать некоторые существенные отличия в формате записи белка CDD_BACSU в БД Proteins на сайте NCBI от формата Uniprot
Произведя поиск в Entrez, находим нужную нам страницу с записью белка.
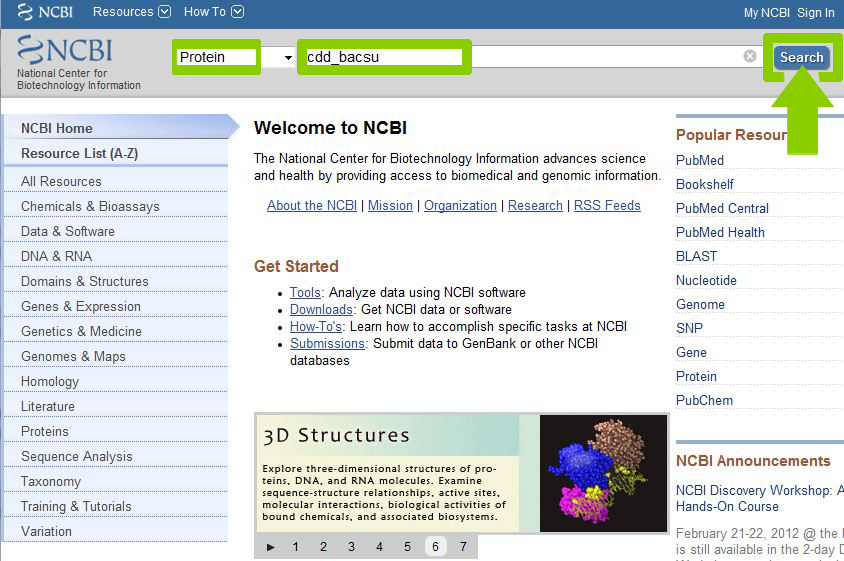
Сравнивая формат записи белка в БД Proteins на сайте NCBI с форматом записи Uniprot, мы может сделать следующие выводы:
- есть различия в расположении некоторых полей информации;
- названия полей в формате на сайте NCBI записаны полностью, в отличие от сокращений в Uniprot (например, KEYWORDS вместо KW), что, возможно, более удобно при использовании; однако при при работе с форматом Uniprot трудностей с восприятием сокращений, на мой взгляд, не возникает;
- при использовании формата на сайте NCBI более приятна и удобна работа с полем FEATURES, сравнивая с форматом Uniprot; например, при нажатии на обозначение участка, к которому относится особенность (gene, Region, Site), выделяется соответствующий участок последовательности, при этом можно легко получить, если это требуется, запись участка в формате .fasta и др.;
- к каждой особенности в первом из рассматриваемых форматов имеются полезные примечания (note, experiment);
- при работе с записью на сайте NCBI легко и быстро можно перейти к графическому режиму (и многим другим режимам) работы с записью с помощью соответствующей ссылки.
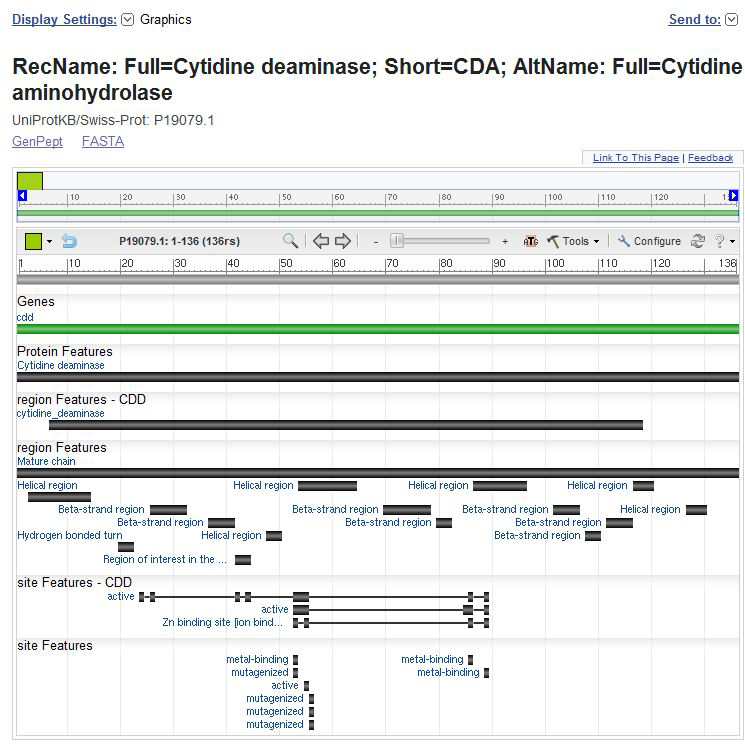
Во многом сравниваемые форматы записей схожи. Работать с записью на сайте NCBI, по моему скромному мнению, не менее удобно, чем с форматом Uniprot, в том числе благодаря наличию удобных настроек отображения необходимой информации и предоставлению средств работы с ней.
Ссылки
- Документ из банка UniProt для белка CDD_BACSU.
- Описание всех записей банка трехмерных структур PDB, относящихся к белку CDD_BACSU.
- Полноразмерные последовательности найденных белков в формате .fasta.
- Список найденных последовательностей с наличием требуемых в задании полей в формате .xls.
- Записи SwissProt, связанные с научными интересами известного российского биолога А.А.Нейфаха.
