Занятие 9: Паттерны и банк PROSITE
Создание паттернов по множественному выравниванию.
Поиск по паттернам в банке данных Swiss-Prot.
В программе JalView рассмотрим множественное выравнивание, полученное при изучении последовательностей белка CDD_BACSU и его гомологов в первом задании предыдущего занятия.
Выберем фрагмент выравнивания дляной 8 - 20 аминокислотных остатков для дальнейшего исследования, стараясь, чтобы 1/3 - 1/2 колонок фрагмента были консервативны на 70 – 100%:

Изображение (1) в оригинальном размере.
Изображение выбранного фрагмента выравнивания с раскраской по по BLOSUM62:
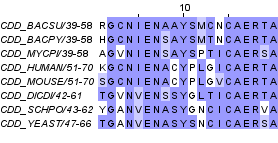
Изображение (2) в оригинальном размере.
Загрузить файл с фрагментов выравнивания.
Рассмотрев выбранный фрагмент выравнивания, создадим три паттерна, пользуясь основными элементами синтаксиса паттернов.
Паттерн - фрагмент последовательности
Первый паттерн в точности является фрагментом последовательности белка CDD_BACSU:
RGCNIENAAYSMCNCAERTA
Проведём поиск последовательностей банка Swiss-Prot, включающих мотивы, соответствующие указанному паттерну, на сайте PROSITE. Для этого на странице ScanProsite tool введём паттерн в поле для поиска мотивов:
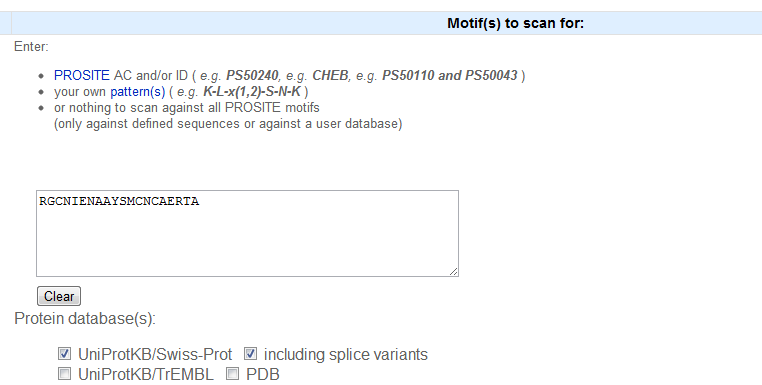
В результате получаем следующее (формат выдачи результата - Plain text tabular):

Таким образом, мотив, удовлетворяющий паттерну, являющемуся фрагментом последовательности белка CDD_BACSU, найден только в одной последовательности банка Swiss-Prot, причём эта последовательность является последовательностью белка CDD_BACSU.
Сильный паттерн
Построим сильный паттерн так, чтобы он распознавал все белки нашей выборки и только их:
[RHAKSTY]-G-[CVA]-N-[IV]-E-N-[AS]-[ASC]-Y-[SPG]-[MPLN]-[CTG]-[NIV]-C-A-E-R-[STV]-A
Проведём поиск последовательностей банка Swiss-Prot, включающих мотивы, соответствующие составленному паттерну:
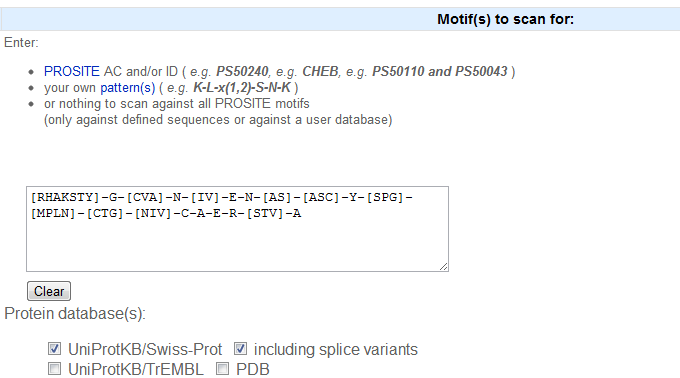
В результате получаем следующее (формат выдачи результата - Plain text tabular):
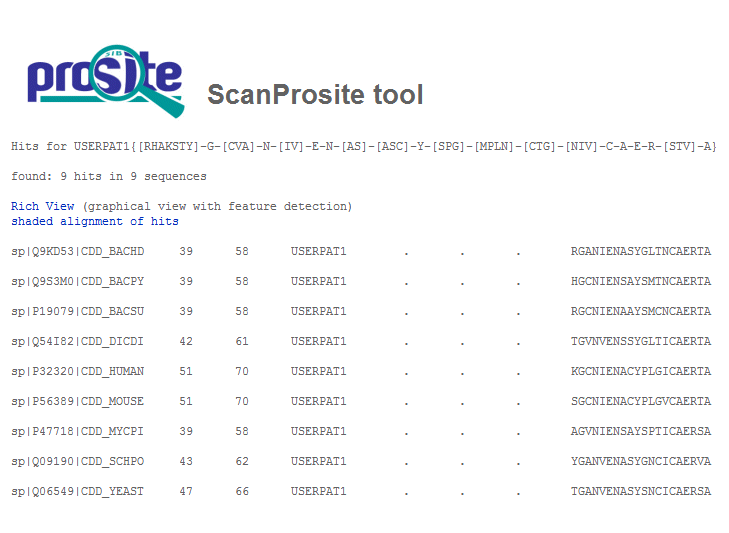
Таким образом, мотив, удовлетворяющий составленному сильному паттерну, найден в девяти последовательностях банка Swiss-Prot, восемь из которых являются последовательностями из рассматриваемого выравнивания. Девятой находке соответствует последовательность белка CDD_BACHD.
Все последовательности из рассматриваемого выравнивания найдены.
Слабый паттерн
Построим слабый паттерн, стремясь к тому, чтобы он находил всех близких родственников белка CDD_BACSU. Например, в позиции 1 заменим список букв символом x; в позициях 5, 8 - 9, 11 - 14, 19 разнообразим список допустимых остатков.
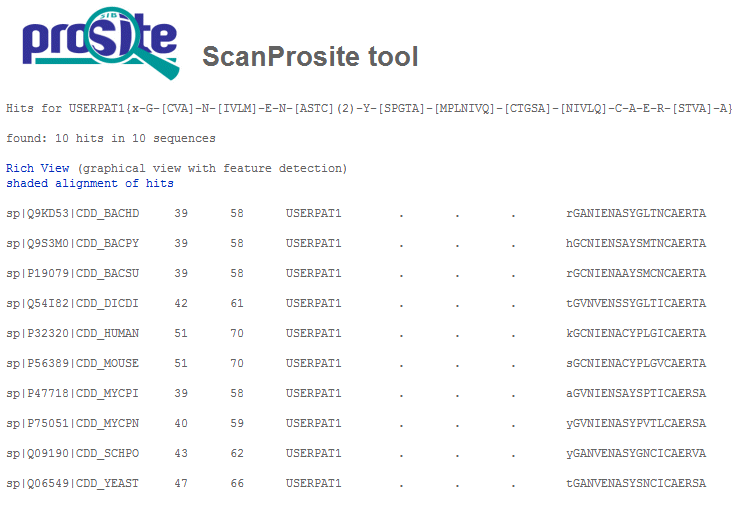
x-G-[CVA]-N-[IVLM]-E-N-[ASTC](2)-Y-[SPGTA]-[MPLNIVQ]-[CTGSA]-[NIVLQ]-C-A-E-R-[STVA]-A
Результат поиска последовательностей банка Swiss-Prot, включающих мотивы, соответствующие составленному паттерну, выглядит следующим образом:
Попробуем ещё ослабить паттерн:
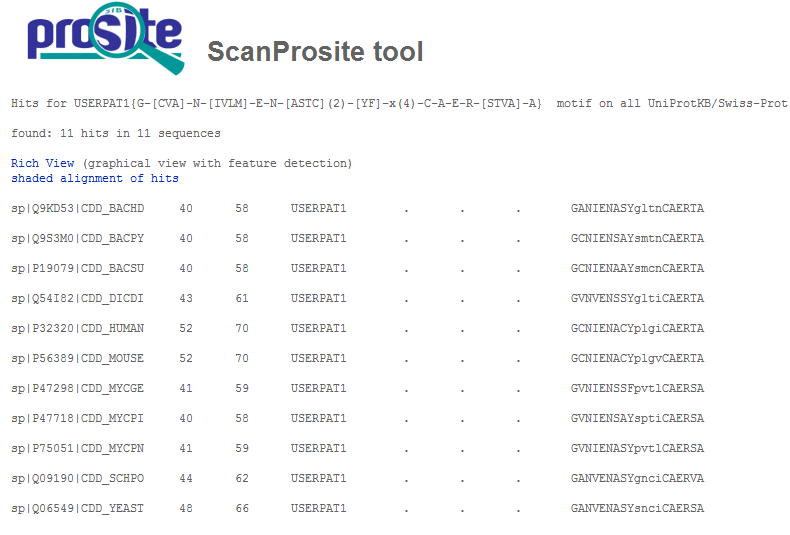
G-[CVA]-N-[IVLM]-E-N-[ASTC](2)-[YF]-x(4)-C-A-E-R-[STVA]-A
Результаты поиска последовательностей банка Swiss-Prot претерпели некоторые изменения:
Произведём сравнение полученных результатов с выдачей программы BLAST (будем искать белки, родственные белку CDD_BACSU (AC P19079):
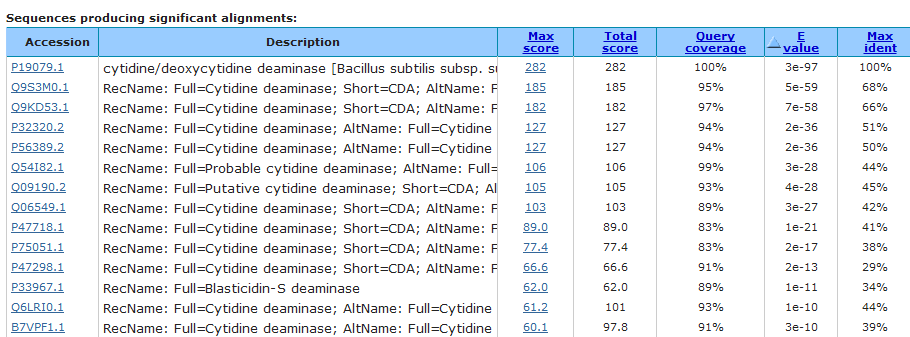
Заметим, что результаты поиска по составленному паттерну включают в себя 11 первых результатов выдачи программы BLAST.
Также обратим внимание на то, что в результатах выдачи программы BLAST с номерами >11 последовательность искомого фрагмента довольно сильно искажается, поэтому далее не встречаются мотивы, соответствующие составленному паттерну. По той же причине ослабление паттерна для соответствия мотивов в последующих последовательностях белков составленному паттерну является задачей трудоёмкой (можно даже поставить под сомнение её выполнимость, учитывая то, насколько различаются выбранные фрагменты в последующих выравниваниях).
И всё же попробуем в очередной раз ослабить паттерн:
G-[CVA]-x(4)-[ASTC](2)-[YF]-x(4)-C-A-[NQDE]-[KR]-[STVA]-A
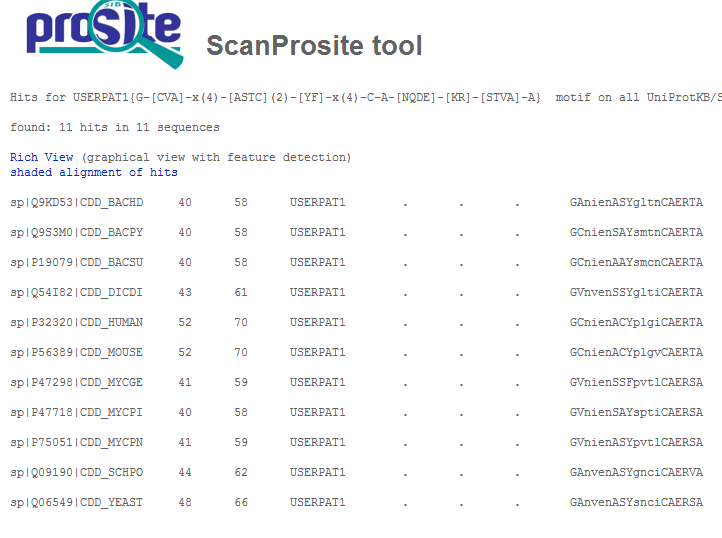
Как видим, даже такое ослабление паттерна не принесло никаких результатов.
Таким образом, мотив, удовлетворяющий составленному слабому паттерну, найден в одиннадцати последовательностях банка Swiss-Prot, восемь из которых являются последовательностями из рассматриваемого выравнивания. Все последовательности из рассматриваемого выравнивания найдены.
Нетрудно заметить, что все найденные по паттерну последовательности банка Swiss-Prot являются последовательностями белка CDD (цитидин-дезаминаза), представленного в различных организмах, в том числе в человеке.
Паттерны и результаты поиска последовательностей банка Swiss-Prot, включающих мотивы, соответствующие каждому из полученных паттернов, запишем в виде таблицы:
Таблица Результаты поиска по паттернам в банке данных Swiss-Prot
| Характеристика паттерна | Паттерн | Число последовательностей банка |
Количество найденных последовательностей из выравнивания |
| Фрагмент последовательности | RGCNIENAAYSMCNCAERTA | 1 (одна) | 1 (одна) |
| Сильный | [RHAKSTY]-G-[CVA]-N-[IV]-E-N-[AS]-[ASC]-Y-[SPG]-[MPLN]-[CTG]-[NIV]-C-A-E-R-[STV]-A | 9 (девять) | 8 (восемь - все последовательности выравнивания) |
| Слабый | G-[CVA]-N-[IVLM]-E-N-[ASTC](2)-[YF]-x(4)-C-A-E-R-[STVA]-A | 11 (одиннадцать) | 8 (восемь - все последовательности выравнивания) |
Поиск последовательностей банка Swiss-Prot, включающих мотивы, соответствующие составленным паттернам, можно также осуществлять при помощи программы fuzzpro пакета EMBOSS. Для составленных паттернов (фрагмент последовательности, сильный паттерн, слабый паттерн) выполним следующие команды соответственно:
fuzzpro -sequence sw:* -pattern RGCNIENAAYSMCNCAERTA -outfile first_pattern.fuzzpro
fuzzpro -sequence sw:* -pattern [RHAKSTY]-G-[CVA]-N-[IV]-E-N-[AS]-[ASC]-Y-[SPG]-[MPLN]-[CTG]-[NIV]-C-A-E-R-[STV]-A -outfile strong_pattern.fuzzpro
fuzzpro -sequence sw:* -pattern "G-[CVA]-N-[IVLM]-E-N-[ASTC](2)-[YF]-x(4)-C-A-E-R-[STVA]-A" -outfile weak_pattern.fuzzpro
В результате получим соответствующие файлы: first_pattern.fuzzpro, strong_pattern.fuzzpro, weak_pattern.fuzzpro. При этом, как мы видим, результаты выполнения задания (т.е. число найденных последовательностей, число последовательностей из выравнивания) не изменились.
Оценка встречаемости паттерна
Необходимо оценить, сколько раз слабый паттерн встретится в случайном банке размером Swiss-Prot. Для этого сначала необходимо вычислить примерную частоту встречаемости каждой буквы в последовательностях, содержащихся в этом банке. Это можно сделать, например, при помощи команды wordcount пакета EMBOSS (оценим встречаемость букв в последовательностях белков Bacillus subtilis):
wordcount sw:*_bacsu bacsu.wordcount -wordsize 1
В результате получим файл bacsu.wordcount, содержащий данные о том, сколько раз каждая буква встретилась в заданной области поиска. Для вычисления частоты встречаемости каждой буквы напишем файл wordcount.py. После запуска этого файла мы получим файл wordcount.txt, содержащий искомые примерные частоты встречаемости каждой буквы. Теперь оценим, сколько раз слабый паттерн встретится в случайном банке размером примерно 20 миллионов букв, произведя математические вычисления:
(0.07 * (0.01 + 0.01 + 0.08) * 0.04 * (0.07 + 0.07 + 0.1 + 0.03) * 0.07 * 0.04 * (0.08 + 0.06 + 0.05 + 0.01)^2 * (0.04 + 0.05) * 0.01 * 0.08 * 0.07 * 0.04 * (0.06 + 0.05 + 0.07 + 0.08) * 0.08) * (20 * 10^6) = 7.10106808 * 10^(-10)
Поиск паттерна в перемешанном банке (значение shuffle в поле randomize databases) результатов не даёт: no hit!. (Это неудивительно, если учесть, какое число мы получили при вычислениях.) Как мы видим, в неперемешанном банке удалось найти на 11 результатов больше. В данном случае действительно можно говорить о родственности найденных по слабому паттерну белков.
Поиск и описание мотивов в белке CDD_BACSU по данным БД Prosite
Таблица Все описанные в PROSITE мотивы в заданном белке CDD_BACSU
| Идентификатор документа Prosite (AC) | Название мотива | Краткое описание мотива | Тип подписи | Паттерн | Специфичность подпись | Число найденных в белке мотивов |
| PS00903 | CYT_DCMP_DEAMINASES | Подпись участка связывания цинка цитидин- и деоксицитидилат-деаминазы | Паттерн | [CH]-[AGV]-E-x(2)-[LIVMFGAT]-[LIVM]-x(17,33)-P-C-x(2,8)-C-x(3)-[LIVM] | Специфична | 1 |
| PS00007 | TYR_PHOSPHO_SITE | Сайт фосфорилирования тирозинкиназы | Паттерн | [RK]-x(2)-[DE]-x(3)-Yor[RK]-x(3)-[DE]-x(2)-Y | Неспецифична | 1 |
| PS00006 | CK2_PHOSPHO_SITE | Сайт фосфорилирования казеин-киназы-2 | Паттерн | [ST]-x(2)-[DE] | Неспецифична | 3 |
| PS00008 | MYRISTYL | Сайт N-миристоилирования | Паттерн | G-{EDRKHPFYW}-x(2)-[STAGCN]-{P} | Неспецифична | 1 |
