|
|
С помощью blast2seq я построила выравнивания геномов (то есть целых хромосом) для разных близкородственных бактерий и архей. На рисунках 1-3 представлены
карты сходства хромосом.
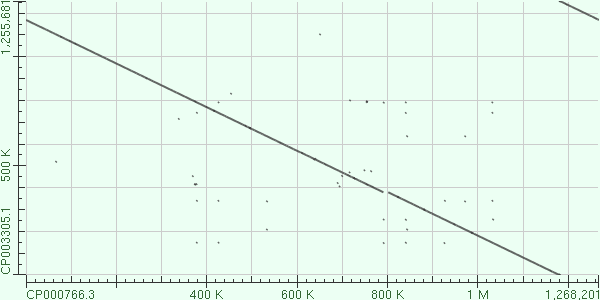
Рисунок 1. Карта сходства геномов двух штаммов бактерии Rickettsia rickettsii: по горизонтали Rickettsia rickettsii str. Iowa
(CP000766.3), по вертикали Rickettsia rickettsii str. Brazil
(CP003305.1).
По рисунку 1 видно, что геномы этих бактерий имеют достаточно высокое сходство. Линия, отображающая совпадающие участки, расположена в другом направлении (как будто инвертированно),
скорее всего, из-за того, что в геноме штамма Brazil была секвенирована комплементарная цепь, при этом с некоторым сдвигом относительно генома штамма Iowa. Появление двух линий на
карте сходства, связано с различием в том, с какого участка начинается запись в базе данных.
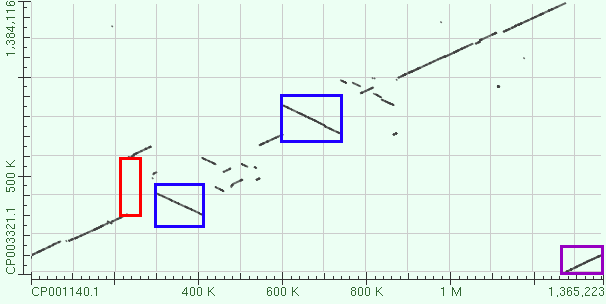
Рисунок 2. Карта сходства геномов двух видов археи Desulfurococcus: по горизонтали Desulfurococcus kamchatkensis штамм 1221n
(CP001140.1), по вертикали Desulfurococcus fermentans штамм DSM 16532
(CP003321.1).
На рисунке 2 разными цветами обведены примеры крупных эволюционных событий: синим показаны инверсии (с 300kb по 410kb и с 600kb по 740kb, координаты указаны по горизонтали),
красным - вставка в Desulfurococcus fermentans (с 300kb по 600kb, координаты по вертикали). Участок, обведенный фиолетовым, возможно, появился из-за того, что секвенирование
хромосом в этих бактериях начиналось в разных местах, то есть начало последовательностей в записях не совпадает.
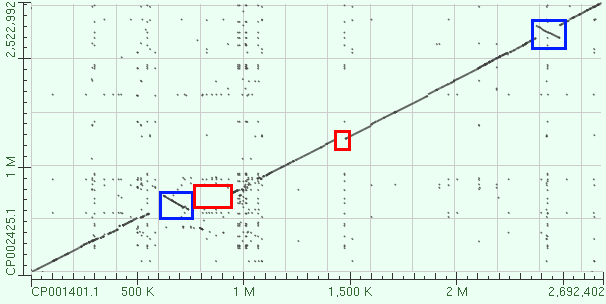
Рисунок 3. Карта сходства геномов двух штаммов археи Sulfolobus islandicus: по горизонтали Sulfolobus islandicus штамм M.16.27
(CP001401.1), по вертикали Sulfolobus islandicus штамм REY15A
(CP002425.1).
На рисунке 3 использованы те же обозначения. Инверсии с 650kb по 750kb и с 2.4Mb по 2.5Mb. Вставки в Sulfolobus islandicus штамм M.16.27 с 750kb по 950kb и с 1.45Mb по 1.5Mb.
Координаты указаны по горизонтали.
Построение нуклеотидного пангенома с помощью NPGE
С помощью пакета NPG-explorer был построен нуклеотидный пангеном для четырех штаммов бактерии Rickettsia rickettsii: str. Brazil
(CP003305.1), str. Iowa (CP000766.3),
str. Arizona (CP003307.1) и str. Colombia (CP003306.1).
Эта грамотрицательная бактерия является облигатным внутриклеточным паразитом, поражает клетки эндотелия сосудов и вызывает пятнистую лихорадку Скалистых гор - заболевание, распространенное в Северной Америке, Бразилии и Колумбии.
Ее геном представлен одной кольцевой хромосомой.
Для построения пангенома я создала файл genomes.tsv с указаниями идентификаторов последовательностей геномных ДНК и аннотаций генов.
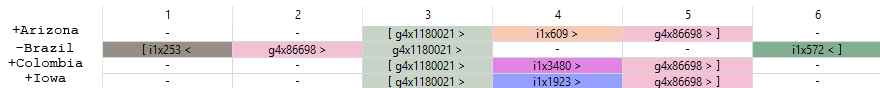
Я начала разбираться с полученными результатами с описания синтеничных участков - g-блоков. Всего их найдено 7: два из них совпадают во всех геномах (g4x86698 и g4x1180021), остальные несколько отличаются
числом повторяющихся участков (i-блоки). Порядок расположения g-блоков во всех хромосомах представлен на рисунке 4 и в файле g-blocks.xslx. Одинаковые во всех геномах блоки отмечены одним цветом.
В геномах Arizona, Iowa и Colombia (названия даны по названиям штаммов) g-блоки расположены в одном и том же порядке, а между ними находятся несколько отличающиеся i-блоки (в i-блоках различное число
повторяющихся участков, однако порядок их расположения в геноме одинаков). Геном Brazil взят в обратном направлении, и в нем произошла перестановка двух g-блоков. Если подробнее рассмотреть i-блоки, можно увидеть,
что два таких блока из генома Brazil соответствуют одному блоку из любого другого изученного генома, то есть порядок расположения подблоков в этих i-блоках одинаков. Однако начало генома Brazil не совпадает с началом
остальных геномов. Если объеденить i-блоки и перевернуть весь геном Brazil, то мы получим то же расположение блоков, что и в других хромосомах. В принципе, это согласовывается с полученной картой сходства для геномов
Brazil и Iowa (рисунок 1).
Рисунок 4. Расположения g-блоков в хромосомах четырех штаммов бактерии Rickettsia rickettsii (обозначены как Arizona, Brazil, Colombia, Iowa) по результатам
пангенома, построенного с помощью пакета NPG-explorer. Знаки "+" и "-" показывают, в каком направлении был взят геном. Квадратные скобки означают границы геномов.
В таблице 1 представлена информация об s-блоках, - блоках, которые встречаются во всех геномах.
|
Таблица 1. Информация об s-блоках из пангенома для четырех штаммов бактерии Rickettsia rickettsii.
|
| Число |
118 |
| Суммарная длина |
1,240,425 |
| Процент от длины генома в среднем |
98.035% |
| Процент консервативных позиций (при объединении всех s-блоков) |
99.8965% |
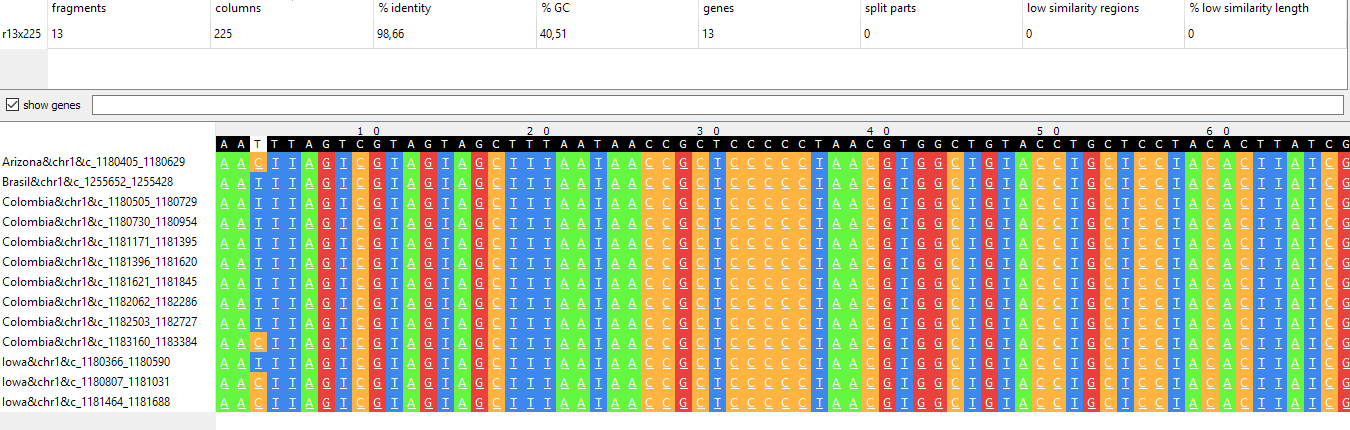
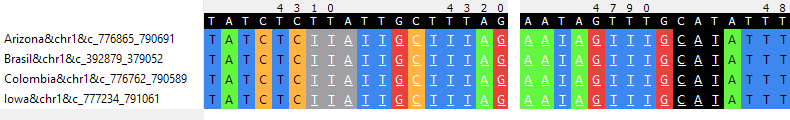
В таблице 2 описано три r-блока (блоки с повторами). На рисунке 5 показан первый из описанных r-блоков (r13x225), изображение получено с помощью визуализатора qnpge.
В первый блок входят по одному участку из геномов Arizona и Brazil, 8 из генома Colombia и 3 из Iowa. В геномах Arizona и Iowa эти участки соответствуют частям генов белка A наружной
мембраны (outer membrane protein A). В геноме Colombia аннотирован гипотетический белок, в геноме Brazil для этого участка нет аннотаций.
Во второй блок входят по два повтора из каждого генома. В геноме Arizona один из повторов не аннотирован, в геноме Brazil этот повтор соответствует учатску гена, кодирующего,
предположительно, АТФ-связывающий белок (putative ATP-binding protein), а в геноме Colombia он соответствует участку гену белка NTP-азы семейства NACHT (NACHT family NTPase). Другие повторы в
этих геномах содержат аннотации двух предположительных генов, кодирующих АТФ-зависимую субъединицу C протеазы (ATP-dependent protease subunit C (ClpC)) и белок, содержащий NACHT-домен
(NACHT domain-containing protein). Возможно, эти последовательности являются генами, так как blast часто выдает соответствующие находки. Такой результат blast возможен, так как вторые повторы
действительно кодируют данные белки. В геноме Iowa оба повтора соответствует участкам генов гипотетических АТФ-связывающих белков.
В третий блок также входят по два повтора из каждого генома. Аннотации есть только в геноме Iowa для обоих участков (части генов гипотетических белков), в остальных геномах аннотаций нет.
|
Таблица 2. Информация о некоторых r-блоках из пангенома для четырех штаммов бактерии Rickettsia rickettsii.
|
| Блок |
r13x225 |
r8x234 |
r8x774 |
| Число фрагментов |
13 |
8 |
8 |
| Число генов (во всех фрагментах из данного блока) |
13 |
3 |
0 |
| Длина |
225 |
234 |
774 |
| Процент консервативных колонок |
98,66% |
93,37% |
99,74% |
Рисунок 5. Один из описанных r-блоков (r13x225). Сверху показаны параметры блока, снизу - участок выравнивания последовательностей из изучаемых геномов в этом блоке.
Изображение получено с помощью визуализатора qnpge.
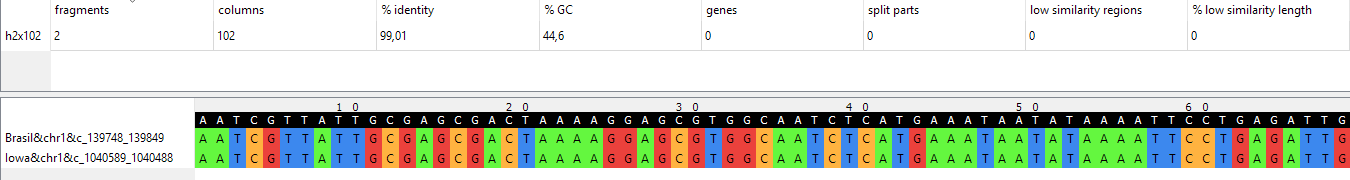
В таблице 3 описано два h-блока. Это такие блоки, в которые входят участки не из всех геномов, то есть в некоторых геномах произошла делеция данного участка. На рисунке 6
представлен один из таких блоков, h2x102.
Первый блок включает участки из геномов Arizona, Colombia и Iowa. Значит, делеция этого участка произошла в геноме Brazil. В геномах Arizona и Colombia в состав этого
участка входят гены, кодирующие 2 гипотетических белка и белки 3-гидроксиацил-CoA дегидрогеназы и амидофосфорибозилтрансферазы. В геноме Iowa помимо указанных генов есть
также гены еще 4 гипотетических белков.
Во второй блок входят участки из геномов Brazil и Iowa. Делеции произошли в двух других геномах. Эти участки не аннотированы.
|
Таблица 3. Информация о некоторых h-блоках из пангенома для четырех штаммов бактерии Rickettsia rickettsii.
|
| Блок |
h3x9671 |
h2x102 |
| Число фрагментов |
3 |
2 |
| Число генов (во всех фрагментах из данного блока) |
15 |
0 |
| Длина |
9,671 |
102 |
| Процент консервативных колонок |
99,89% |
99,01% |
Рисунок 6. Один из описанных h-блоков (h2x102). Сверху показаны параметры блока, снизу - участок выравнивания последовательностей из двух геномов (Brazil и Iowa),
в которых имеются соответствующие последовательности. Изображение получено с помощью визуализатора qnpge.
Найден один уникальный блок (u1x115), в который входит участок из генома Iowa. Этот участок соответствует части гена, кодирующего гипотетический белок. Я проверила, есть ли гомологи
данной последовательности в других организмах с помощью blastn.
Гомологичные последовательности встречаются в геномах других штаммов бактерии Rickettsia rickettsii (например, в штаммах R, Hauke, Hino, 'Shelia Smith'). Еще две находки с E-value
0.027 обнаружены в геномах рыб Oryzias latipes и Danio rerio. Горизонтальный перенос из этих организмов (или в них из бактерий) маловероятен. Остальные находки имеют слишком
низкое сходство, чтобы считать их продуктами горизонтального переноса, несмотря на то, что среди найденных организмов есть много бактерий. Скорее всего, эта последовательность возникла
именно у данного вида бактерий Rickettsia rickettsii, но как она передавалась между штаммами, установить трудно.
На рисунках 7 и 8 представлены расхождения между аннотациями генов из одного блока, а также соответствующие участки выравнивания последовательностей в стабильных блоках.
Первый пример - из блока s4x14676. Сверху показано описание генов из генома Arizona (рисунок 7a), снизу - из генома Iowa (рисунок 7b). Указаны те гены, которые аннотированы в штамме Arizona
(и в других штаммах, Brazil и Colombia), но не отмечены в штамме Iowa (гены обведены красной рамкой). Для того, чтобы подтвердить, что данные записи относятся к одному и тому же месту в геноме,
на рисунках показаны и окружающие совпадающие гены. Также на рисунках 7c и 7d показаны старт- и стоп-кодоны отмеченных генов, что подтверждает их ортогональность, так как они расположены точно
друг под другом в выравнивании блока. На этих двух рисунках видно, что ген не аннотирован в геноме Iowa, но аннотирован в остальных.
Второй пример - из блока s4x13828. Сверху - геном Colombia (рисунок 8a), снизу - геном Iowa (рисунок 8b). Отмечен ген, который аннотирован во всех четырех геномах, но в геноме Iowa указана
другая функция продукта: гипотетический цитоплазматический белок в штамме Iowa; белок RimP, участвующий в метаболизме рибосом (ribosome maturation protein RimP), - в других штаммах.
Последовательности и координаты этих генов во всех геномах практически совпадают. На рисунке 8с представлено выравнивание этого участка последовательности, показывающее, что это один и
тот же ген во всех геномах.
В целом, больше всего отличий возникает между геномом Iowa и остальными тремя геномами. Возможно, это вызвано тем, что запись для штамма Iowa последний раз изменена 3 февраля 2015 года, и это уже третья
версия, а записи для других штаммов не изменялись с 31 января 2014 года.

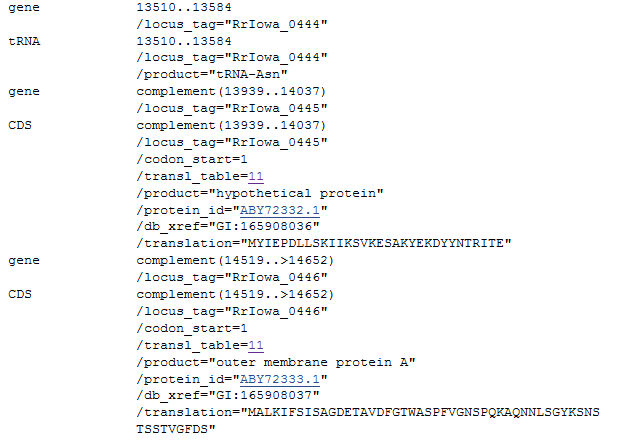
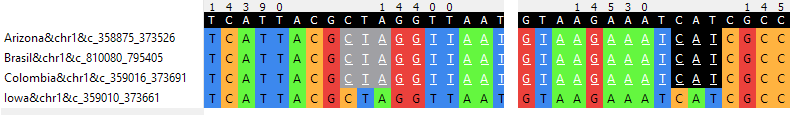
Рисунок 7a (сверху). Аннотации генов из генома Arizona, блок s4x13828. Красной рамкой обведены гены, не описаны в штамме Iowa.
Рисунок 7b (снизу). Аннотации генов из аналогичного участка генома Iowa, блок s4x14676, координаты участка 13510-14652.
Рисунок 7c (сверху). Выравнивание отмеченного гена рекомбиназы B (cassette chromosome recombinase B). Слева показан стоп-кодон гена (отмечен серым), справа - старт-кодон (черный).
Ген закодирован на комплементарной цепи. Розовым выделено перекрывание генов, так как в этом месте на прямой цепи записана последовательность Asn-тРНК.
Рисунок 7d (снизу). Выравнивание отмеченного гена Plasmid maintenance system antidote protein. Слева показан стоп-кодон гена (отмечен серым), справа - старт-кодон (черный).
Ген закодирован на комплементарной цепи.
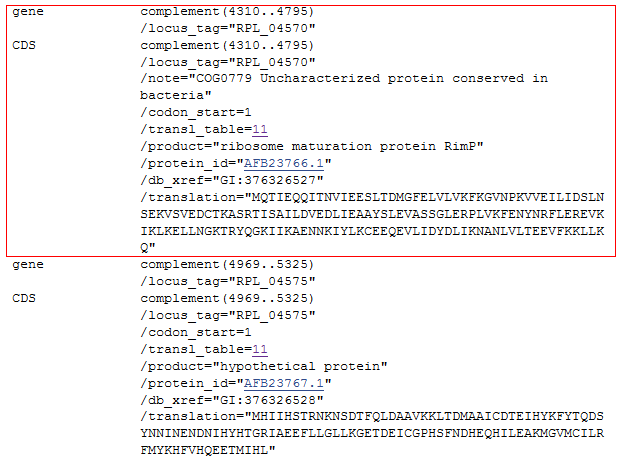
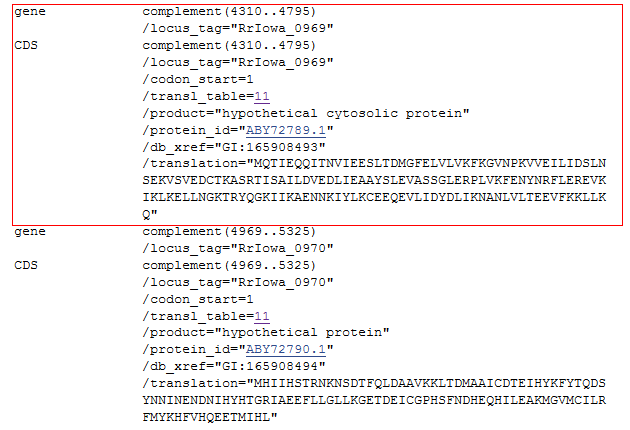
Рисунок 8a (сверху). Аннотации генов из генома Colombia, блок s4x13828. Красной рамкой обведены гены с раличающимися аннотациями.
Рисунок 8b (снизу). Аннотации генов из аналогичного участка генома Iowa, блок s4x14676.
Рисунок 8c. Выравнивание отмеченного гена, кодирующего разные продукты в геномах Iowa и остальных штаммах. Слева показан стоп-кодон гена (отмечен серым),
справа - старт-кодон (черный). Ген закодирован на комплементарной цепи.
Построение нуклеотидных пангеномов близкородственных бактерий или архей позволяет определить глобальные изменения, произошедшие при эволюции различных видов
или штаммов, такие как инверсии или дупликации генов, сравнить число повторяющихся участков в геномах, выделить отдельные эволюционные группы штаммов. Также этот метод
позволяет исправить аннотации генов и их продуктов, дополнить уже известные данные.
|