|
|||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
Для тРНК из PDB-структуры 1QU2 (ссылка на pdb) я сравнила реальную вторичную структуру с предсказанными программами einverted
и RNAfold.
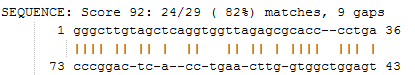
Рисунок 1. Инвертированные участки в последовательности тРНК из 1QU2, полученные с помощью программы einverted при запуске с параметрами gap penalty 2; minimum score threshold 20; match score 5; mismatch score -2).
Как можно заметить, данный способ недостаточно эффективен для предсказания вторичной структуры тРНК, так как найден только акцепторный стебель (нуклеотиды 1-7 и 67-73).
Такой результат может быть связан с тем, что эта программа не учитывает возможные некомплементарные пары оснований, которые в тРНК встречаются достаточно часто.
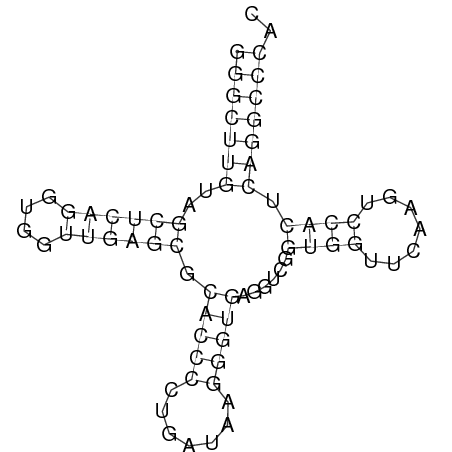
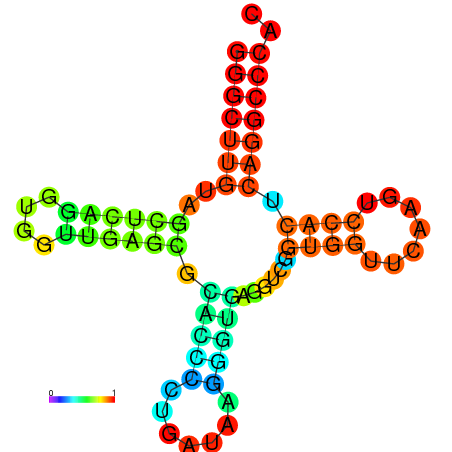
Рисунок 2 (слева). Вторичная структура тРНК из 1QU2, полученная с помощью алгоритма Зукера, реализованного программой RNAfold.
На рисунке 4 представлена вторичная структура тРНК с названиями стеблей и петель. Изображение взято из статьи О.О.Фаворовой (ссылка на pdf). 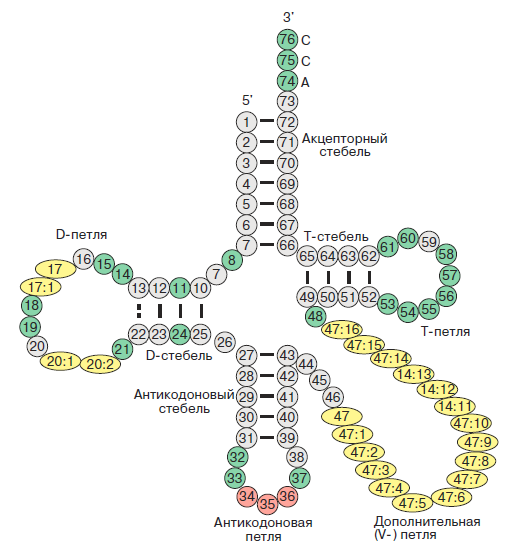
Рисунок 4. Вторичная структура тРНК. В таблице 1 произведено сравнение вторичных структур тРНК, полученных с помощью программ find_pair, einverted и RNAfold. Указано количество пар нуклеотидов в стеблях тРНК по результатам трех программ.
* - через символ "::" обозначено, между какими основаниями образуются водородные связи; номера нуклеотидов указаны от 5'-конца к 3'
Далее я работала с ДНК-белковым комплексом из PDB-структуры 1EFA (ссылка на pdb). Из белка я брала только цепи A и B.
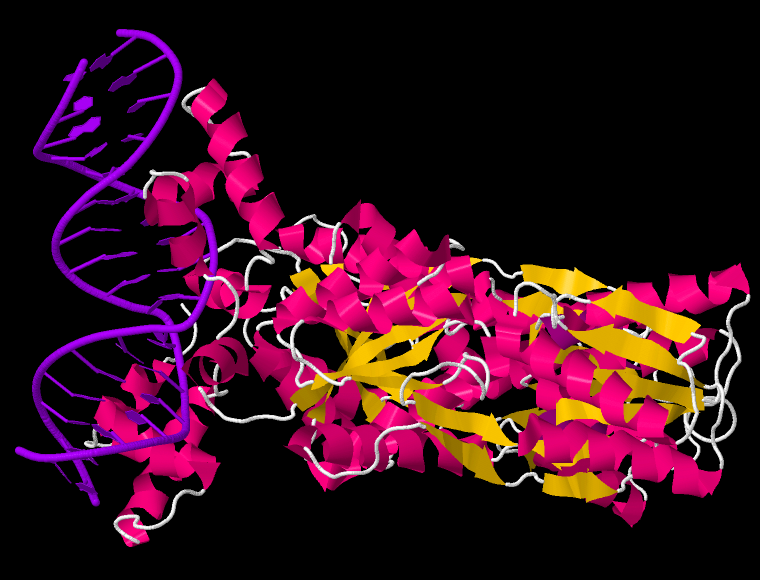
Рисунок 5. ДНК-белковый комплекс из PDB-структуры 1EFA. Представлены только A- и B-цепи белка. Изображение получено с помощью программы JMol, способ отображения cartoons, раскраска по структуре. 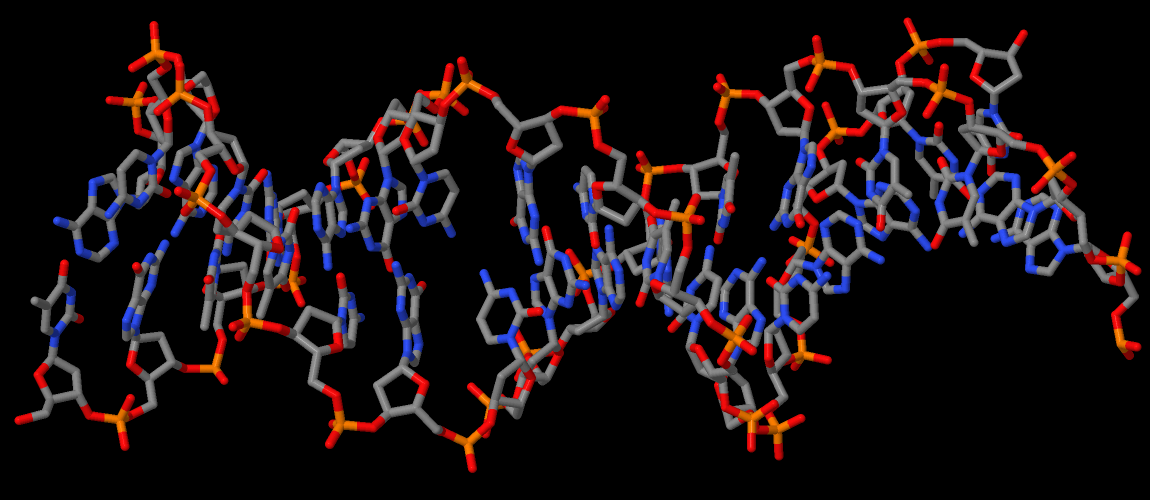
Рисунок 6. ДНК из комплекса 1EFA. Раскраска по cpk, способ отображения wireframe. 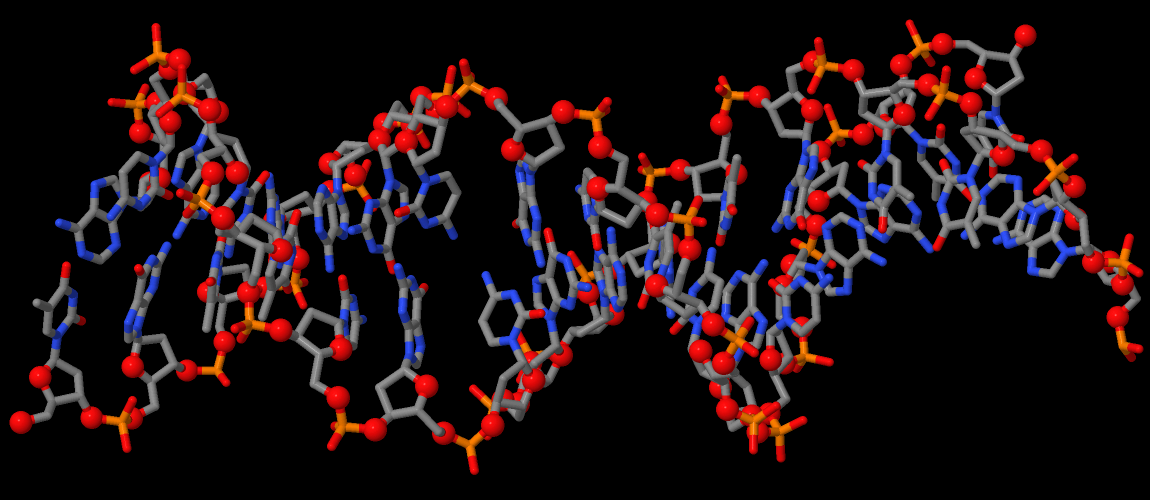
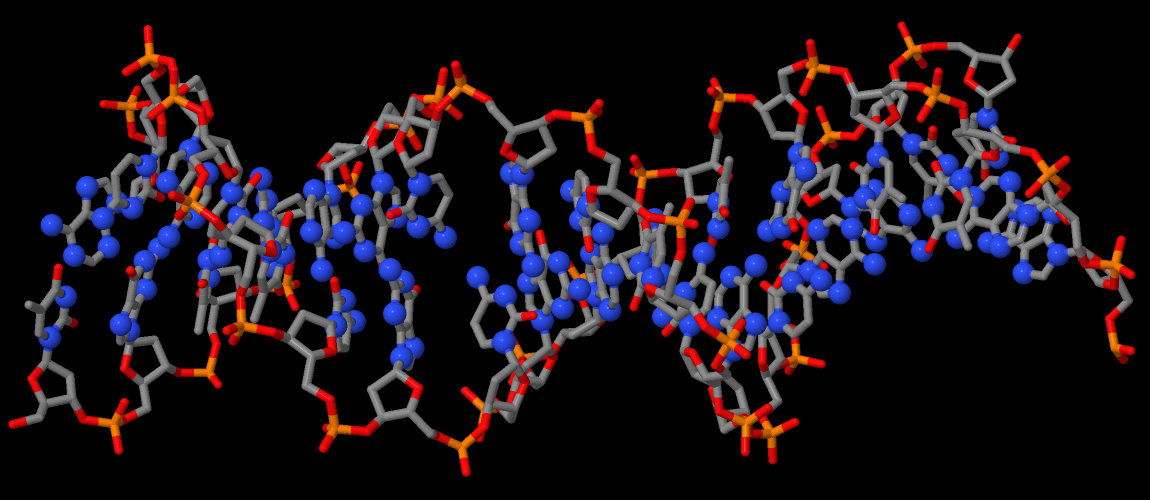
Рисунок 7 (сверху). ДНК из комплекса 1EFA. Шариками показаны атомы кислорода 2'-дезоксирибозы (set1).
В таблице 2 представлена информация о контактах разного типа (полярных и неполярных) между ДНК и белком в структуре 1EFA. Полярными атомами считаются атомы кислорода и азота, неполярными - атомы углерода, фосфора и серы. Полярный контакт - ситуация, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5Å. Неполярный контакт - пара неполярных атомов на расстоянии меньше 4.5Å.
С помощью программы nucplot была построена популярная схема ДНК-белковых контактов. Она представлена на рисунке 10. 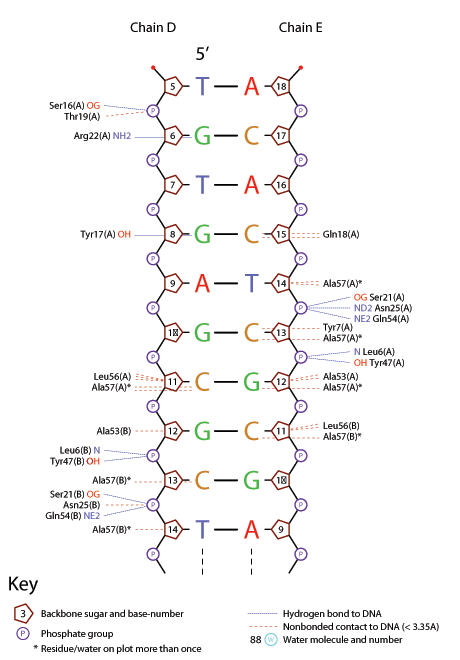
Рисунок 10. Схема ДНК-белковых контактов в структуре 1EFA для цепей белка A, B и для цепей ДНК D, E. Изображение получено с помощью программы nucplot. На рисунке 10 видно, что белок образует достаточно много контактов с сахаро-фосфатным остовом ДНК. Скорее всего, эти контакты нужны для того, чтобы белок удерживался на ДНК. Однако контактов с остатками азотистых оснований также достаточно много. Эти контакты отвечают за распознавание белком специфической последовательности нуклеиновой кислоты, что является важным в данном случае, так как в структуре 1EFA представлено взаимодействие лактозного репрессора с оператором. Также можно заметить, что взаимодействующие аминокислотные остатки одинаковы для A и B цепи, то есть эти цепи имеет одинаковую аминокислотную последовательность (для подтверждения fasta-файл со структурой 1EFA). В таблице 2 видно, что контактов с сахаро-фосфатным остовом в сумме 84, контактов с остатками азотистых оснований 50, то есть оба типа контактов важны для данного ДНК-белкового комплекса. При этом со стороны большой бороздки образуется больше контактов, чем со стороны малой, что вполне логично, так как большая бороздка глубже и, следовательно, удобнее для образования взаимодействий. Также можно сказать, что неполярные контакты встречаются чаще, чем полярные, но, скорее всего, именно полярные контакты отвечают за специфическое распознование. На рисунках 11-13 я постаралась продемонтрировать контакты некоторых аминокислотных остатков с ДНК. С помощью рисунка 10 я определила остаток с наибольшим числом контактов с ДНК - Ala57(A), который образует 2 контакта с цитозином в C11(D), 1 с гуанином в G12(E), 1 с цитозином в C13(E) и 2 с остатком дезоксирибозы в T14(E), в сумме 6 контактов. Такой же остаток другой цепи белка, Ala57(B), образует 3 контакта: 1 с цитозином C11(E), 1 с цитозином C13(D) и 1 с остатком дезоксирибозы T14(D). Однако все эти контакты обозначены как nonbonded, то есть у этих остатков просто есть атомы, находящиеся от ДНК на расстоянии меньше, чем 3.35Å. Поэтому я показала также остаток Arg22(A), образующий с гуанином в G6(D) водородную связь. Контакты ДНК с Ala57(A), Ala57(B) и Arg22(A) представлены на рисунках 11, 12 и 13 соответственно. 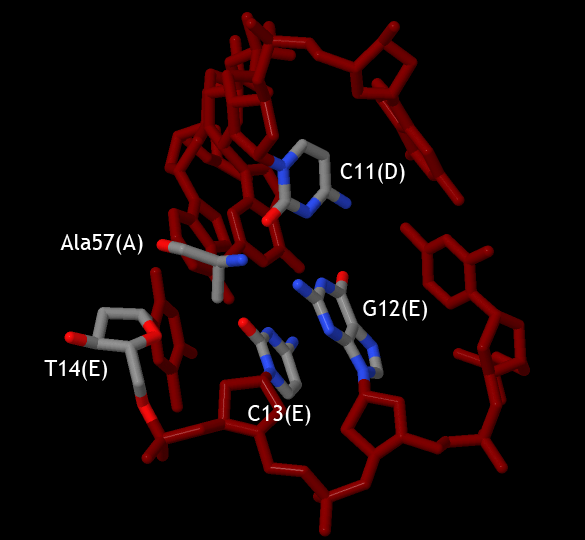
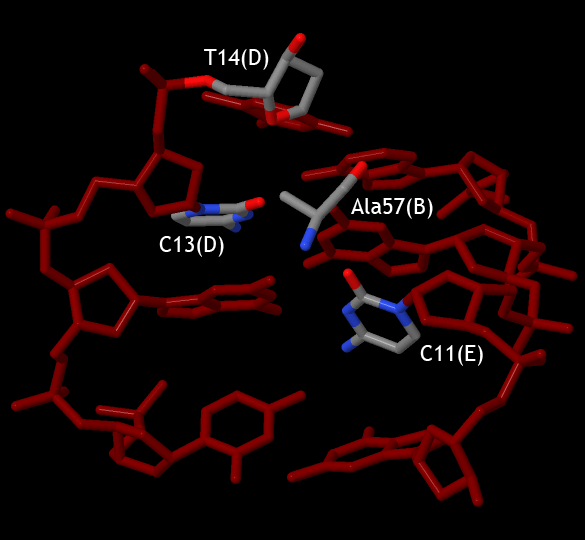
Рисунок 11 (слева). Контакты аминокислотного остатка Ala57 цепи A белка с ДНК в структуре 1EFA. Аминокислотный остаток и части ДНК, с которыми
он взаимодействует, обозначены темно-красным цветом и подписаны, остальные атомы раскрашены по cpk. На рисунке представлен участок ДНК 9-12:D и 11-14:E.
Изображение получено с помощью программы JMol, способ отображения wireframe.
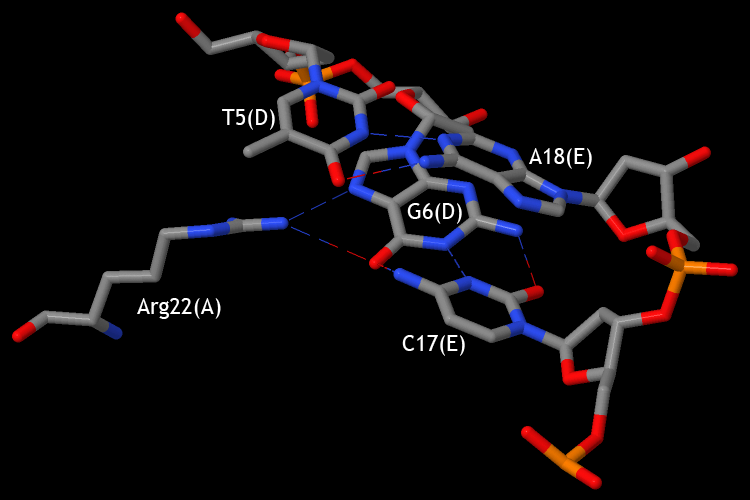
Рисунок 13. Контакты аминокислотного остатка Arg22 цепи A белка с ДНК в структуре 1EFA. На рисунке представлен участок ДНК 5-6:D и 17-18:E. Водородные связи обозначены пунктирными линиями. |
||||||||||||||||||||||||||||||||||||||||||||||||||||
|
© Наталия Кашко, 2015 |
|||||||||||||||||||||||||||||||||||||||||||||||||||||