|
|
|
|
Для аннотации генов прокариот я выбрала архею Methanothermobacter thermautotrophicus (рисунок 1). Это метаногенная архея, получающая энергию, переводя водород и углекислый газ в метан, является термофильным организмом с оптимальной температурой роста около 65-70°C. Я взяла хромосому NC_000916.1, представляющую полный геном Methanothermobacter thermautotrophicus str. Delta H. Для этого штамма характерными условиями обитания являются осадки сточных вод, где он был впервые обнаружен в 1971 в штате Иллинойс, США. Его геном был полностью отсеквенирован и имеет длину 1,751,377 п.о. 
Рисунок 1. Methanothermobacter thermautotrophicus, домен Archaea, класс Methanobacteria, семейство Methanobacteriaceae. Полная таксономия доступна на сайте ncbi.
Исходная таблица аннотаций генов из базы данных GenBank (получена из NCBI Gene) доступна по ссылкам: в формате excel и в текстовом формате.
Всего генов аннотировано 1858, из них 38 кодируют тРНК и 7 - рРНК.
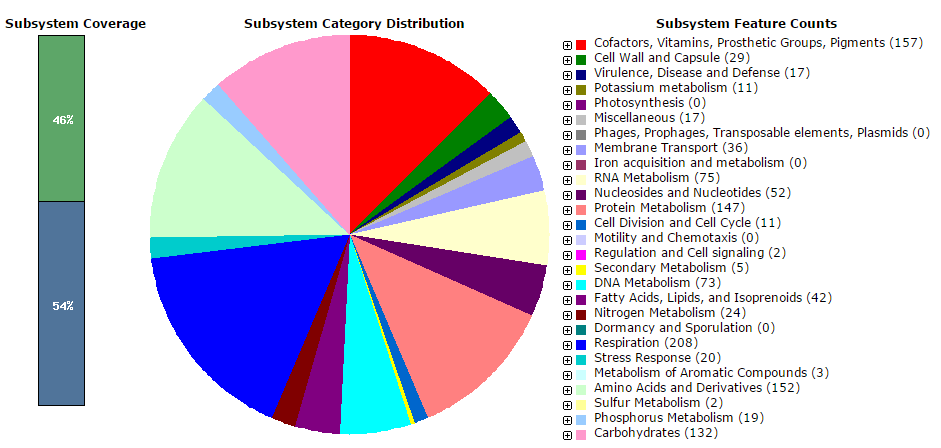
Рисунок 2. Диаграмма распределения генов археи Methanothermobacter thermautotrophicus по функциональным группам. Изображение получено с помощью сервера RAST.
Полученные аннотации я сравнила с уже имеющимися в записи GenBank. Результаты сравнения представлены в таблице.
Для некоторых белков с несовпадающими аннотациями я проверила их функции с помощью blast (blastx). На рисунке 3 представленны выбранные белки. 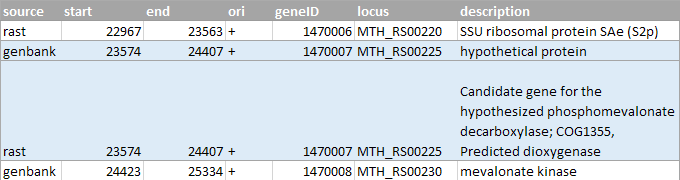
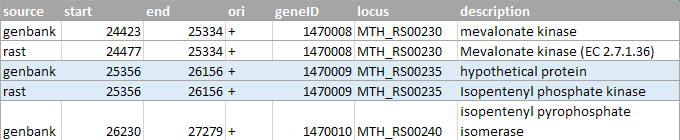
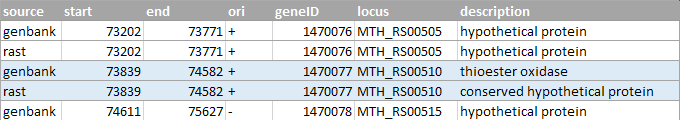
Рисунок 3a (сверху). Первая пара белков (выделена голубым).
Результаты поиска первой последовательности (кодирующей первый белок) в blast показали, что данный белок принадлежит семейству MEMO1 и суперсемейству экстрадиол дезоксигеназ. Белки семейства MEMO1
являются эффекторами ErbB2 рецепторной тирозинкиназы, участвующей в регуляции подвижности клетки. В целом, описание более соответствует аннотации RAST.
Таким образом, можно заключить, что RAST является хорошим сервером для аннотации генов прокариот, так как он позволяет обнаружить до 90% известных генов, при этом в некоторых случаях указывает более точные описания функций их продуктов. |
|
© Наталия Кашко, 2015 |