|
|
|
|
В данном практикуме мы анализировали результаты секвенирования по Сангеру разных последовательностей. Капиллярный секвенатор выдает
хроматограммы для прямой и обратной последовательностей в формате .ab1
В прямой последовательности первые 50 нуклеотидов прочитать сложно из-за высокой интенсивности шума и нечеткости сигналов. Однако для обратной
последовательности, из которой была получена комплементарная ей цепь с помощью функции Reverse+Complement, хроматограмма имеет нормальное
качество, начиная с третьего неклеотида. Первый читаемый нуклеотид в прямой цепи соотвествует 68 нуклеотиду обратной цепи (номера указаны для уже
отредактированных цепей после удаления нечитаемых нуклеотидов в начале и конце последовательностей). По прямой цепи качество хроматограммы
ухудшается к концу последовательности, однако трудности с определением нуклеотидов возникают только за несколько пиков до конца. По обратной цепи,
напротив, последние 40 нуклеотидов не читаемы. Последний нуклеотид обратной цепи (343) соотвествует 276 нуклеотиду прямой цепи.
Я отредактировала прямую и обратную последовательность, выданную программой на основании хроматограммы, проверяя каждый нуклеотид и сравнивая участки этих последовательностей. В результате было получено 2 нуклеотидных последовательности (прямой и обратной цепей). Маленькими буквами отмечены нуклеотиды, которые я изменила при проверке хроматограммы. Для этих последовательностей было построено выравнивание (рисунок 1), проект с выравниванием в формате .jvp доступен по ссылке. На основании полученных данных можно сказать, что исходная ДНК имеет следующую последовательность WS3004.fasta длиной 404 п.о. 
Рисунок 1. Выравнивание отредактированных последовательностей прямой и комплементарной к обратной цепей. Маленькими буквами отмечены измененные нуклеотиды. Изображение получено с помощью программы Jalview, раскраска по нуклеотидам. Далее на странице представлены проблемные участки хроматограмм, примеры хороших участков, а также нечитаемые хроматограммы для других последовательностей. На рисунке 2а представлен участок прямой цепи с очень высоким уровнем шума. Скорее всего, это пятно краски. На рисунке 2b показан тот же участок обратной цепи. Видно, что сама программа в некоторых случаях справилась с решением этой проблемы. Так как на второй цепи в окружении этого участка не наблюдается никаких серьезных загрязнений или отклонений, можно с достаточной уверенностью восстановить правильную последовательность. Символы N на позициях 43 и 47 были заменены на С, вместо двух символов N на позициях 40 и 41 были записаны 3 символа C, в соотвествии с последовательностью обратной цепи. 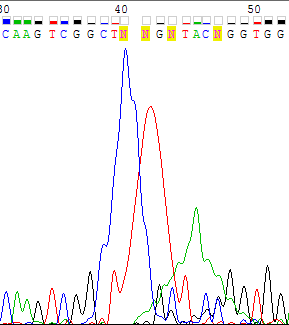
Рисунок 2а. Участок прямой цепи с 30 по 52 нуклеотиды. Изображение из программы Chromas (Lite). 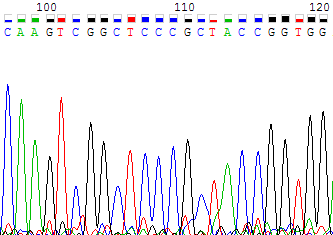
Рисунок 2b. Участок обратной цепи с 97 по 120 нуклеотиды, соотвествующий поблемному участку прямой цепи. На рисунке 3a показано сразу несколько проблем: по обратной цепи нуклеотиды 206 и 207 программа не определила (N), нуклеотид 200 однозначно указать сложно. Позиции 206 и 207 не вызывают никаких затруднений, потому что там отчетливо видны сигналы, соответствующие C и G. На 200 нуклеотиде шум по интенсивности равен сигналу, поэтому точно восстановить правильную последовательность можно только по второй цепи. Соответствующий нуклеотид прямой цепи - 133, С. Замененные нуклеотиды 206 и 207 также совпадают в прямой цепи (позиции 139 и 140). В этом месте в прямой цепи сигналы отчетливые и не вызывающие сомнений (рисунок 3b). 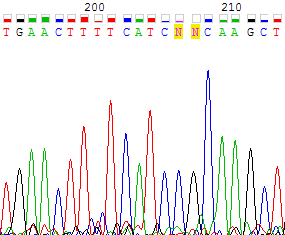
Рисунок 3а. Участок обратной цепи с 193 по 213 нуклеотиды. 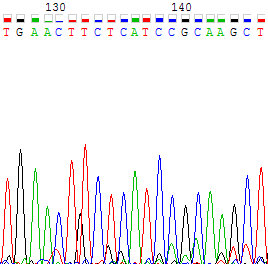
Рисунок 3b. Участок прямой цепи с 126 по 146 нуклеотиды, соотвествующий поблемному участку обратной цепи. На рисунке 4a представлен участок обратной цепи с 316 по 340 нуклеотиды. Видно, что сигналы становятся менее отчетливыми, сливаются и смещаются, усиливается интенсивность шума, так как это уже конец цепи, анализ результатов секвенирования затруднен. Однако по прямой цепи соответствующий участок хроматограммы имеет высокое качество (рисунок 4b), поэтому по второй цепи можно восстановить последовательность. Например, неопределенные нуклеотиды 322, 323, 325, 330 обратной цепи были заменены на T, C, C, A соотвественно (по 259, 260, 262, 266 нуклеотидам прямой цепи). На прямой цепи также есть один неопределенный нуклеотид 265, его я заменила на G, так как пик сигнала гуанина чуть выше шума, а на обратной цепи виден отчетливый сигнал (329). 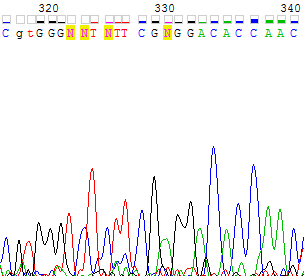
Рисунок 4а. Участок обратной цепи с 316 по 340 нуклеотиды. 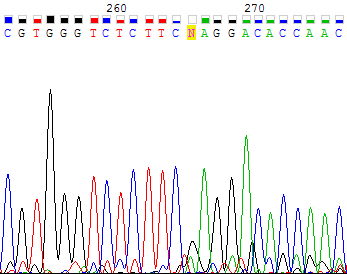
Рисунок 4b. Участок прямой цепи с 252 по 276 нуклеотиды, соотвествующий поблемному участку обратной цепи. На рисунке 5a показан еще один участок обратной цепи с 242 по 267 нуклеотиды, где из-за высокой интенсивности шума невозможно определить нуклеотиды 247-258. В соответствующем участке второй цепи хорошие и отчетливые пики (рисунок 5b, позиции 180-191). По прямой цепи можно восстановить последовательность GG(A)CTT(C)AAGAC, в скобках указаны нуклеотиды, определенные программой в обратной цепи. В принципе, на обратной цепи видны все пики, соответствующие данным нуклеотидам. 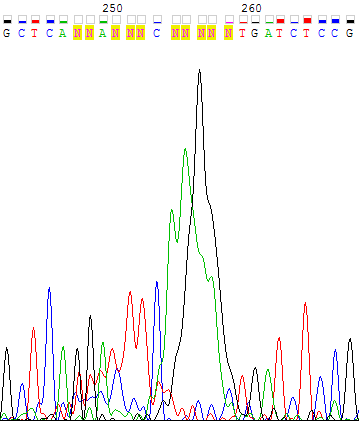
Рисунок 5а. Участок обратной цепи с 242 по 267 нуклеотиды. 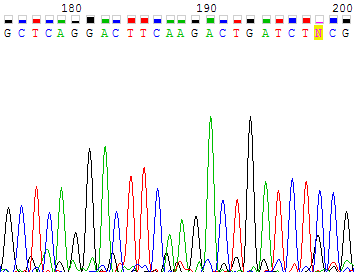
Рисунок 5b. Участок прямой цепи с 175 по 200 нуклеотиды, соотвествующий поблемному участку обратной цепи. На рисунке 6a - участок прямой цепи с 77 по 97 нуклеотиды. На позициях 82-92 уровнем шума выше, чем в среднем по цепи, поэтому результаты, выданные программой, нуждаются в проверке. Однако по такому же участку обратной цепи (рисунок 6b) видно, что последовательности, определенные программой, совпадают для обеих цепей. Таким образом, программа сама справилась с этой проблемой. 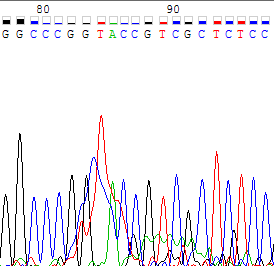
Рисунок 6а. Участок прямой цепи с 77 по 97 нуклеотиды. 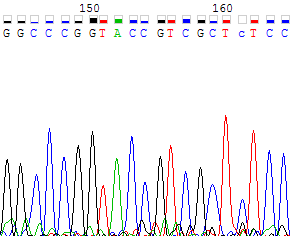
Рисунок 6b. Участок обратной цепи с 144 по 164 нуклеотиды, соотвествующий поблемному участку прямой цепи. На рисунках 7a и 7b представлены участки хроматограмм прямой и обратной цепи соответственно. На этих участках сигналы отчетливые, последовательность нуклеотидов определяется однозначно. 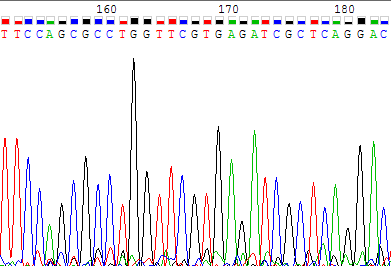
Рисунок 7а. Участок прямой цепи с 151 по 183 нуклеотиды. 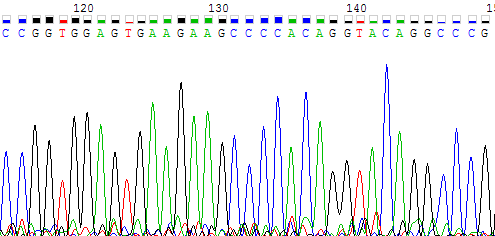
Рисунок 7b. Участок обратной цепи с 114 по 149 нуклеотиды. На рисунках 8 и 9 представлены примеры нечитаемых хроматограмм. Первую из них читать невозможно, так как уровень шума часто совпадает с уровнями сигналов и иногда имеет ту же интенсивность, что и обычный чистый сигнал. Таким образом, нельзя определить, является ли самый высокий пик сигналом правильного нуклеотида или шумом. Во второй хроматограмме заметен резкий скачок интенсивности сигналов. Неясно, чем мог быть вызван такой переход и как он повлиял на правильность полученных данных. При этом пики сигналов часто сливаются в один удлиненный пик, что также вносит нечеткость в определение последовательности. 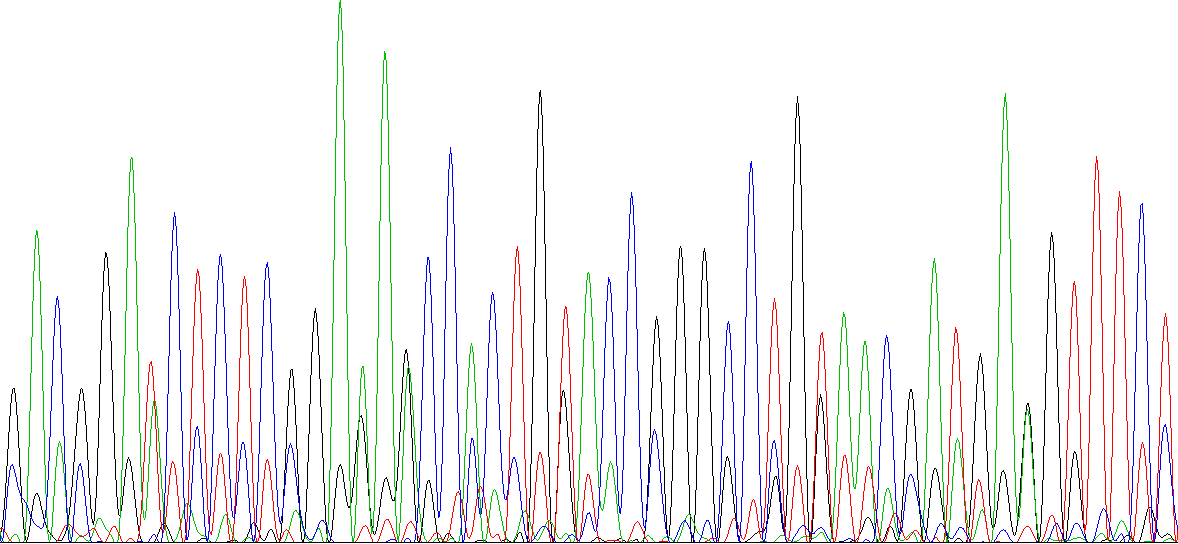
Рисунок 8. Пример нечитаемой хроматограммы из файла WSWS2931.ab1 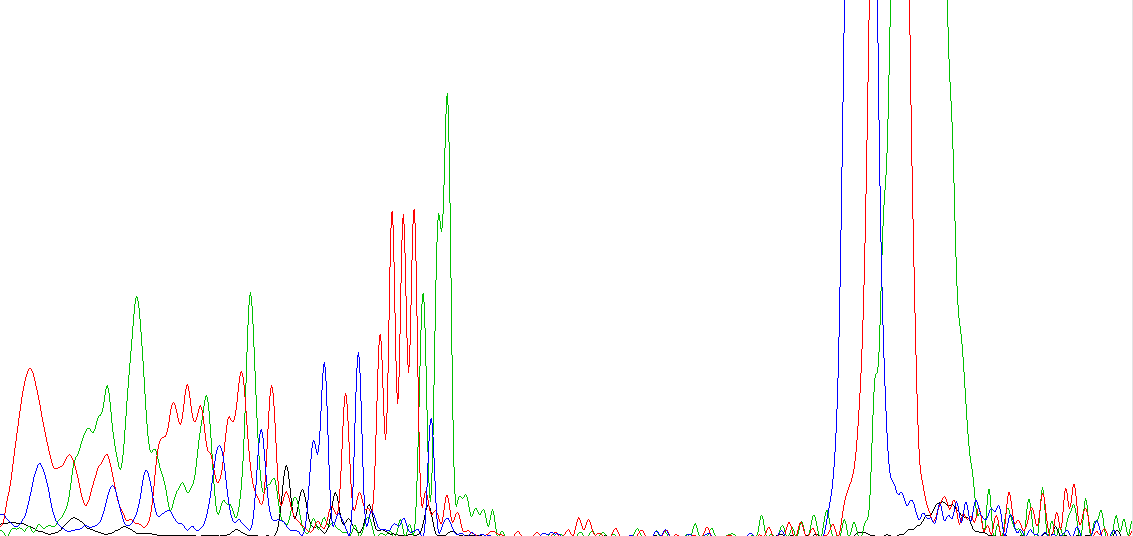
Рисунок 9. Пример нечитаемой хроматограммы из файла WS2943.ab1 |
|
© Наталия Кашко, 2015 |