|
|||||||||||||||||||||||||||||||||||||||||||
|
|
Предсказание генов с помощью AUGUSTUS
Из сборки генома X5 я выбрала один скэффолд (scaffold-266), длина которого составляет 47841 п.о. Ссылка на последовательность этого скэффолда в формате fasta:
scaffold266.fasta. Для этого скэффолда с помощью сервера AUGUSTUS были
предсказаны входящие в его состав гены.
В файле genes.xlsx представлена информация о найденных генах, их экзон-интронной структуре и наличии достоверных гомлогов их продуктов. Более подробную информацию о гомологах можно посмотреть в таблице 1. Для пяти продуктов генов, у которых были найдены гомологи, я проверила, правильно ли предсказаны кодирующие их гены на основании генов гомологичных белков. Поиск гомологов осуществлялся алгоритмом blastp.
Шестой ген кодирует белок, который имеет относительно слабое сходство с найденным гомологом. Возможно, это вызвано тем, что найденные экзоны не соответствуют реальным экзонам гена. Выравнивание, представленное на рисунке 1, начинается с 71 остатка в subject и с 54 остатка в query, при этом высокое сходство имеют только отдельные участки. Ген Helicosporidium sp. имеет 5 экзонов, в то время как ген исследуемого организма - только 2. Скорее всего, AUGUSTUS не нашел некоторые экзоны в начале гена. При этом найденные экзоны могут иметь немного неправильные границы, так как середина вырванивания имеет меньшее сходство, чем крайние регионы. Однако эти отличия могут быть вызваны реальным расхождением в последовательностях белков двух разных организмов. 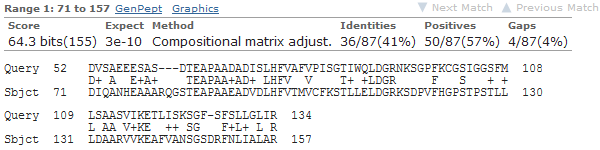
Рисунок 1. Выравнивание продукта шестого гена с лучшей находкой blastp из организма Helicosporidium sp..
Восьмой ген кодирует субъединицу белка АТФазы, высококонсервативного фермента эукариот. Большое сходство последовательностей говорит о том, что найденные экзоны, скорее всего, правильные. Однако
гены, кодирующие эту субъединицу у Mortierella verticillata и Allomyces macrogynus (вторая по счету находка, E-value составляет 5e-55, query cover также 94%), имеют 8 и 5
экзонов соответственно. Гомологичные белки имеют гораздо большую длину (примерно 500 а.о.), чем искомый (139 а.о.). Можно предположить, что AUGUSTUS нашел только 2 первых экзона этого гена,
но сказать, сколько именно должно быть экзонов, нельзя.
Двенадцатый ген кодирует белок с неизвестными функциями. При поиске в blastp последовательности продукта этого гена было найдено два участка сходства с лучшей находкой. Скорее всего, эти находки соответствуют двум экзонам двенадцатого гена. Так как сходство достаточно высокое, эти экзоны можно считать правильными, несмотря на то, что в гене Stylonychia lemnae экзоны не указаны. Однако находка не покрывает весь гомологичный белок, поэтому можно предположить, что должен быть еще экзон до предсказанного гена и еще один между имеющимися экзонами. Однако длина интрона составляет всего 666 п.о. Возможно, этот ген предсказан ошибочно или белок имеет другого, более близкого гомолога. Сравнение аннотаций Refseq и AUGUSTUS гена DGCR8
У человека ген DGCR8 кодирует белок DGCR8 (DiGeorge syndrome chromosomal/critical region 8), являющийся компонентом микропроцессора. Функции данного комплекса связаны с процессингом микроРНК. Белок DGCR8
связывается с ферментом Drosha, являющимся РНКазой III, и образует микропроцессор, который разрезает при-микроРНК - длинную двухцепочечную РНК, транскрибирующуюся в клетке. После разрезания образуются короткие
двухцепочечные фрагменты с концевой шпилькой, называемые пре-микроРНК. Далее эти фрагменты преобразуются в микроРНК с помощью фермента Dicer. В целом, микроРНК осуществляют в клетке важные (хотя и не до
конца изученные) регуляторные функции.

Рисунок 2. Окно геномного браузера, представляющее экзон-интронную структуру гена DGCR8 по аннотациям Refseq (сверху, синий) и AUGUSTUS (снизу, зеленый). Использована последняя сборка генома человека hg38. Координаты отображаемого участка chr22:20,080,232-20,111,877. Изображение получено с помощью геномного браузера UCSC. Подробная информация об экзон-интронной структуре данного гена представлена в таблице dgcr8.xlsx. Сравнение аннотаций показало, что AUGUSTUS предсказал экзон-интронную структуру достаточно точно. Неправильно определены границы гена (ошибка меньше, чем на 100 п.о. с каждой стороны) и, следовательно, начало первого и конец последнего экзонов. Также в аннотации Refseq указан один экзон, который может вырезаться или не вырезаться при альтернативном сплайсинге (экзон № 8). AUGUSTUS не определил этот экзон, поэтому в аннотации AUGUSTUS экзонов всего 13, а в аннотации Refseq - 14. |
||||||||||||||||||||||||||||||||||||||||||
|
© Наталия Кашко, 2015 |
|||||||||||||||||||||||||||||||||||||||||||