|
|
|
|
Выравнивание белковых последовательностей
Для выбранных бактерий было построено "реальное" дерево, исходя из последовательностей белков пептидил-тРНК гидролаз (мнемоника PTH).
Последовательности использованных белков в формате fasta: pth.fasta.

Рисунок 1. Выравнивание последовательностей пептидил-тРНК гидролаз из выбранных видов бактерий. Раскраска ClustalX. На следующих рисунках показаны блоки, в которых заметна гомологичность всех последовательностей. Видимо, это активные центры белков. Однако уже в этих консервативных участках наблюдаются некоторые отклонения, отраженные на дереве. Например, на первом рисунке видны отличия белков из HAEIN и PASMU от остальных. На нижнем рисунке в первом блоке - различия между классом Gammaproteobacteria (нижние пять последовательностей) и остальными, а также замены, характерные для класса Betaproteobacteria (вторая и третья строки). 

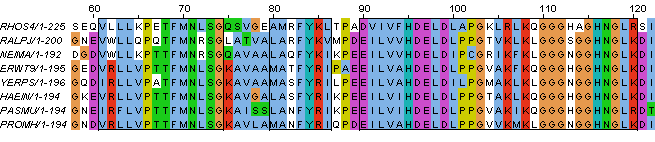
Рисунок 2. Участки гомологии всех исследуемых последовательностей. Блоки выделены черной рамкой. Заметно, что первая последовательность, принадлежащая бактерии из класса Alphaproteobacteria, сильнее всего отличается от остальных, хотя бы потому, что она длиннее. В участках между блоками ее аминокислотный набор также не похож на другие последовательности. Некоторые отличия показаны на рисунке 3. 
Рисунок 3. Отличия последовательности RHOS4 от остальных: длина и вставка. Далее показаны отличия, характерные для других классов (Betaproteobacteria и Gammaproteobacteria), а также для семейства Pasteurellaceae, в которое входят последовательности HAEIN и PASMU. Отмеченные замены, возможно, отражают пути эволюции данных групп. 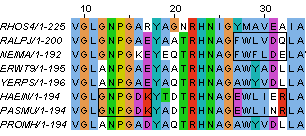
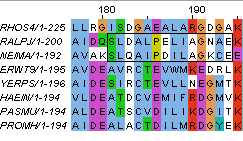
Рисунок 4. Отличия класса Betaproteobacteria (последовательности RALPJ и NEIMA) от остальных: F27, S181, L182, P186, A190, E194. Также показаны отличия семейства Pasteurellaceae (последовательности HAEIN и PASMU): D16, K17, E27, L29, R32. 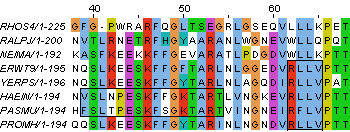
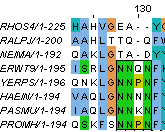
Рисунок 5. Отличия класса Gammaproteobacteria (нижние 5 последовательностей) от остальных: R62, V65, N128, N129, N131. Также показаны отличия семейства Pasteurellaceae (последовательности HAEIN и PASMU): P43, K50, T54. Алгоритм Neighbour-Joining
JalView предлагает три вида алгоритма Neighbour-Joining: с использованием процента идентичных позиций (% Identity), PAM 250 или BLOSUM62.
Также один алгоритм реализован в программе MEGA.
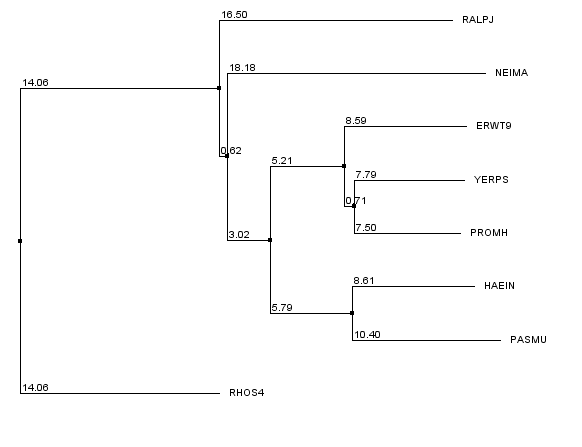
Рисунок 6. Филогенетическое дерево, построенное с использованием алгоритма Neighbour-Joining Using % Identity. 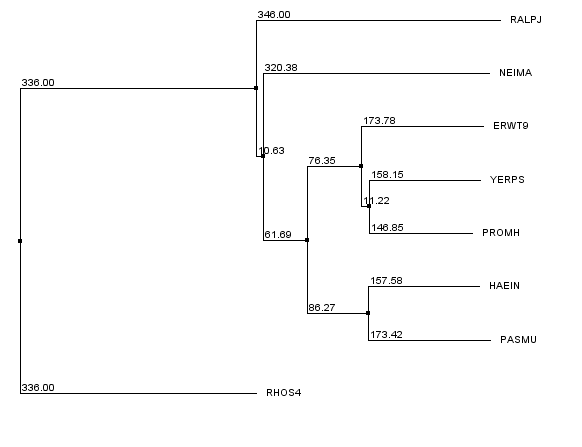
Рисунок 7. Филогенетическое дерево, построенное с использованием алгоритма Neighbour-Joining Using BLOSUM62. На рисунке 8 представлено дерево, построенное программой MEGA. Оно отличается от предыдущих деревьев и является наиболее приближенным к реальному дереву, полученному исходя из дерева видов. Отличия заключаются во взаимном расположении бактерий внутри семейства Enterobacteriaceae. По дереву видов ERWT9 и YERPS составляют одну кладу, а PROMH находится отдельно. По деревьям из JalView кладу составляют YERPS и PROMH. Однако ни одна из реализаций этого алгоритма не привела к абсолютно правильному дереву. Различия наблюдаются в классе Betaproteobacteria, так как последовательности NEIMA и RALPJ должны быть объединены и располагаться отдельно от остальных классов. 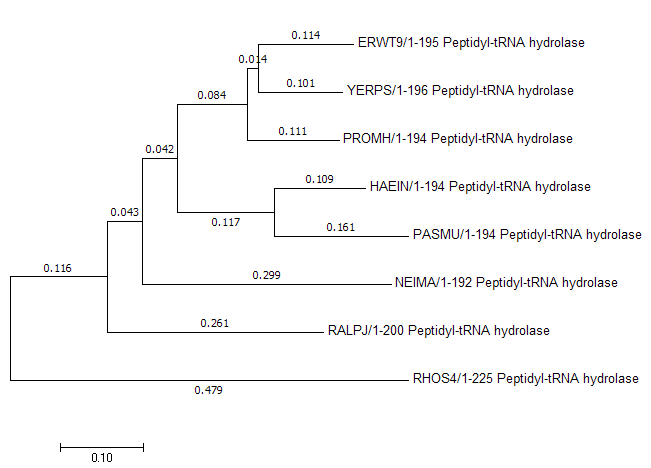
Рисунок 8. Филогенетическое дерево, построенное с использованием алгоритма Neighbour-Joining в программе MEGA. |
|
© Наталия Кашко, 2016 |