|
|||||||||||||||||||||||||||||||
|
|
Построение дерева по нуклеотидным последовательностям
Для построения дерева были взяты последовательности 16s рРНК перечисленных в таблице 1 бактерий. Они были получены из
базы полных геномов NCBI.
Последовательности генов рРНК и тРНК записаны в файлах с расширением .frn. В большинстве случаев такой
файл был один, за исключением RALPJ и RHOS4. У этих двух видов в базе были представлены две хромосомы, для каждой из
которых имелся свой файл с последовательностями РНК.
С помощью Muscle было построено выравнивание полученных последовательностей (рисунок 1). Выравнивание в fasta-формате: alignment.fasta. 
Рисунок 1. Выравнивание последовательностей 16s рРНК из бактерий, описанных в таблице 1. Построено в JalView с помощью Muscle. Раскраска Nucleotide. С помощью программы MEGA было построено филогенетическое дерево последовательностей по выравниванию. Использованные методы: Neighbor-Joining, Minimum evolution и Maximum likelihood. Первые два метода выдают деревья с одинаковой топологией (рисунок 2), последним же было построено дерево (рисунок 3), которое отличается взаимным расположением видов внутри семейства Enterobacteriaceae (YERPS, PROMH и ERWT9). Если сравнивать с исходным деревом (рисунок 4), полученным из общего дерева протеобактерий, то правильными оказываются деревья, построенные методами Neighbor-Joining и Minimum evolution. 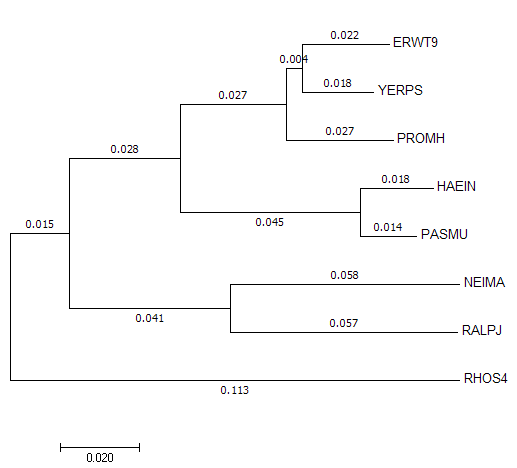
Рисунок 2. Филогенетическое дерево, построенное по последовательностям 16s рРНК алгоритмом Neighbor-Joining. 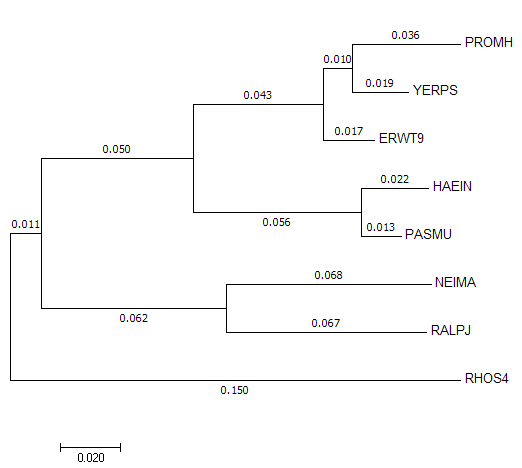
Рисунок 3. Филогенетическое дерево, построенное по последовательностям 16s рРНК алгоритмом Maximum Likelihood. 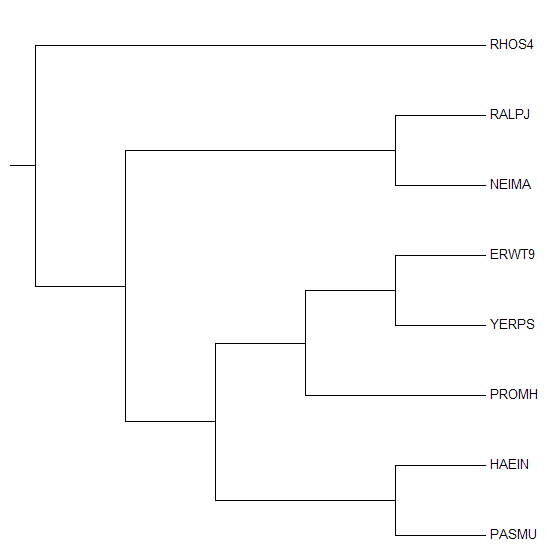
Рисунок 4. Филогенетическое дерево, построенное по общему дереву протеобактерий. Я использовала все три метода, так как полученные результаты были прямо противоположными к тем, которые наблюдались при построении деревьев по последовательностям белков. В этом случае правильным было дерево, построенное алгоритмом Maximum likelihood (рисунок 5), а деревья, полученные методами Neighbor-Joining и Minimum evolution (рисунок 6), имели одинаковую топологию и отличались от исходного взаимным расположением RHSO4, RALPJ и NEIMA. 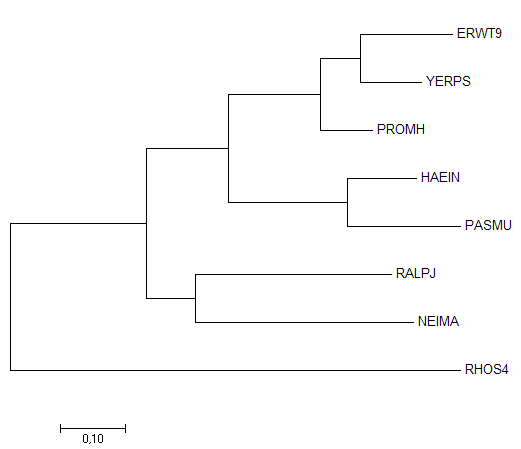
Рисунок 5. Филогенетическое дерево, построенное по последовательностям пептидил-тРНК гидролаз алгоритмом Neighbor-Joining. 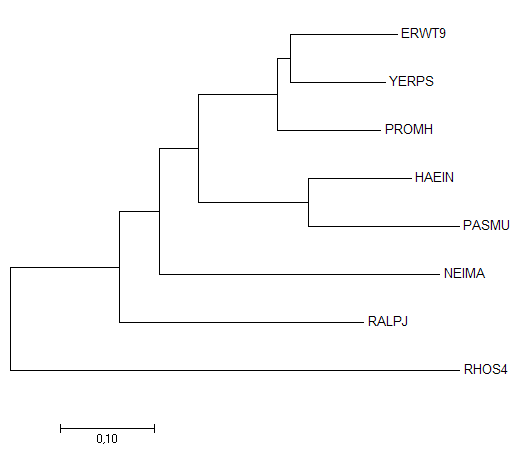
Рисунок 6. Филогенетическое дерево, построенное по последовательностям пептидил-тРНК гидролаз алгоритмом Maximum Likelihood. В целом, существенных различий в качестве построения деревьев по белковым или по нуклеотидным последовательностям в моем случае не наблюдалось. Разные алгоритмы в разных ситуациях оказывались наиболее подходящими и верными. Построение и анализ дерева, содержащего паралоги
В протеомах выбранных бактерий были найдены достоверные гомологи белка CLPX_ECOLI.
С порогом E-value 0.001 было получено 34 находки (выдача blast), однако реально
наиболее сходными являются первые 23 находки (с E-value ниже 1e-20). Они соответствуют белкам CLPX и HSLU. Представители этих
семейств белков есть во всех рассматриваемых бактериях и имеют в них одинаковые функции. Функции остальных найденных белков
сильно различаются.
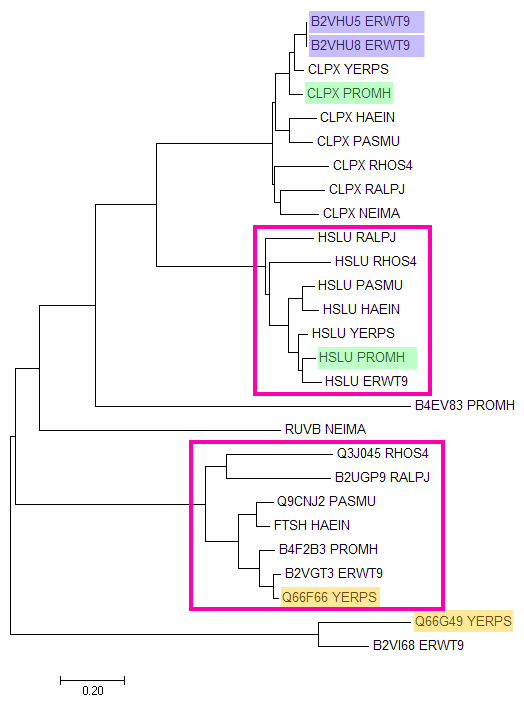
Рисунок 7. Филогенетическое дерево гомологичных белков, построенное алгоритмом Neighbor-Joining. Голубым, зеленым и оранжевым отмечены пары паралогов, розовыми рамками выделены группы попарно ортологичных белков. Паралогами называются два гомологичных белка из одного организма. На рисунке 7 некоторые пары паралогов отмечены голубым, зеленым и оранжевым. Ортологами называются два гомологичных белка, если они из разных организмов и разделение их общего предка на линии, ведущей к ним, произошло в результате видообразования. На рисунке 7 некоторые группы попарно ортологичных белков выделены розовым. На дереве можно отметить следующие примеры эволюционных событий: дупликация гена (например, B2VHU5 и B2VHU8 из ERWT9, отмечено голубым) и разделение путей эволюции белков в результате видообразования (например, белки RUVB_NEIMA, Q66G49_YERPS и B2VI68_ERWT9 являются гомологичными, однако первый - хеликаза, участвующая в расплетании структур Холлидея, а два других - магниевые хелатазы). |
||||||||||||||||||||||||||||||
|
© Наталия Кашко, 2016 |
|||||||||||||||||||||||||||||||