|
|
|
|
Работа с KEGG ORTHOLOGYДля сравнения определенных в базе данных KEGG ORTHOLOGY ортологических рядов белков, катализирующих одну и ту же реакцию, были выбраны два ряда: K01655 и K10977. Данные ферменты катализируют одну из реакций, относящихся к метаболическому пути биосинтеза лизина - превращение 2-оксоглутарата в цитрат. Реакция отмечена на рисунке 1 (желтым цветом). 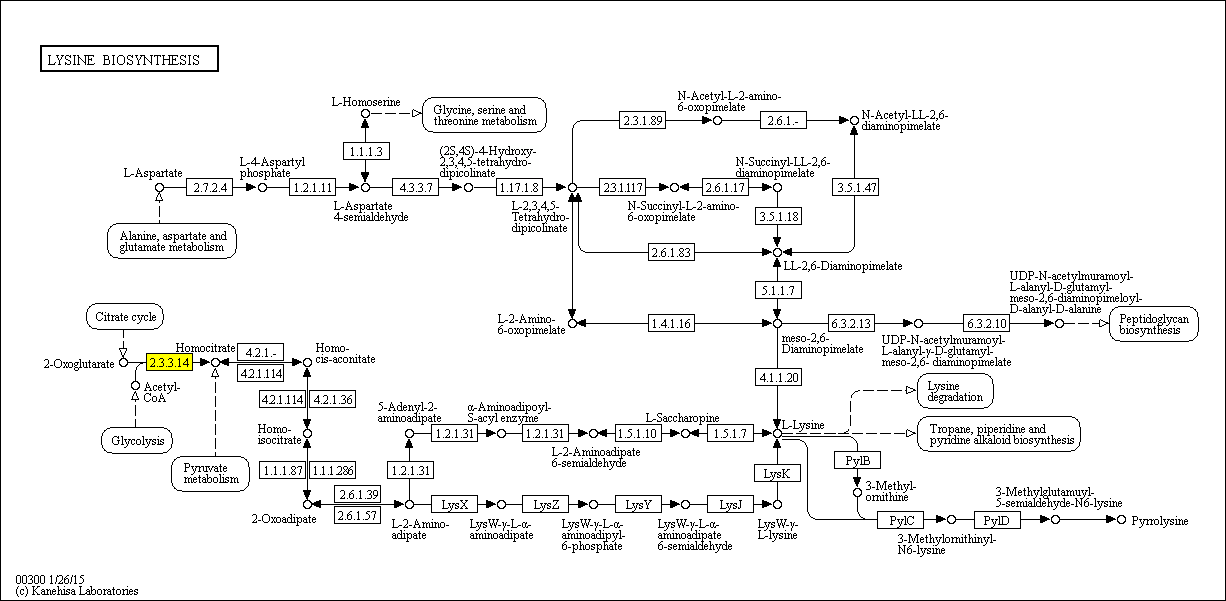
Рисунок 1. Метаболическая карта биосинтеза лизина, полученная из базы данных KEGG. Желтым выделена реакция, для которой изучалась гомологичность белков из разных ортологических рядов. Из базы данных UniProt были получены последовательности белков из приведенных ортологических рядов. Последовательности в формате fasta: K01655 (159 белков) и K10977 (70 белков). К идентификаторам белков были добавлены идентификаторы их ортологических рядов. Затем белки были выровнены с помощью программы Muscle (файл с выравниванием в формате fasta). На выравнивание можно посмотреть в проекте JalView. Гомологичность белков в выравнивании
На основании построенного выравнивания можно сделать вывод, что белки из данных ортологических рядов гомологичны между собой (как внутри
одного ряда, так и из разных рядов). Это можно подтвердить тем, что по всему выравниванию есть достаточно много консервативных колонок,
расположенных не поодиночке, а в сходном окружении. При этом консервативные колонки достаточно часто состоят из заряженных аминокислот,
которые могут быть важны для функционирования белка.
Проверка выравнивания
В выравнивании часто встречаются белки, которые относительно плохо выровнены с остальными белками. Однако говорить о том, что эти белки
относятся к отдельному ортологическому ряду, нельзя, так как в них также есть остатки, входящие в состав консервативных колонок. Отличия
же наблюдаются вне блоков. При этом можно выделить отдельные группы (по 2 и более белков), основываясь на сходствах в тех участках,
которые не совпадают для всех белков (аминокислотный состав в них варьирует). Эти различия используются для построения дерева.
В проекте JalView можно проследить за моими действиями в ходе выполнения данной части работы. Первое окно sequences - исходные последовательности белков. Второе окно alignment - выравнивание этих белков с помощью Muscle (без изменений). Третье окно short-sequences - удалены короткие последовательности (это выравнивание использовалось для построения дерева). Четвертое окно filtred приведено для удобства рассмотрения. В нем удалены короткие последовательности, а также полностью гэповые колонки и несовпадающие участки в начале и в конце белков. Построение дереваНа основе полученного выравнивания было построенно дерево. Для этого использовалась программа MEGA, алгоритм Neighbour-Joining со 100 бутстрэп-репликами. Полученное дерево представлено на рисунке 2. 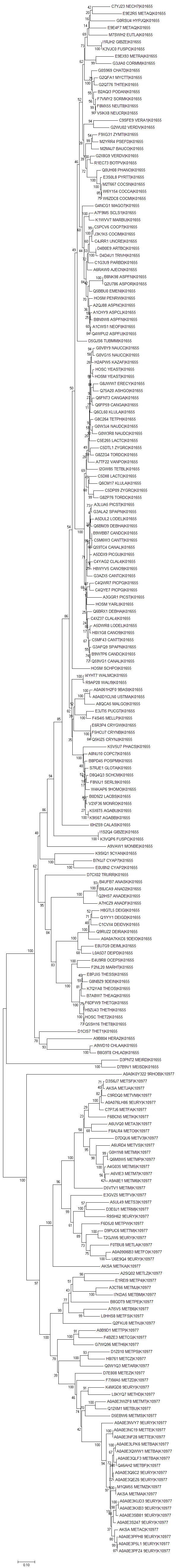
Рисунок 2. Филогенетическое дерево ферментов из двух ортологических рядов, катализирующих одну и ту же реакцию. Построено алгоритмом Neighbour-Joining со 100 бутстрэп-репликами с помощью программы MEGA.
Как видно из рисунка 2, дерево распадается на клады, соответствующие ортологическим рядам. При этом ветви, отделяющие данные клады от
остального дерева, достоверны (их поддержка на основании бутстрэп-анализа равна 100%).
|
|
© Наталия Кашко, 2016 |