|
|
|
|
Анализ множественного выравнивания трансмембранных белковДля изучения структуры трансмембранных белков был использован белок 4QO2 - ромбоидная мембранная протеаза GlpG. Эта сериновая протеаза разрезает трансмембранные белки по их внутримембранным участкам. На рисунках 1, 2 и 3 представлены ее изображение, структура и расположение в мембране соответственно. 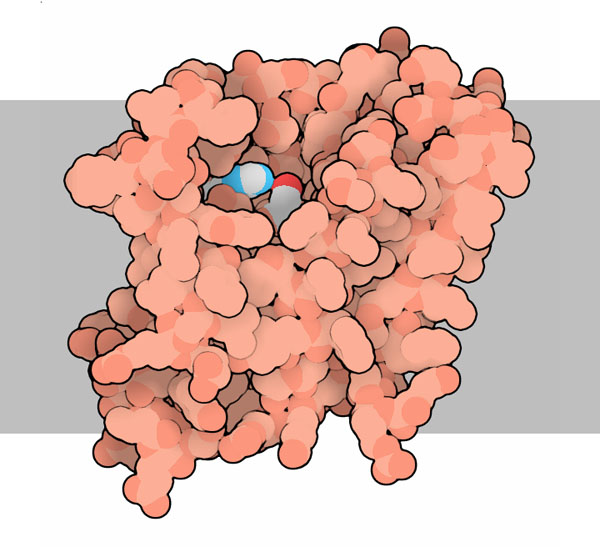
Рисунок 1. Белок GlpG в мембране клетки. Изображение из статьи Rhomboid Protease GlpG. 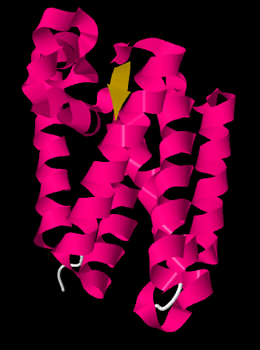
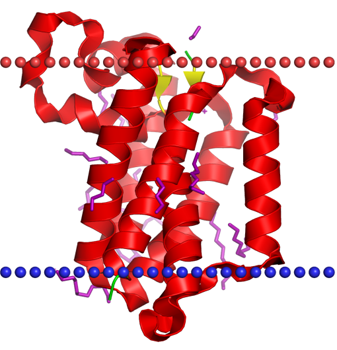
Рисунок 2 (слева). 3D-структура белка GlpG (PDB 4QO2, цепь A). Часть белка, ориентированная в сторону
p-мембраны, сверху. Изображение получено с помощью JMol.
Для составления репрезентативной выборки гомологов данного белка был произведен поиск по базе UniRef50 с помощью
Blast EBI. Я взяла 200 находок, e-value
лучшей - 1.3e-114, худшей - 9.2e-11. Результаты поиска можно посмотреть в виде картинки по
ссылке.
Затем был создан проект JalView (ссылка). На основании известной структуры была добавлена строка аннотации TM_REAL
с указанием трансмембранных участков (помечены буквой M). Примечание: в выравнивании JalView были скрыты некоторые практически полностью гэповые
колонки для, как мне кажется, лучшего восприятия структуры.
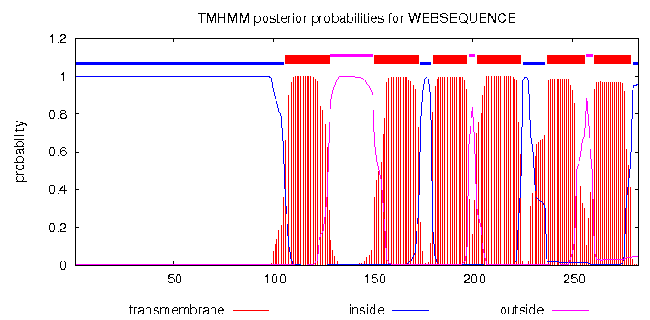
Рисунок 4. Результат предсказания трансмембранных участков для белка A0A0J8GTN0 с помощью сервиса TMHMM. На рисунках 5 и 6 представлен белок, остатки которого покрашены с использованием цветовой схемы Hydrophobicity и вручную заданной цветовой схемы соответственно. На рисунках 7 и 8 - те же изображения, но для установленного порога идентичности 12%. 

Рисунок 5. Белок GlpG с окраской по гидрофобности (JalView Hydrophobicity). Часть белка, обращенная к p-стороне мембраны,
находится снизу.
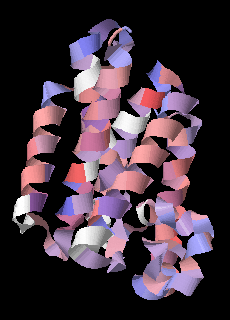
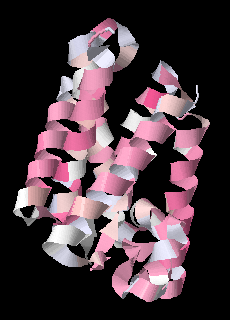
Рисунок 7. Белок GlpG с окраской по гидрофобности (JalView Hydrophobicity) и порогом идентичности 12%.
По изображениям видно, что в белке встречаются достаточно консервативные позиции в трансмембранных спиралях и в участках между спиралями. В спиралях часто встречаются L, M, W, V и другие гидрофобные аминокислоты, а также глицин. Иногда попадаются гидрофильные остатки, например, S и H. Возможно, они отвечают за каталитическую внутримембранную активность. Серин, возможно, располагается в активном центре фермента, так как GlpG относится к сериновым протеазам. В цитоплазматических участках гораздо больше гидрофильных, и более того, заряженных аминокислот, хотя гидрофобные также встречаются. Однако говорить о том, что многие участки консервативны, сложно, потому что изображения получены для низкого порога консервативности (я выбрала такой порог, чтобы цвета были в принципе различимы, на 30% окрашенными оставалось лишь пара колонок). |
|
© Наталия Кашко, 2016 |