Результаты секвенирования по Сэнгеру
Сборка последовательности по прочтениям
Исходные файлы: Forward Read , Reverse Read
Последовательность действий для получения итоговых выравниваний:
1. Определяем нечитаемые участки хроматограмм.
Нечитаемые участки в F - 5'-1...26- 3', 5'- 378...381- 3'
Нечитаемые участки в R - 3'-1...35- 5', 3'-387- 5'
2. В начале делаем выравнивание, где используем прямую цепочку как референсную последовательность,
а обратную как прочтение выравниваемое по ней. Так получаем промежуточный контиг. Затем повторяем выравнивание с использованием
промежуточного контига как референсной последовательности, а в прочтении ставим прямую обратную цепочку.
Так получаем референсную последовательность.
3. Анализируем сопоставление хроматограмм из последнего выравнивания и по максимуму распознаем неопознанные нуклеотиды в референсной последовательности.
Так получаем консенсусную последовательность.
4. После этого по консенсусной последовательности исправляем прямую и обратную цепочки и добавляем к ним нечитаемые концы.
Таким образом, получаем прямую, обратную и референсную последовательности без удаления концов. (исправления показаны маленькими буквами)
Анализ проблемных нуклеотидов
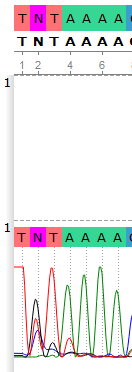
Рис.1
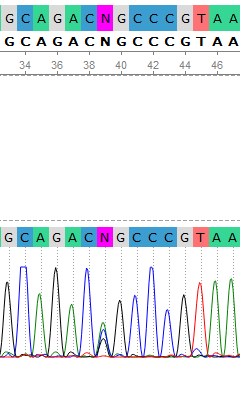
Рис.2
На позициях 2 и 39 возможны полиморфизмы. Нуклеотиды на этих позициях не удалось однозначно распознать ни программно, ни визуально. По результатам распознавания, на 2-ой позиции: B (С or G or T), на 39-ой: V (A or С or G)
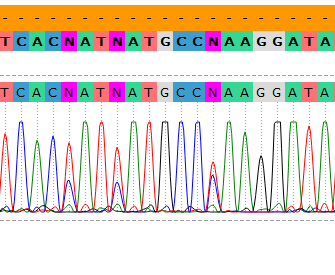
Рис.3
Нуклеотиды на 375, 378 и 384 позициях также, возможно, являются полиморфизмами. Их номера не попали на изображения, т.к. в начале они не попали на нумерацию референсной последовательности. По результатам распознавания на всех спорных позициях: Y (С or T)
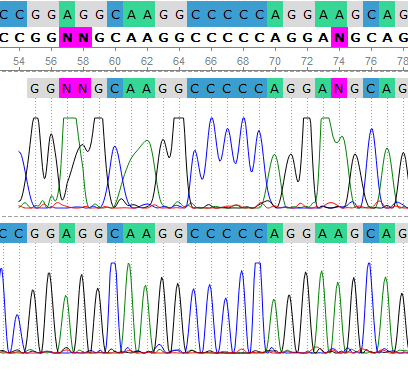
Рис.4
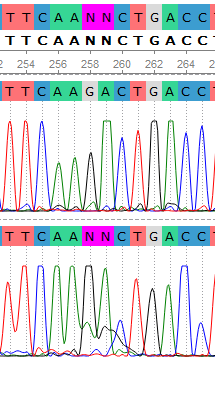
Рис.5
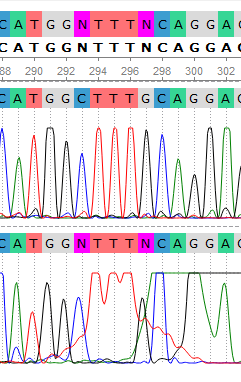
Рис.6
На позициях 57,58, 74, 258, 259, 293, 297 программно верно разобраны первоначально нечитаемые нуклеотиды. Визуально они тоже легко различимы.
По результатам разбора, на позициях: 57 - A, 58 - G, 74 - A, 258 - G, 259 - A, 293 - C, 297 - G.
Характеристика хроматограммы
Обе хроматограммы качественные. Нечитаемые участки находятся только на концах последовательности. Пятен краски почти нет. Локальное понижение качества хроматограмм возникает в основном из-за местного повышения уровня шума. Качество прочтения показано на рисунке ниже (рис.7)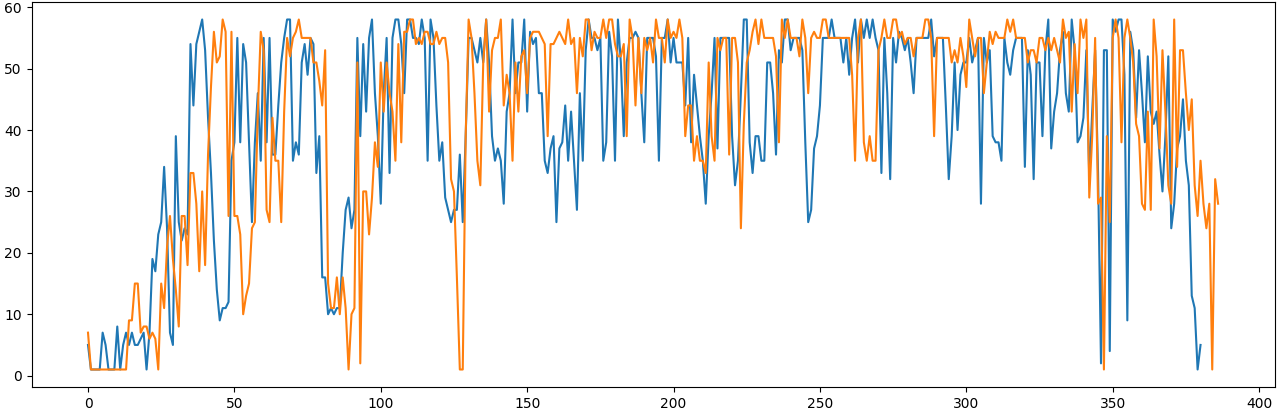
Рис.7. По оси X – номер нуклеотида, по оси Y – качество прочтения, синий график – качество прочтения прямой последовательности, оранжевый – качество прочтения прямой обратной.
Нечитаемый фрагмент хроматограммы
Пример: файл WSWS2950_H3_F_G09_2013-06-11-22-39-58.ab1 Был рассмотрен участок 70-103. Качество хроматограммы низкое из-за большого количества пятен краски и высокого уровня шума. Это может быть связано с ошибками во время проведения фореза.