Трансмембранные белки
Знакомство с базой данных OPM
Был выбран белок Poly-beta-1,6-N-acetyl-D-glucosamine export protein - Экспортный белок поли-бета-1,6-N-ацетил-D-глюкозамина, находящийся на наружной мембране грамотрицательных бактерий. Uniprot - PGAA_ECOLI, PDB - 4Y25| Толщина гидрофобной части | 23.8 Å |
| Координаты трансмембранных участков | 1(517-528), 2(540-548), 3(555-563), 4(577-584), 5(591-596), 6(607-615) , 7(622-627), 8(646-654), 9(661-667),10(678-685),11(695-703), 12(720-729),13(740-751), 14(760-770),15(778-786),16(796-806) |
| Среднее количество остатков в одном β-тяже белка | ~9 |
| Локализация | Внутренняя мембрана грамотрицательных бактерий |
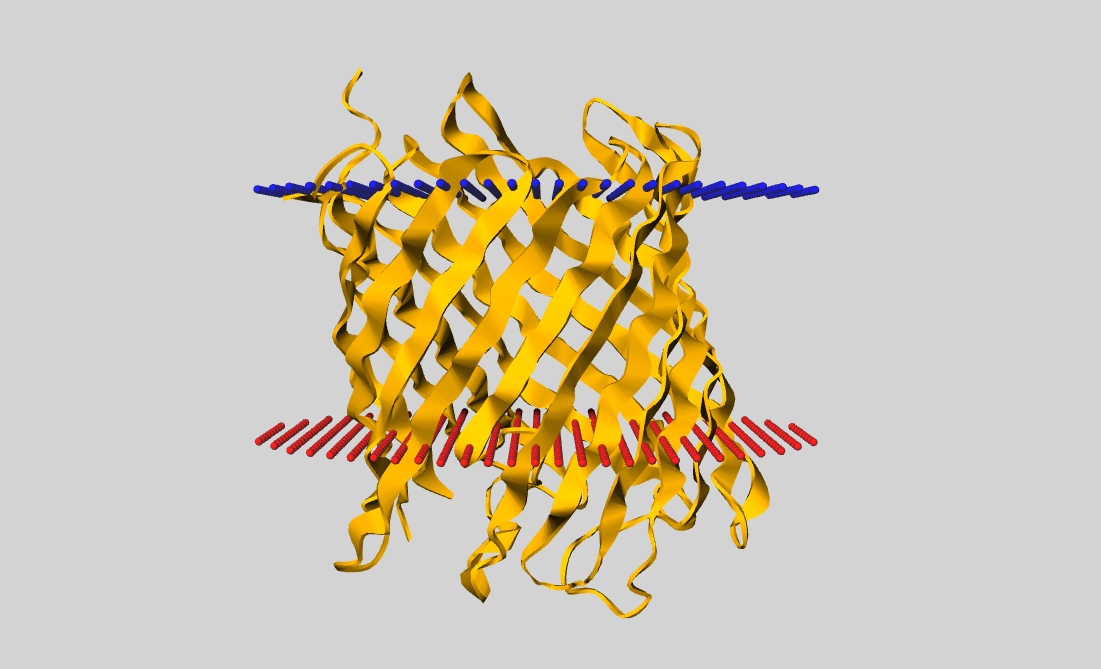
Рис.1. Изображение белка p-сторона направлена вниз

3line gff3
3line gff3
PPM: Предсказание положения выданного белка в мембране Выданный белок - Intermembrane transport protein PqIA (трансмембранный транспортный белок PqIA), принадлежащий Escherichia coli (strain K12).

Рис.4. Параметры PPM
| Толщина гидрофобной части | 27.8 ± 1.5 Å |
| Координаты трансмембранных участков | 1( 53- 67), 2( 96- 120), 3( 141- 163), 4( 172- 188), 5( 254- 272), 6( 301- 324), 7( 347- 366), 8( 381- 399) |
| Среднее количество остатков в одном β-тяже белка | ~20 |
| Локализация | Внутренняя мембрана грамотрицательных бактерий |

Рис.5. Изображение белка, p-сторона мембраны направлена вниз
4. Сравнение алгоритмов предсказания трансмембранных спиралей
| DeepTMHMM | PPM | |
|---|---|---|
| 1 | 55-71 | 53-67 |
| 2 | 99-121 | 96-120 |
| 3 | 138-157 | 141-163 |
| 4 | 174-189 | 172-188 |
| 5 | 255-275 | 254-272 |
| 6 | 304-325 | 301-324 |
| 7 | 347-371 | 347-366 |
| 8 | 380-400 | 381-399 |
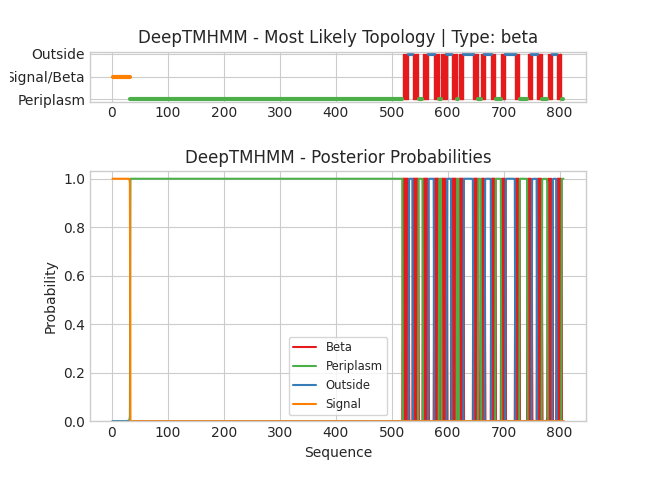
| DeepTMHMM | OPM | |
|---|---|---|
| 1 | 520-530 | 517-528 |
| 2 | 538-547 | 540-548 |
| 3 | 556-565 | 555-563 |
| 4 | 576-584 | 577-584 |
| 5 | 589-598 | 591-596 |
| 6 | 607-616 | 607-615 |
| 7 | 620-628 | 622-627 |
| 8 | 646-654 | 646-654 |
| 9 | 660-666 | 661-667 |
| 10 | 678-685 | 678-685 |
| 11 | 696-703 | 695-703 |
| 12 | 721-728 | 720-729 |
| 13 | 743-750 | 740-751 |
| 14 | 760-768 | 760-770 |
| 15 | 779-787 | 778-786 |
| 16 | 795-803 | 796-806 |
Из сравнительных таблиц видно, что результаты алгоритмов очень близки: общее число и положение трансмембранных структур совпадают. В обоих случаях между работой алгоритмов нет значимых различий смещений в чью-либо сторону по результатам. Однозначно судить о том, влияет ли на работу PPM уровень достоверности модели AplhaFold, так как сам по себе уровень достоверности на большей части модели достаточно высок, а по результатам работы PPM не было обнаружено какого-либо чёткого смешения в предсказании, которое одинаковым образом распространялось бы на весь результат работы
База данных TCDB
Белок PQIA_ECOLI имеет ТС-код 9.A.69.1.1: 9 - Не полностью охарактеризованные транспортные системы; A - унипортеры, антипортеры, симпортеры (подкласс); 153 - Семейство межмембранных фосфолипидных транслоказ (IMPL-T).
Белок PGAA_ECOLI имеет ТС-код 1.B.55.1.1: 1 - Канал; В - предполагаемый белок-транспортер; 55 - Семейство полиацетилглюкозаминовых поринов (PgaA).