Нуклеотидный локальный BLAST
Поиск гомологов белков в неаннотированном геноме
В практикуме 5 был произведен анализ хроматограммы секвенирования по Сэнгеру и получен консенсус для исследуемой последовательности.
Для установления таксономического положения и функции данной последовательности был применён blastn, так как про эту последовательность не было известно ничего,
что могло бы как-то ограничить или уточнить поиск.
Параметры запуска: database: nr/rt , max target sequences: 100, expect threshold: 0.05, word size: 7.
Результат: выдача blastn
Функция: данная последовательность почти гарантированно является участком гена или полным геном гистона H3, который участвует в свёртке ДНК и
регуляции транскрипционной активности. Это подтверждается тем, что в 99 из 100 лучших результатах выдачи с E-value меньше 5е-130 и процентом совпадения выше 87%
ген именно этого гистона соответствует записи.
Tаксономия: данная последовательность с высокой вероятностью принадлежит кольчатому многощетинковому червю(Polychaeta) семейства Sedentaria.
Можно предположить, что последовательность принадлежит Brada inhabilis, так как наиболее высокие по Identity находки в blastn принадлежат именно этому виду.
Поиск генов белков в неаннотированной нуклеотидной последовательности
Для работы был выбран контиг из сборки генома Ursus maritimus (белого медведя). Выбранный контигДля поиска использовался blastx (т.к. необходим поиск по генам в данном контиге, то лучше будет протранслировать его в пептид и поискать гомологичные), со следующими параметрами: database: swissprot (по достоверно существующим белкам), organism: exclude white bear (taxid:29073), expect threshold: 0.05, word size: 2.
Выдача blastx
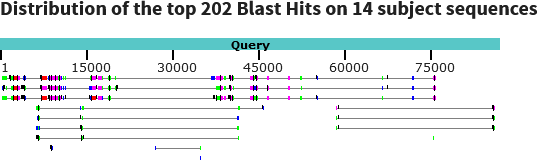
С достаточно большой вероятностью в контиге присутствует обратная транскриптаза элемента LINE-1(LINE-1 retrotransposable element ORF2 protein), которая также обладает эндонуклеазной активностью.
Интепретация карты локального сходства гомологичных хромосом двух бактерий
Для сравнения были выбраны геномы двух представителей класса Альфа-протеобактерии с Complete genome assembly: Rhizobium leguminosarum bv. viciae (AM236080.1) и Rhizobium etli bv. mimosae str. Mim1(CP005950.1)
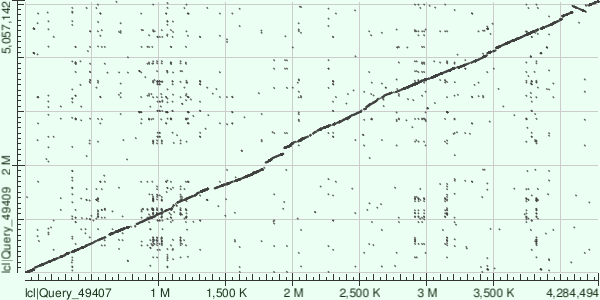
Используя megablast (базовые настройки), была получена карта локального сходства двух последовательностей. По представленному выше dotplot'у можно видеть,
что выбранные последовательности являются сходными, но присутствует большое количество шумов, что может быть обусловлено разницей в длине геномов.
На участках 600-650К, 800-850К, 1400-1450К, 1800-1850К, 1950-2000К присутствуют индели, а на участке 4100-4250К находится инверсия - это свидетельствует
об отличиях в геномах родственных бактерий.