Ортологи и Паралоги
| Название | Мнемоника |
|---|---|
| Bacteria; Terrabacteria group; Actinobacteria; Actinomycetia; Acidothermales; Acidothermaceae; Acidothermus; Acidothermus cellulolyticus | ACIC1 |
| Bacteria; Terrabacteria group; Actinobacteria; Actinomycetia; Micrococcales; Micrococcaceae; Arthrobacter; Arthrobacter sp. | ARTS2 |
| Bacteria; Terrabacteria group; Actinobacteria; Actinomycetia; Micrococcales; Microbacteriaceae; Clavibacter; Clavibacter michiganensis | CLAMS |
| Terrabacteria group; Actinobacteria; Actinomycetia; Corynebacteriales; Mycobacteriaceae; Mycolicibacterium; Mycolicibacterium vanbaalenii | MYCVP |
| Bacteria; Terrabacteria group; Actinobacteria; Actinomycetia; Corynebacteriales; Mycobacteriaceae; Mycobacterium; Mycobacterium tuberculosis complex; Mycobacterium tuberculosis | MYCTU |
| Bacteria; Terrabacteria group; Actinobacteria; Actinomycetia; Corynebacteriales; Corynebacteriaceae; Corynebacterium; Corynebacterium efficiens | COREF |
| Bacteria; Terrabacteria group; Actinobacteria; Rubrobacteria; Rubrobacterales; Rubrobacteraceae; Rubrobacter; Rubrobacter xylanophilus | RUBXD |
| Bacteria; Terrabacteria group; Actinobacteria; Actinomycetia; Streptomycetales; Streptomycetaceae; Streptomyces; Streptomyces avermitilis | STRAW |
Задание 1
С использованием команд провели поиск гомологов белка CLPX_ECOLI и получили Список находок, из которого были отдельно выписаны их AC.Задание 2
В программу Jalview 2.11.2.4 был загружен список находок, там же было построено выравнивание соответствующих последовательностей алгоритмом Muscle. Выравнивание импортированно в прорамму MEGAX методом Analyze. Получено дерево (Newick) алгоритмом Neighbor-joining.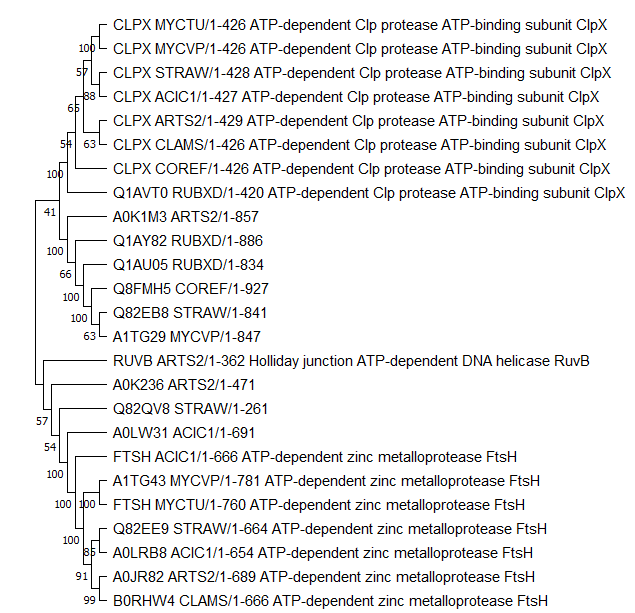
Рис.1. Изображение дерева
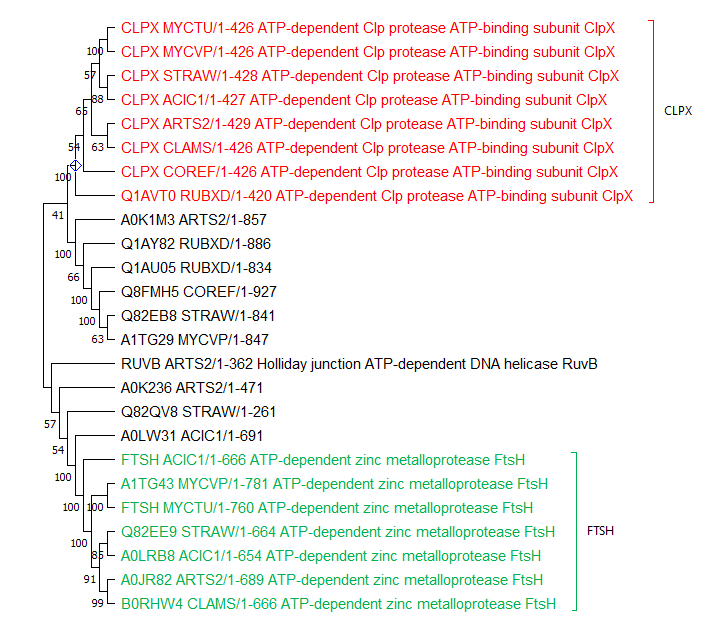
Рис.2. Изображение дерева с отмеченными ортологичными группами
Паралоги: A0LRB8_ACIC1 и FTSH_ACIC1, Q1AY82_RUBXD и Q1AU05_RUBXD, A0K1M3_ARTS2 и A0K236_ARTS2

Рис.3. Изображение дерева со "схлопнутыми" ортологичными группами
Зеленая ортологичная группа соответствует АТФ-зависимой цинковой металлопротеазой FtsH. Этот белок не был найден у бактерий COREF и RUBXD. Без учёта недостающих белков топология дерева совпадает с деревом из первого практикума.