Построение и визуализация электронной плотности
При помощи этого скрипта на pymol были получены все использующиеся в практикуме изображения. В качества исследуемой структуры была выбрана структура 4GUW, доступная для скачивания в PDB. Данные о структуре представлены в таблице 1.
Таблица. 1. Информация о структуре.
| PDB id | 4GUW |
|---|---|
| Разрешение структуры | 1.6 |
| Длина цепи белка, аа | 246 |
| Известность сервису EDS (Electron Density Server) | Да |
Вначале посмотрим на электронную плотность вокруг полипептидной цепи структуры (таблица 2). В одном случае будем изображать только ее, во втором - плотность во всей ячейке. Видим, что на уровне обрезки 1.5 шум почти полностью убирается (видна только плотность от единицы белка и соседних в ячейке), при этом большая структуры все еще покрыта ею. На уровне обрезки 3 видим, что часть структуры электронной плотности не покрыта
Таблица. 2. Визуализация электронной вокруг всей структуры.
| Уровень подрезки (σ) | Электронная плотность только вокруг цепи | Электронная плотность во всей ячейке |
|---|---|---|
| 0.5 | 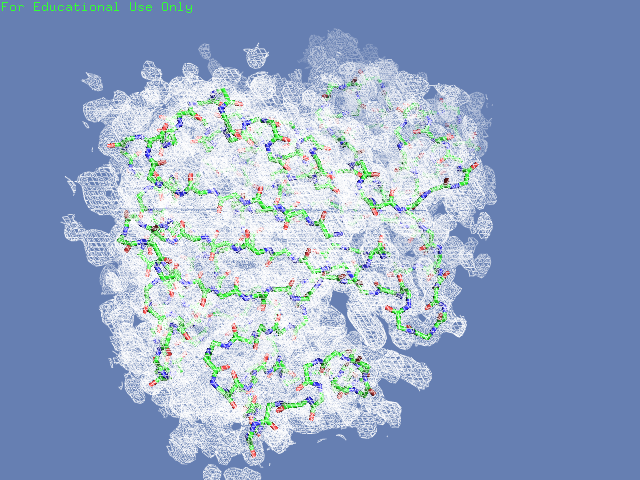 |
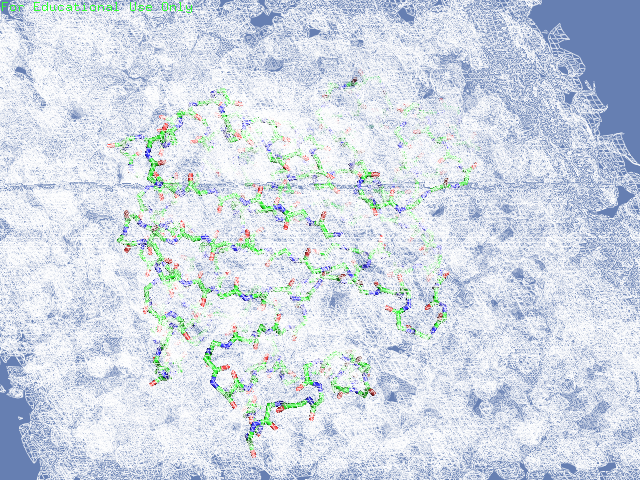 |
| 1 | 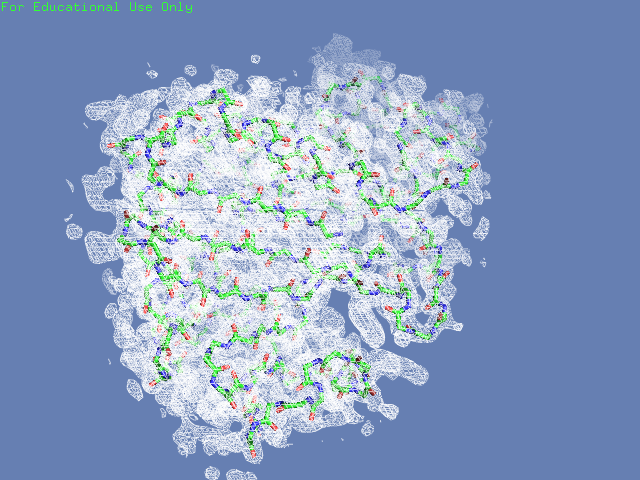 |
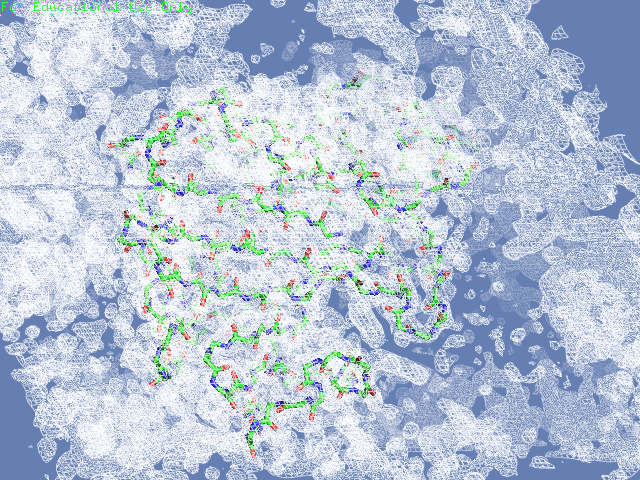 |
| 1.5 | 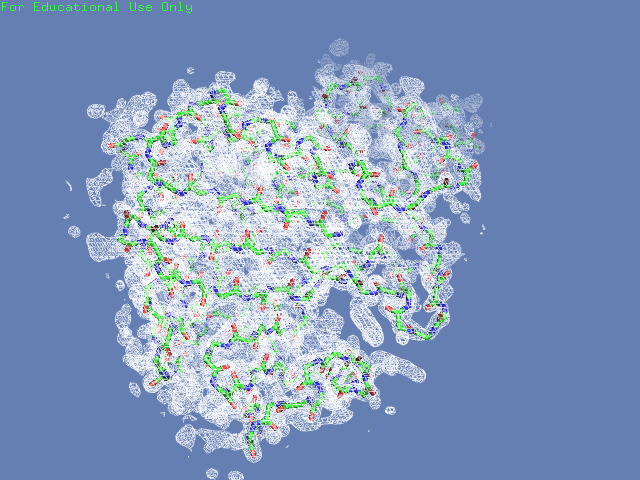 |
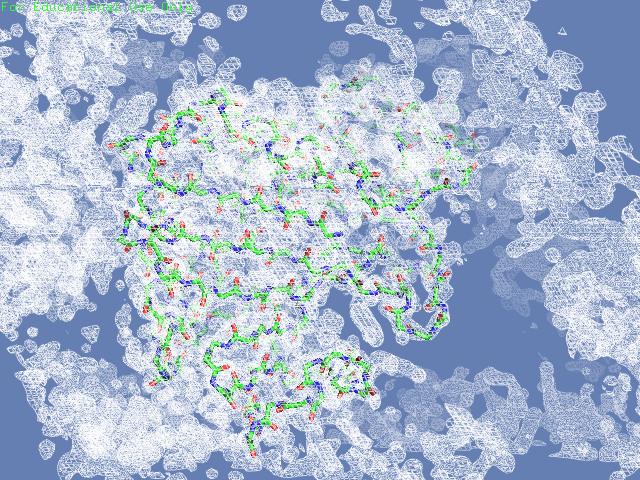 |
| 2 | 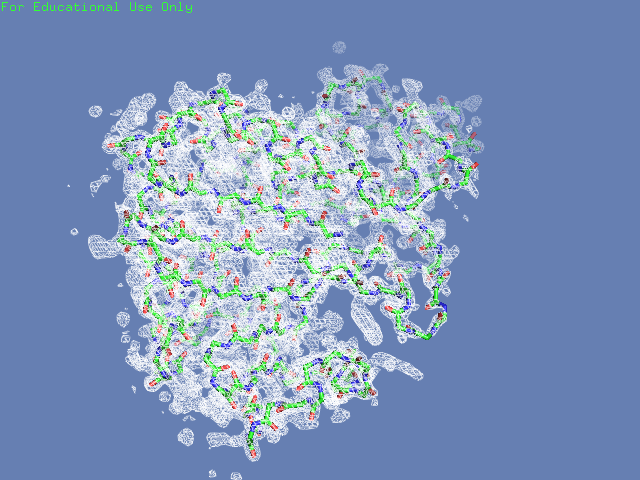 |
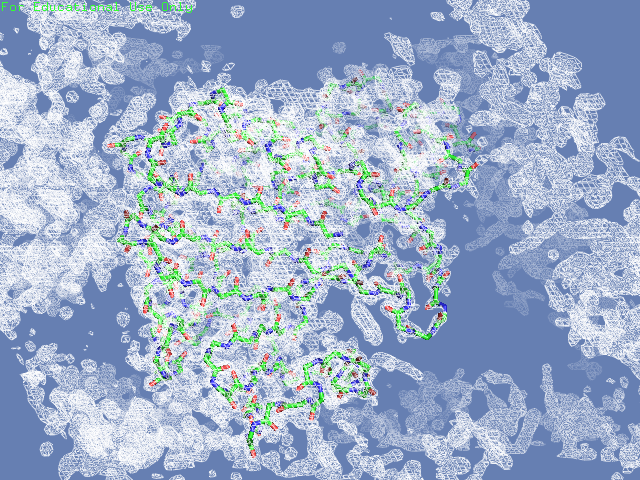 |
| 2.5 |  |
 |
| 3.0 |  |
 |
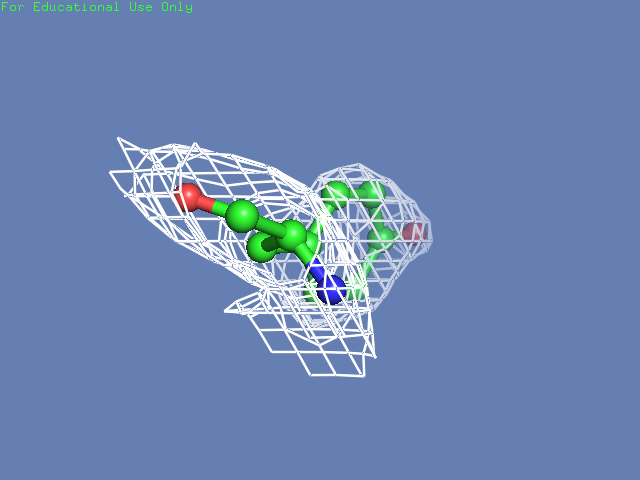
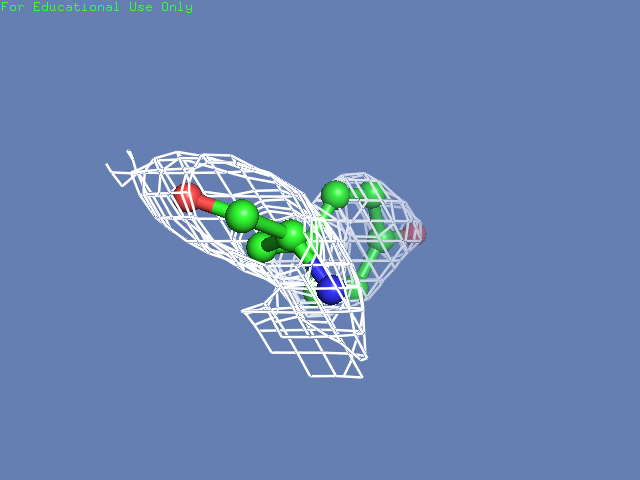
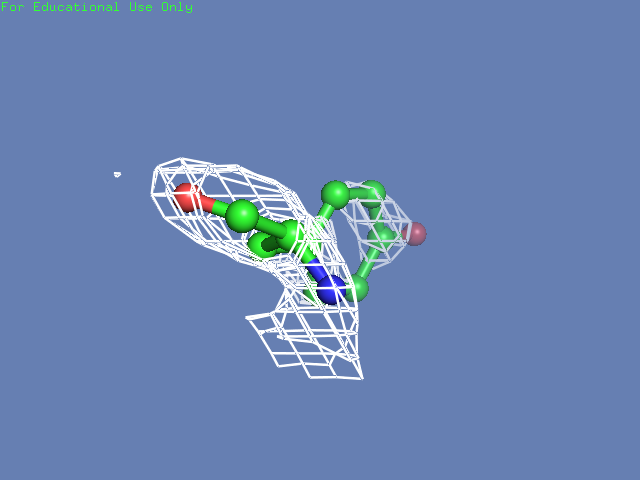
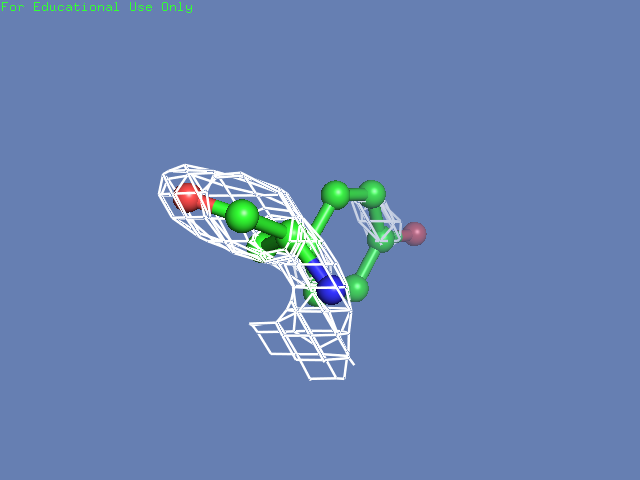
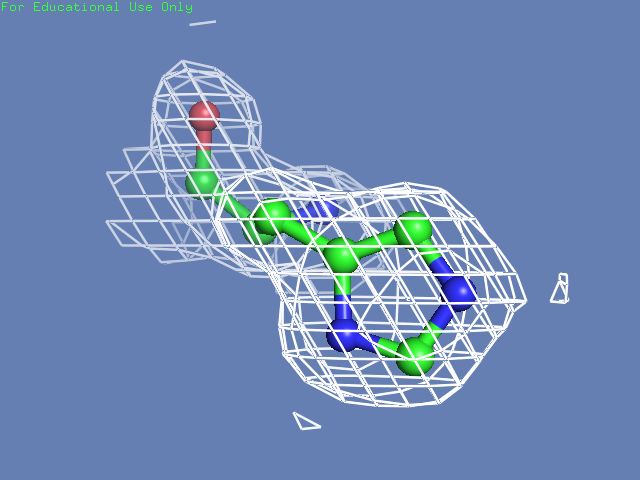
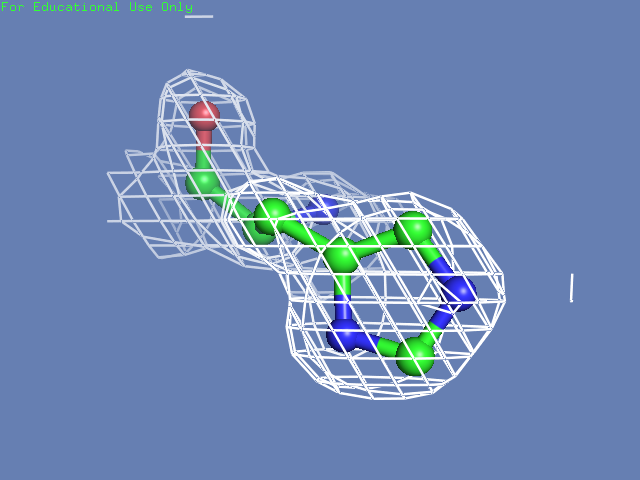
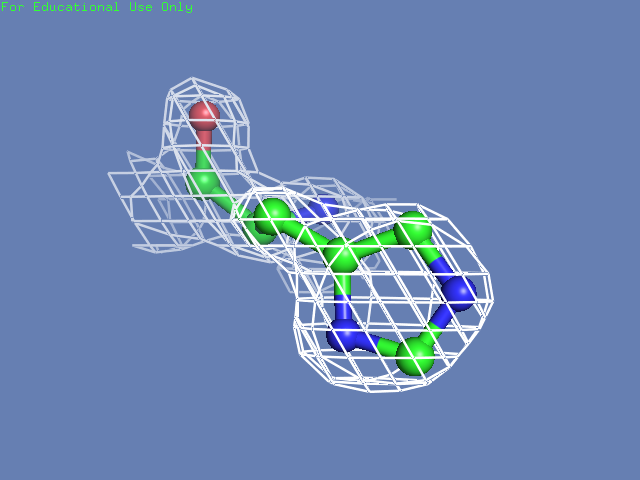
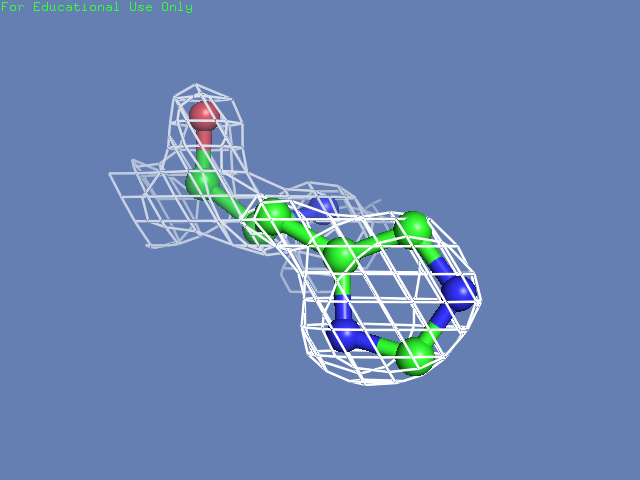
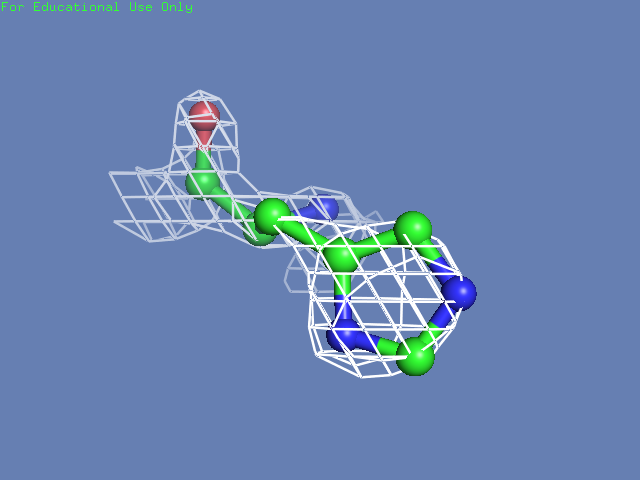
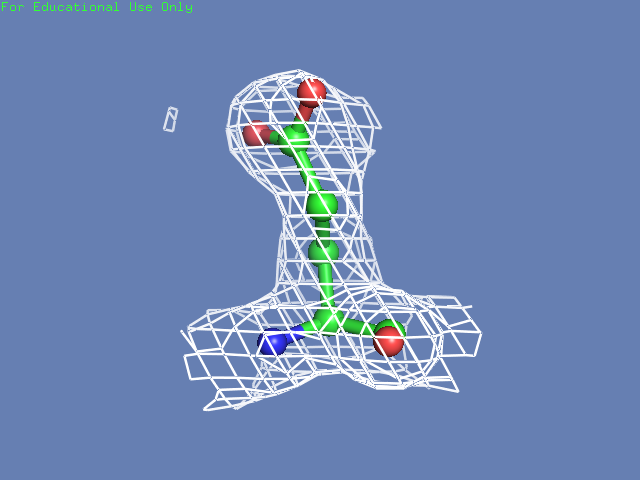
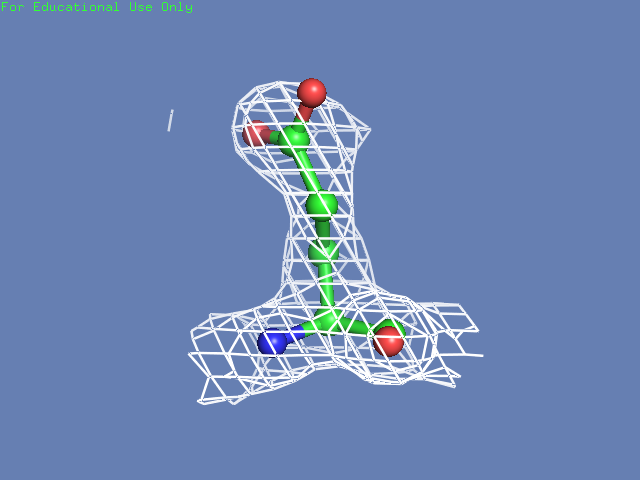
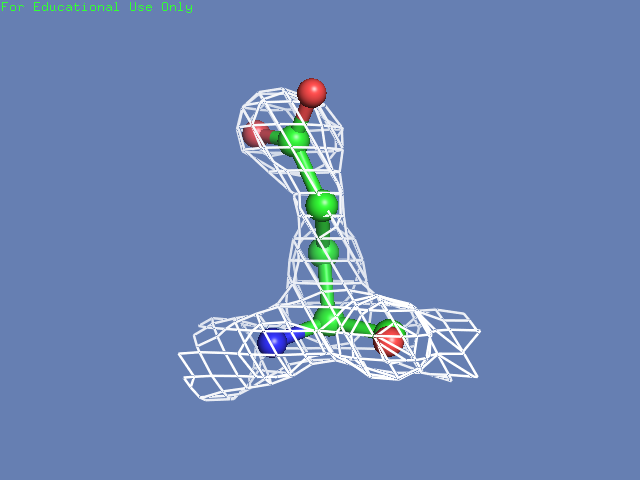
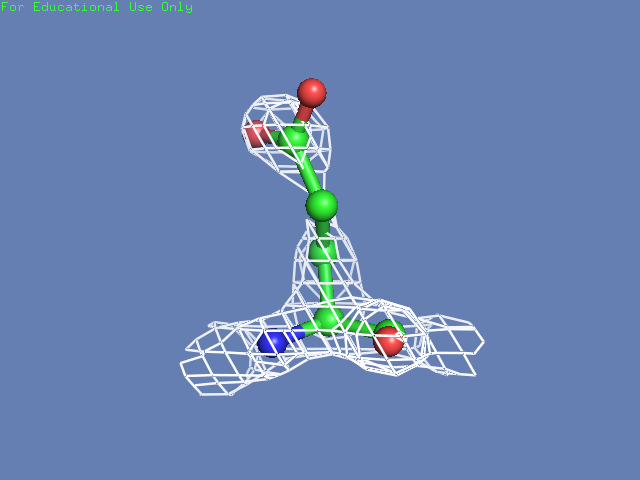
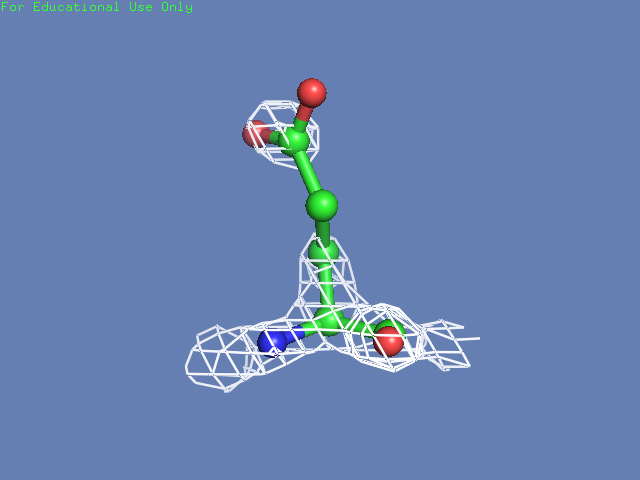
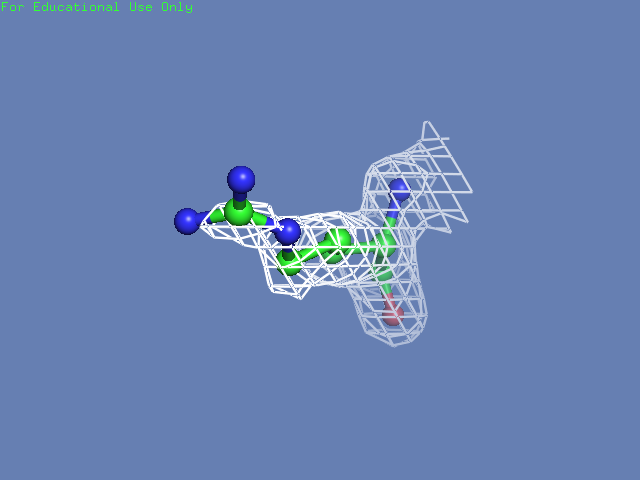
Теперь посмотрим на плотность в районе различных остатков. (таблица 3). Видим, что для большинства остатков положение остовной части можно определить и при уровне подрезки равном 3, а вот положение боковой цепи уже 1.5 у Arg100 определить невозможно, а при уровне подрезки 3 ее можно определить только у Glu85 и His51
Таблица. 3. Визуализация электронной вокруг отдельны
| Остаток | Уровень подрезки 0.5 σ | Уровень подрезки 1 σ | Уровень подрезки 1.5 σ | Уровень подрезки 2 σ | Уровень подрезки 2.5 σ | Уровень подрезки 3 σ |
|---|---|---|---|---|---|---|
| Pro26 |  |
 |
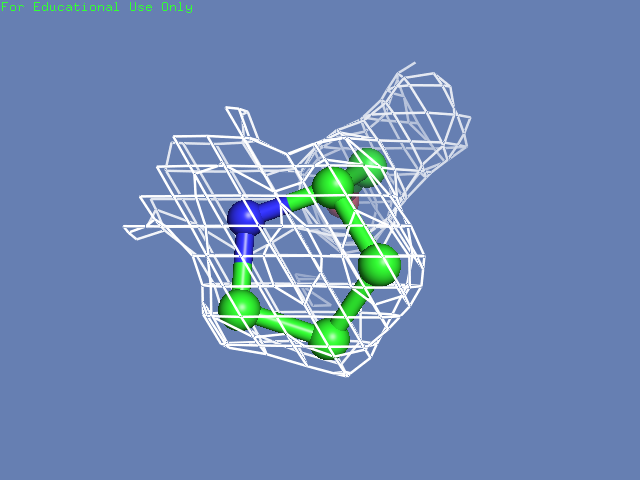 |
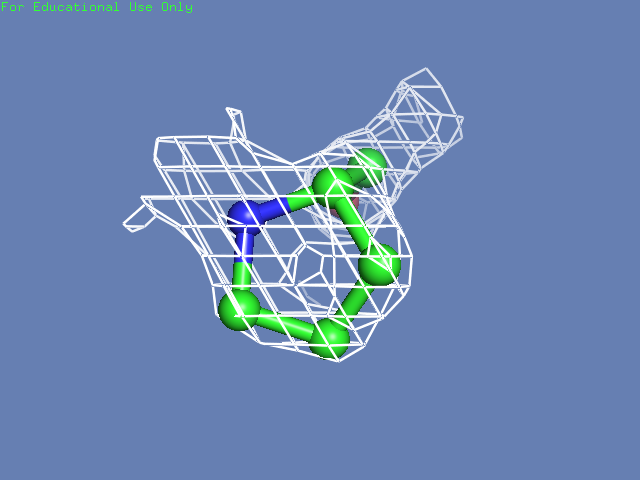 |
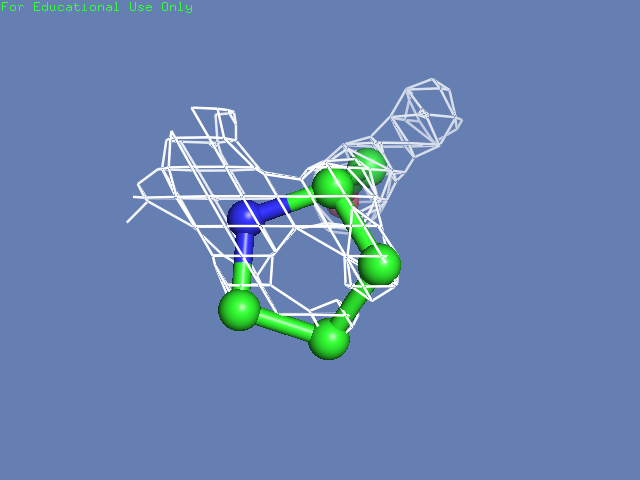 |
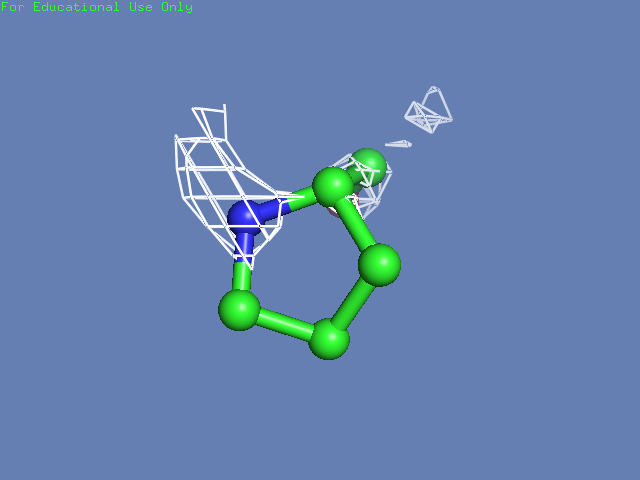 |
| Tyr32 | 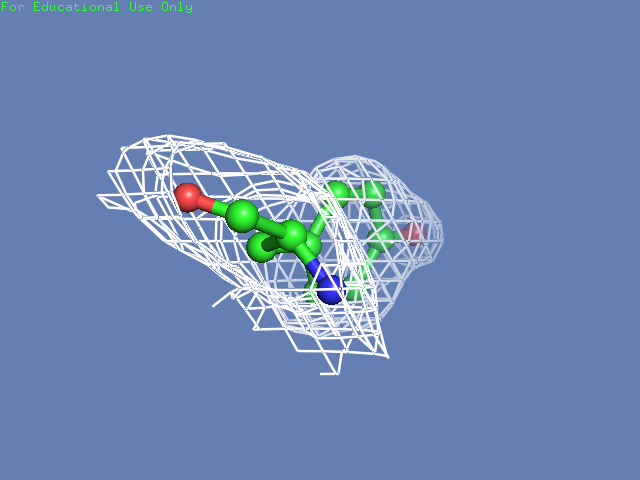 |
 |
 |
 |
 |
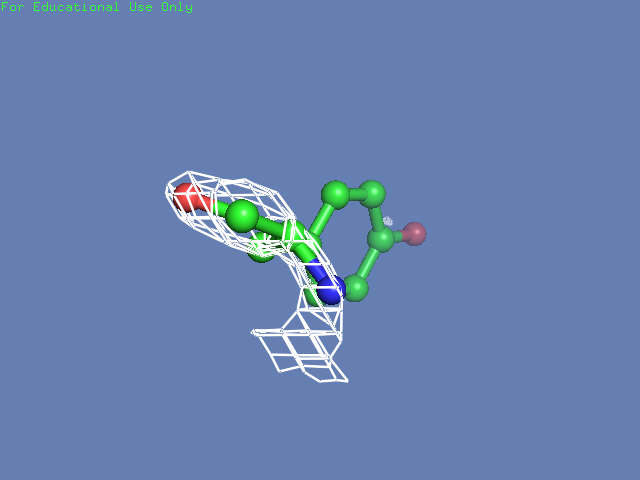 |
| His51 | 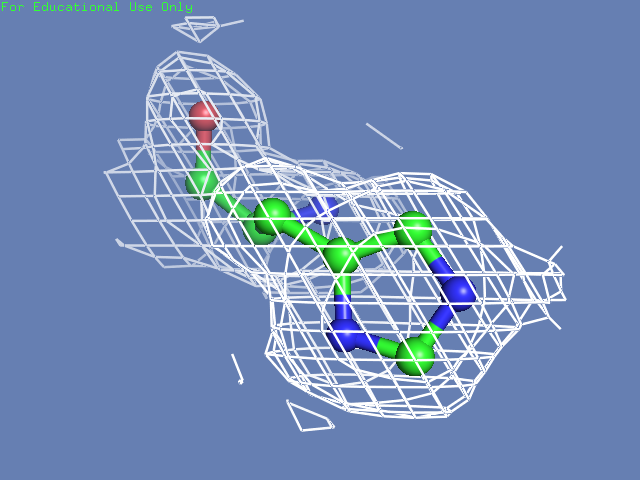 |
 |
 |
 |
 |
 |
| Glu85 | 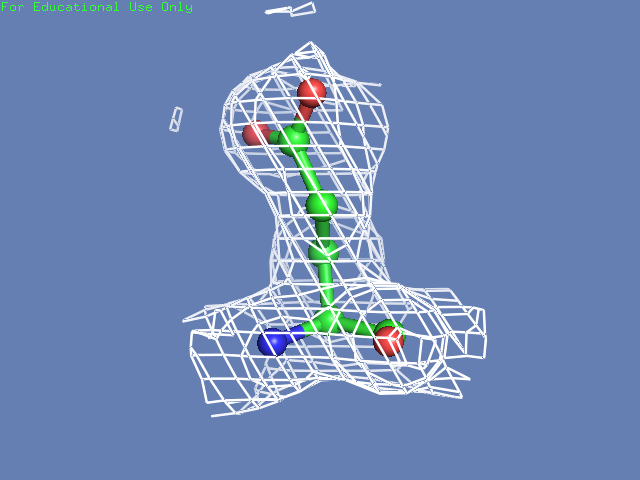 |
 |
 |
 |
 |
 |
| Arg100 | 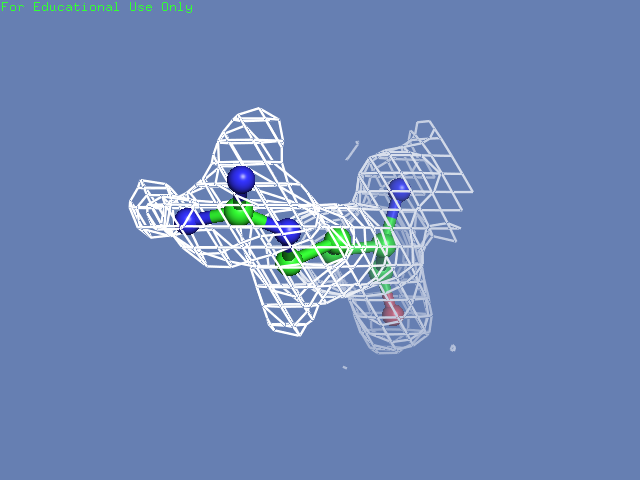 |
 |
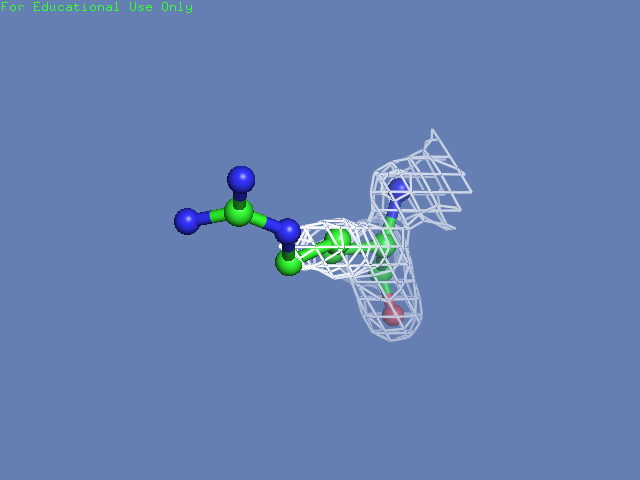 |
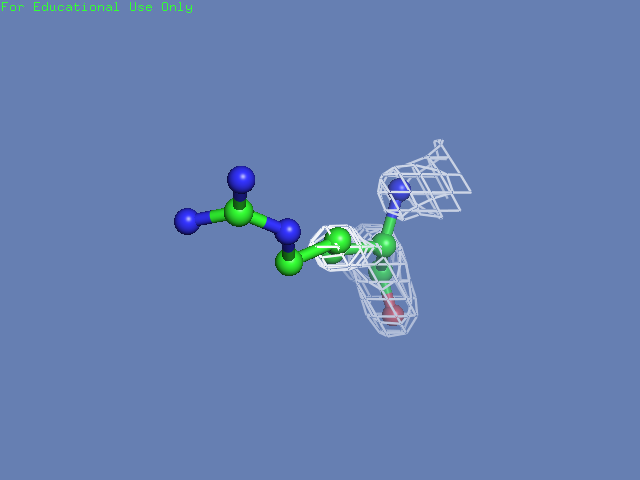 |
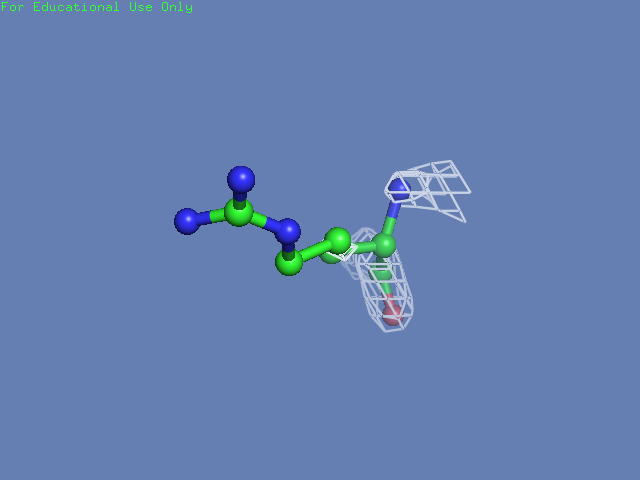 |
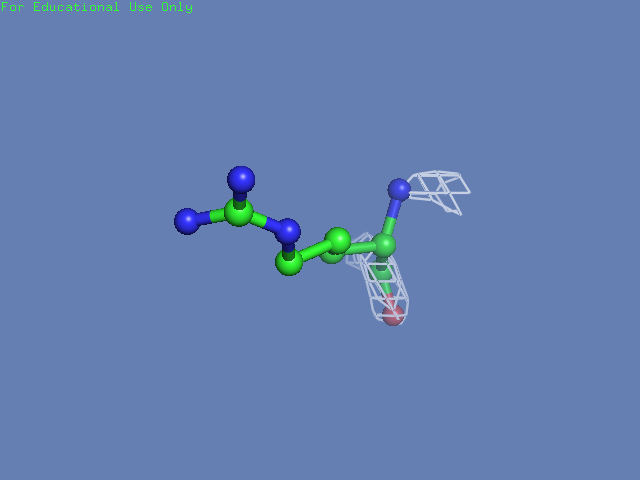 |
Несмотря на это можно заключить, что качество структуры достаточно высокое (требование к определению положения боковой цепи на большом уровне подрезки достаточно избыточно, да и понятно, что боковые цепи детектировать значительно сложнее). Потому структура прекрасно подходит для дальнейшей работы.
