Построение поверхности
В данном практикуме необходило было выбрать комплекс димера пуринового рецептора с ДНК. Мною был выбран комплекс 1vpw.pdb. С помощью достроения симметричной ячейки с помощью команды:
symexp sym, 1vpw, 1vpw, 4и удалением лишних единиц белка, я получил димер данного белка. Далее для него было построено 3 изображения:
- поверхности контакта мономера белка с симметричным мономером на фоне остовной (ribbon) модели мономера - рис. 1;
- поверхности контакта димера белков с двойной спиралью ДНК на фоне остовной модели части белка, вовлечённой в контакт - рис. 2;
- поверхности контакта ДНК с димером белков на фоне проволочной (sticks) модели двойной спирали - рис. 3;
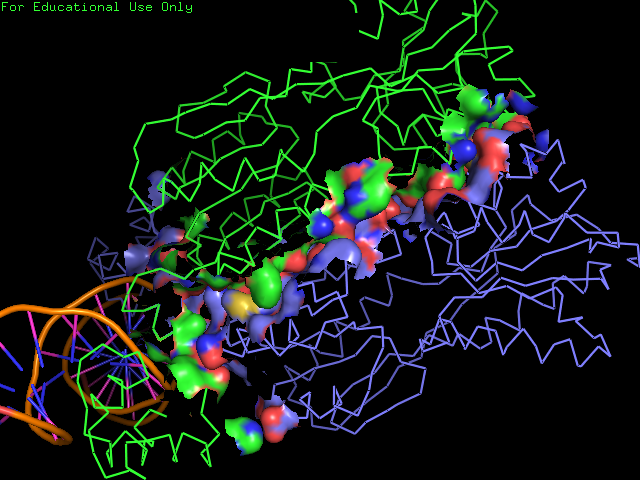
Рисунок 1. Поверхность контакта мономера белка с симметричным мономером
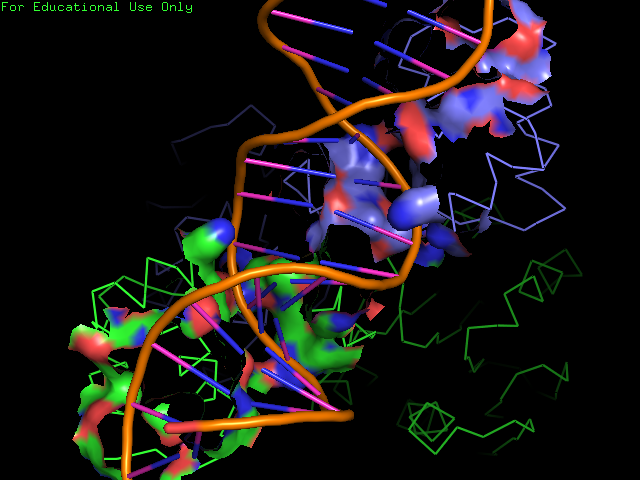
Рисунок 2. Поверхность контакта димера белков с двойной спиралью ДНК
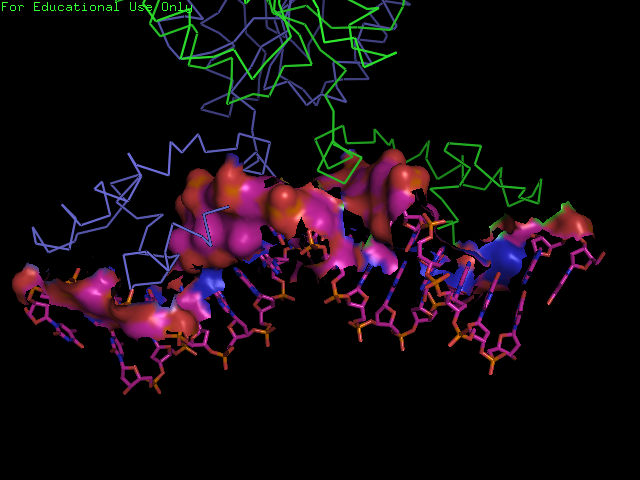
Рисунок 3. Поверхность контакта ДНК с димером белков
Далее с помощью сервиса CLUD были получены гидрофобные кластеры в данной структуре. Затем с помощью Jupyter Notebook, которую можно скачать этой ссылке оно было преобразовано в скрипт для Pymol, при этом маленькие сластеры были удалены. Затем было сделано изображение, как на рисунке1, но на котором поверхность, относящаяся к атомам, входящим в найденные гидрофобные кластеры, выделена цветом ( рис. 4). Скачать сессию паймол, в которой все подготовлено для получения изображений этого практикума можно по этой ссылке.
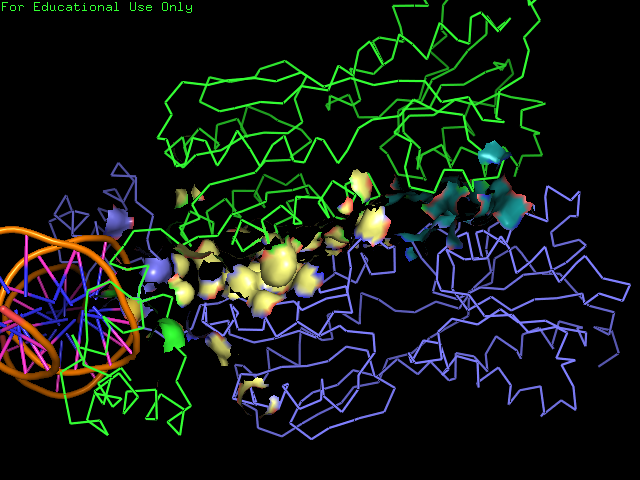
Рисунок 4. Поверхность контакта ДНК с димером белков
