| Учебный сайт Саши Погорельской |

|
|
||||||
| Главная | Семестры | Скрипты | Обо мне | Ссылки | ||
|
|
Поиск гомологов с помощью BLAST BLAST (Basic Local Alignment Search Tool) позволяет находить участки сходства последовательностей. Программа сравнивает исходную последовательность с находящимися в выбранной базе данных и вычисляет статистическую вероятность гомологии между ними, основываясь на качестве локальных выравниваний. Работать с программой можно на этом сайте. Для нахождения гомологов белков используют blastp (protein-protein BLAST), то есть поиск по базе данных белков аминокислотной последовательности. Я искала гомологи белка-регулятора транскрипции из 1 семестра (можно посмотреть файл с последовательностью в fasta-формате) в базе данных Uniprot/SwissProt. Фрагмент полученной таблицы с характеристикой одной из находок представлен ниже: Таблица 1. Описание одной из находок BLAST
В колонке Description содержится имя последовательности. Одна находка может содержать несколько участков сходства, тогда, максимальный вес (Max score) их этих участков будет не равен сумме этих весов (Total score), указаны нормированные веса. То, какую часть запроса (query) покрывает находка показано в колонке Query cover. Процент совпавших аминокислотных остатков - Ident, а идентификатор и ссылка на запись в базе данных - Accession. Параметр E-value при значениях меньше e-3 приблизительно равен вероятности ошибки, для этого анализируется банк случайно перемешанных последовательностей. Участок, найденный BLAST располагается с 11 по 61 аминокислоты исходной последовательности, а в находке с 16 по 66. При этом в выравнивании нет гепов. Из 51 аминокислоты совпадают 21 (41%), имеют положительное значение в матрице выравнивания 37 (66%). При использовании BLAST для сравнения 2 последовательностей он выдает более 1 участка сходства. Выравнивания этих участков можно посмотреть здесь. Это удобно рассматривать в графическом представлении - на карте локального сходства, для исходной и найденной последовательностей она приведена на рисунке 1 (query по горизонтали). 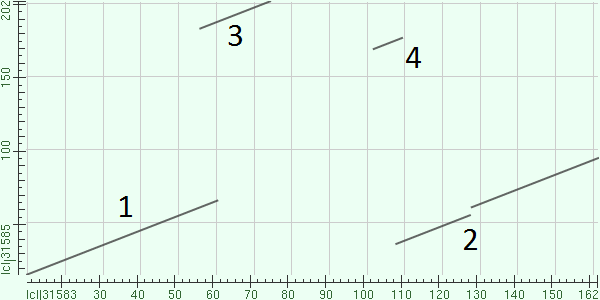
Рисунок 1. Карта локального сходства между запросом и выбранной находкой. Участки сходства пронумерованы в том же порядке, что и в файле выравнивания По рисунку 1 можно понять, что в участке сходства 2 в находке произошла вставка (или в query делеция). У участков 1 и 2 для находки есть одинаковые части, это, скорее всего, результат дупликации. Более короткие сходства (3 и 4), вероятно, являются случайными или дупликацями. BLAST позволяет выполнять поиск по определенным таксонам. Поиск по эукариотным организмам гомологов белка-регулятора транскрипции дал результаты с E-value больше 0,3. Это означает, что вероятность того, что эти последовательности гомологичны является почти нулевой. Я искала белки, гомологичные регулятору транскрипции, из близких таксонов. Критерием гомологии для этого задания принят порог E-value меньше 1e-03. В базе данных SwissProt поиск по видам рода Syntrophus (кроме Syntrophus aciditrophicus), по родам семейства Syntrophaceae (кроме Syntrophus), семействам порядка Syntrophobacterales (кроме Syntrophaceae) и по классу Deltaproteobacteria (кроме Syntrophobacterales) не дал результатов. Поэтому я выбрала другую базу данных - Reference protein (RefSeq_protein). В семействе Syntrophaceae нашелся род Desulfomonile, представители которого содержат гомологичные последовательности. В таблице 2 представлена информация о нескольких находках этого поиска. Таблица 2. Описание одной 7 находок BLAST по роду Desulfomonile
Выравнивание этих последовательностей в формате fasta можно посмотреть здесь, а скачать проект JalView отсюда. Участков сходства довольно мало. Абсолютно консервативных колонок почти нет (всего 4), однако заметен консервативный участок, представленный на рисунке 2. С 31 по 76 аминокислотные остатки не встречается гепов, а также довольно много функционально консервативных колонок - 11. Все этой части выравнивания встречается еще 4 такие колонки (одна из них абсолютно консервативна, во всех последовательностях - глицин). То есть всего 2,4% абсолютно консервативных позиций и 9% функционально. 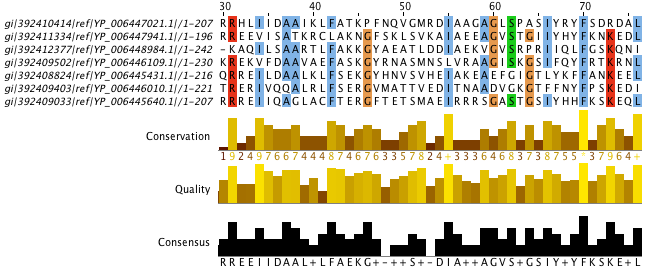
Рисунок 2. Консервативный участок выравнивания 7 находок |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| © Pogorelskaya Sasha | Last modification date: 19.02.15 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||