| Учебный сайт Саши Погорельской |

|
|
||||||
| Главная | Семестры | Скрипты | Обо мне | Ссылки | ||
|
|
Совмещение структур. 3D поиск Сервис PDBeFold находит 692 структурных гомолога одной из цепей регулятора транскрипции семейства TetR бактерии Syntrophus aciditrophicus SB (идентификатор PDB 3S5R). Находка с худшим RMSD (4.42) имеет сходство 70% вторичных структур. Для построения структурного выравнивания я выбрала 4 гомолога. Информация о них представлена в таблице 1. Для всего пространственного выравнивания RMSD равен 2.735, при этом выровнено 5 вторичных структур и 148 аминокислотных остатков. Таблица 1. Гомологи 3S5R, выбранные для построения пространственного выравнивания
Белки очень похожи структурно, практически все α-спирали расположены очень близко друг к другу, лишь незначительно отклоняясь (рисунок 1). Подвижнее всего, как и ожидается, петли. 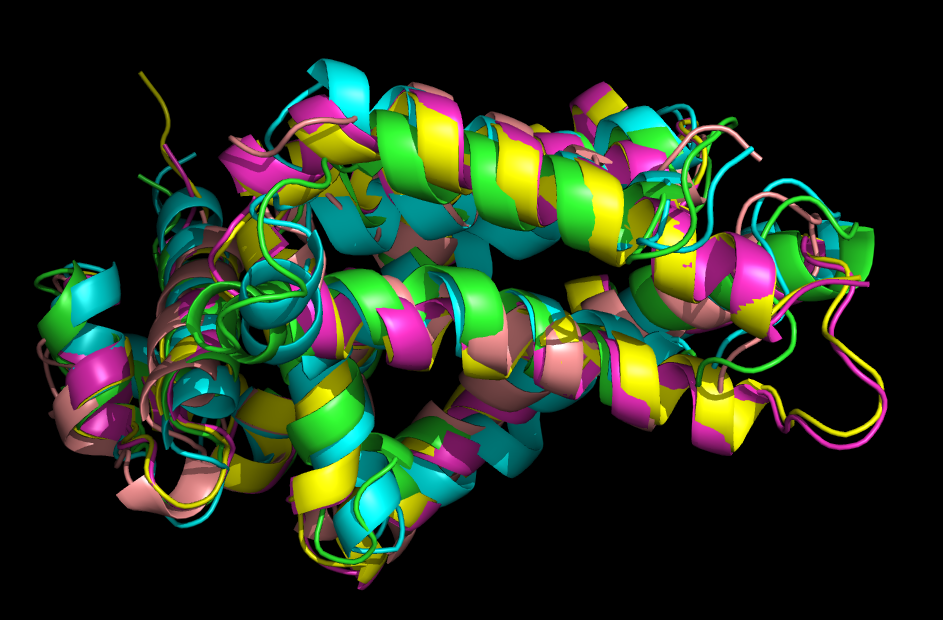
Рисунок 1. Пространственное совмещение 3S5R и его 4 гомологов. Изображение получено с помощью Pymol Выравнивание последовательностей по этому совмещению структур можно скачать тут (его часть представлена на рисунке 2). На рисунке 3 приведена часть множественного выравнивания, построенного с помощью алгоритма muscle. Эти 2 выравнивания очень похожи, отличаются только деталями. Например, случай отличия выделен на рисунках 2 и 3 красным прямоугольником. В выравнивании, соответствующем пространственному совмещению есть дополнительный геп в каждой из последовательностей, обусловленный наличием только трех совмещенных аминокислотных остатков из четырех, выделенных в прямоугольник. С точки зрения множественного выравнивания, гидрофобные остатки Val (позиция 49) лучше поставить в одну колонку с Met, а не с Arg, как это происходит на самом деле. Из-за этого в выравнивании по пространственному совмещению есть дополнительный геп. 
Рисунок 2. Часть выравнивания аминокислотных последовательностей согласно пространственному (такие аминокислоты указаны большими буквами). Изображение получено с помощью JalView 
Рисунок 3. Часть выравнивания аминокислотных последовательностей, полученного с помощью muscle. Изображение получено с помощью JalView Гибкое совмещение Часто жесткого совмещения, при котором модель белка сохраняется неизменной, недостаточно для определния сходства. Например, на рисунке 4 представлено жесткое совмещение 2 белков (идентификаторы PDB 2SAS и 1JFJ), видно, что белки совершенно непохожи. Если же построить совмещение алгоритмом, предсполагающим некоторое количество изгибов цепи, получается намного более хорошее - рисунок 5. 
Рисунок 4. Жесткое пространственное совмещение 2SAS и 1JFJ (алгоритм jFatCat-rigid). 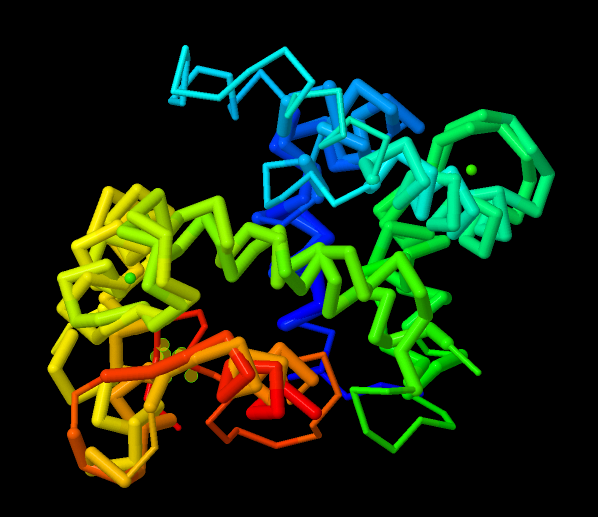
Рисунок 5. Гибкое пространственное совмещение 2SAS и 1JFJ (алгоритм jFatCat-flexible). |
||||||||||||||||||||||||||||||||||||
| © Pogorelskaya Sasha | Last modification date: 10.01.17 | |||||||||||||||||||||||||||||||||||||