BLAST
Задание 1
При помощи BLASTP были найдены последовательности, сходные с последовательностью белка Elongation factor 1-alpha археи Acidilobus saccharovorans 345-15 (идентификатор в базе данных RefSeq YP_003816619.1). Поиск выполнялся по базе данных Swiss-Prot, т.к. при поиске в базе данных Refseq_protein было найдено слишком большое количество "хороших" последовательностей и ни одной "плохой".
Для выполнения задания на сайте NCBI была выбрана программа BLAST, которая ищет последовательности, схожие с данной.
Затем был выбран protein blast, т.к. необходимо найти сходные аминокислотные последовательности белков.
В окно для запрашиваемой последователности (Query sequence) был введен AC данного белка из базы данных RefSeq (YP_003816619).
Было внесено изменения в параметры алгоритма: максимальное число находок было увеличено до 20000, чтобы найти максимальное количество схожих
последователностей.
Здесь можно ознакомиться с параметрами поиска.
Было найдено 3418 последовательностей, из них 2779 принадлежат бактериям, 183 - археям, 455 - эукариотам и 1 - вирусу Acanthamoeba polyphaga mimivirus.
Для лучшей, худшей находок и находки из середины списка в Таблицу 1 были занесены параметры выравнивания:
- Длина выравнивания
- Bit score - характеристика, показывающая, насколько сильно последователность совпадает с Query (чем выше, тем "лучше" последовательность)
- Процент идентичных остатков
- Процент сходных остатков
- E-value - число находок с таким же или лучшим Score в случайном банке (чем меньше, тем "лучше" последовательность)
В качестве лучшей находки был выбран белок Elongation factor Tu из организма Hyperthermus butylicus DSM 5456 (верхняя строка списка), в качестве худшей - SPBc2 prophage-derived uncharacterized protein YorJ [Bacillus subtilis subsp. subtilis str. 168] (нижняя строчка), в качестве находки из середины списка - Ribosomal back-translocase LepA [Xanthobacter autotrophicus Py2]
| Таблица 1. Сравнения последовательностей, схожих с белком YP_003816619.1, найденных программой BLAST | |||||||
|---|---|---|---|---|---|---|---|
| Находка | Название белка | Организм | Длина выравнивания | Bit score | Процент идентичных остатков, % | Процент сходных остатков, % | E-value |
| Лучшая | Elongation factor Tu | Hyperthermus butylicus DSM 5456 | 435 | 712 | 78 | 88 | 0.0 |
| Из середины списка | Ribosomal back-translocase LepA | Xanthobacter autotrophicus Py2 | 242 | 73.9 | 28 | 43 | 7е-13 |
| Худшая | prophage-derived uncharacterized protein YorJ | Bacillus subtilis subsp. subtilis str. 168 | 369 | 32.3 | 24 | 47 | 8.6 |
Ниже (Рис. 1, Рис. 2, Рис. 3) представлены выравнивания исходной последовательности с приведенными в таблице.
![Выравнивание последовательности белка Elongation factor Tu[Hyperthermus butylicus DSM 5456] с исходной](pic2.PNG)
Рис. 1. Выравнивание последовательности белка Elongation factor Tu[Hyperthermus butylicus DSM 5456] с исходной
![Выравнивание последовательности белка Ribosomal back-translocase LepA [Xanthobacter autotrophicus Py2] с исходной](pic1.PNG)
Рис. 2. Выравнивание последовательности белка Ribosomal back-translocase LepA [Xanthobacter autotrophicus Py2] с исходной
![Выравнивание последовательности белка SPBc2 prophage-derived uncharacterized protein YorJ [Bacillus subtilis subsp. subtilis str. 168] с исходной](pic3.PNG)
Рис. 3. Выравнивание последовательности белка SPBc2 prophage-derived uncharacterized protein YorJ [Bacillus subtilis subsp. subtilis str. 168] с исходной
Гомологами исходной последовательности из всех находок можно считать 1181 последовательность. По условному критерию последовательность
можно считать гомологичной, если E-value < 1e-3 и не менее 70% запроса вошло в полученное выравнивание (Query cover).
На Рис. 4 представлено графическое изображение результатов поиска. Показано всего 100 последовательностей, максимальное значение E-value
установлено на 1e-3, однако отображенные
находки имеют гораздо меньшее E-value, т. к. в графическое представление вошло 100 первых находок из списка, которые имеют наименьшее E-value)
Рис. 4. Графическое представление результатов поиска. Показано 100 находок, значение E-value не более 1е-3.
Задание 2.
В этом задании были найдены последовательности в организме человека, схожие с исходной.
Для этого в параметрах в поле Organisms было указано "human" (параметры поиска).
Всего было найдено 25 последовательностей. Среди находок была выбрана последовательность HBS1-like protein, которая также была найдена
при первоначальном запуске BLAST (для первоначального результата с помощью окна Formatting options были отобраны находки, относящиеся
к человеку (Organism: human), там была найдена искомая последовательность.
О том, что это та же самая находка, свидетельствуют одинаковые названия, длина последовательностей (Length: 684) и одинаковые ID (sp|Q9Y450.1|HBS1L_HUMAN).
Изменилось только значение E-value: при первом запросе оно равнялось 2e-117, при втором (с указанием организма) - 1e-118.
Это можно объяснить тем, что E-value показывает число находок с таким или большим Score в случайном банке, а так как первый банк больше,
то вероятность найти в нем подходящую последовательность будет больше.
Задание 3.
Для последовательности HBS1-like protein из организма человека было выполнено выравнивание (параметры поиска) и получена локальная карта сходства (Рис. 5).
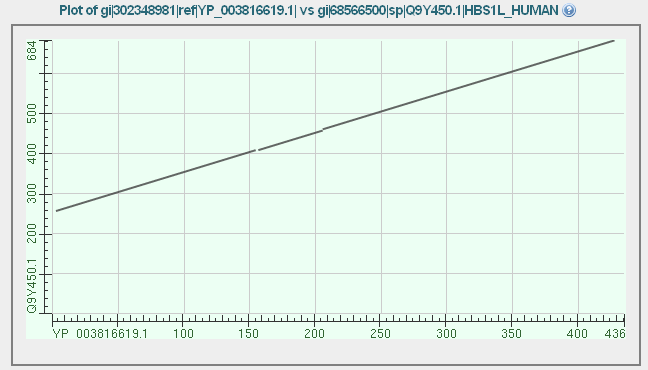
Рис. 5. Локальная карта сходства последовательности HBS1-like protein и исходной последовательности.
Как видно из Рис. 5, последовательности совпадают практически на всем протяжении, во всем выравнивании присутствует всего 2 гэпа, соответствующие инделям (пробелы на графике).
Задание 4.
Для выполнения данного задания была создана база данных, полученная из выравнивания последовательностей, рассмотренными в практикуме 8
(ссылка на fasta-файл), из которого предварительно были удалены все гэпы (ссылка на fasta-файл).
Для этого на сервере kodomo была запущена команда makeblastdb с параметром -dbtype prot. Так была получена база данных с названием lib,
сожержащая 8 последовательностей.
Затем был совершен поиск последовательностей, схожих с белком Elongation factor 1-alpha (ссылка на fasta-файл). Всего программа признала значимыми 5 находок: участки последовательностей BUTPB, DESOD, ENTFO, ROSHA, LISML. Для лучшей находки (BUTPB) в Таблице 2 перечислены основные характеристики: длина выравнивания, % идентичных и сходных остатков, bit score и E-value. Выравнивание представлено на Рис. 6.
| Таблица 2. Основные характеристики находок | |||||
|---|---|---|---|---|---|
| Находка | Длина выравнивания | Bit score | Процент идентичных остатков, % | Процент сходных остатков, % | E-value |
| BUTPB | 8 | 16.9 | 75 | 88 | 1.7 |
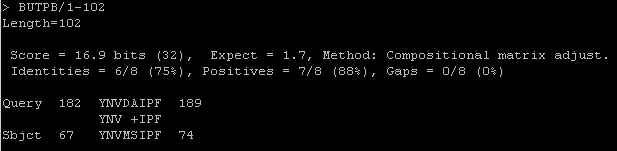
Рис. 6. Выравнивание последовательности белка Elongation factor 1-alpha археи Acidilobus saccharovorans 345-15 c последовательностью BUTPB из новой базы данных.
Матрица и штрафы за гэпы - параметры выравнивания по новой базе данных - совпадают с предыдущими запусками, нопоявились 2 новых параметра:
Neighboring words threshold: 11
Window for multiple hits: 40
Длина выравниваний значительно меньше, чем при предыдущих запусках программы BLASTP, Bit score довольно мал, но процент идентичных и
сходных колонок примерно такой же, т.к. полученные выравнивания слишком коротки. Значение E-value для новой базы данных очень велико
(минимум - 1.7, максимум - 7.6). E-value слишком большой, а Score маленький, поэтому можно судить об отсутствии гомологии и считать, что
полученные совпадения случайны.