Прочтение последовательностей по Сэнгеру
Задание 1.
В данном задании требовалось по результатам хроматограмм из капиллярного секвенатора по Сэнгеру и последовательности, сгенерированной программой, получить
последовательность фрагмента ДНК. Для выполнения задания были использованы данные из файлов с прямой
и обратной цепями ДНК. Скачать эти файлы с расширением .ab можно, нажав на выделенные слова.
Для работы была использована программа Chromas Lite.
Для выравнивания цепей сперва нужно было создать цепь, комплементарную обратной, при помощи опции reverse complement.
Нечитаемые участки:
- 5'-конец прямой цепи: 1-23, т.к. с 24-го нуклеотида уже видны хорошие пики
- 3'-конец прямой цепи: 381
- 5'-конец цепи, комплементароной к обратной: нуклеотидов нет на прямой цепи
- 3'-конец цепи, комплементароной к обратной: 364-379
После удаления участков координаты изменились, так что приведенные выше соответствуют только исходным цепям.
Характеристика качества хроматограммы прямой цепи:
- Сигнал превышает шум в среднем примерно в 7-9 раз
- Распределение уровня сигнала в целом равномерно, шум же распределен неравномерно, т.к. в некоторых местах его почти нет, а в некоторых он мешает точно определить сингал
- Различия в уровнях сигналов могут достигать 3-х раз, при этом уровни сигналов аденина и гуанина выше, чем тимина и цитозина
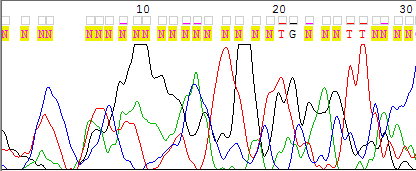
- особенностью данной хроматограммы является наличие широких кратных пиков в начале, что проиллюстрировано на Рис.1.
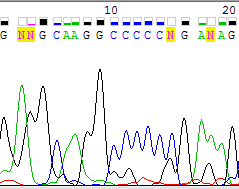
Рис. 1. Кратные пики в хроматограмме
Характеристика качества хроматограммы цепи, комплементароной к обратной:
- Уровень сигнала выше уровня шума в среднем примерно в 7-9 раз, так же, как и на предыдущей хроматограмме
- Распределение уровней сигнала менее равномерно, чем в предыдущей хроматограмме, ближе к концу цепи уровень сигнала сначала сильно возрос, а затем стал снижаться снова (Рис. 2)
- Уровни различных сигналов могут различаться в 2,5-3 раза, причем уровни тимина и цитозина выше, чем аденина и гуанина
- Кроме широких пиков в конце хроматограммы, похожих на широкие пики в начале предыдущей, примерно на 275-277 нуклеотидах на данной хроматограмме происходит сильное наложение сигнала, которое похоже на пятно краски и скорее всего является ошибкой программы (Рис. 3)
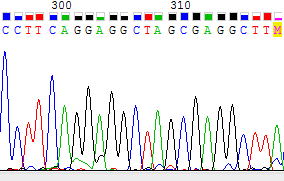
Рис. 2. Изменение уровней сигнала в хроматограмме |
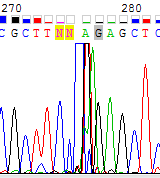
Рис. 3. Сильное наложение пиков |
Редактирование последовательности
Далее будет приведен список некоторых из проблем, с которыми пришлось столкнуться при редактировании последовательности.
Проблема 1. Наложение пиков. Из-за наложения пиков аденина и гуанина друг на друга в прямой цепи (Рис. 4) программа не смогла определить нуклеотид, но на цепи,
комплементарной обратной, в этом месте пики читались однозначно, что позволило определить нуклеотиды на прямой цепи.
Проблема 2. Вместо нескольких пиков - один широкий. На участке хроматограммы, представленном на Рис. 4, видно, что на прямой цепи вместо нескольких пиков
цитозина стоит один широкий. К тому же, уровень шума в конце этого широкого пика был значительно выше среднего, что также помешало программе прочесть
нуклеотид. Но при взгляде на вторую хроматограмму четко видны 6 отдельных подряд идущих пиков цитозина. Это позволило заменить "N" на "c" в этом месте.
Проблема 3. Два нуклеотида подряд нечитаемы. Из-за наложения пиков, о котором говорится в обсуждении проблемы 1, и высокого уровня шума на прямой цепи программа не смогла
прочитать 2 нуклеотида подряд. на цепи, комплементарной обратной, в этом месте проблем нет и видны 2 отдельных пика аденина и гуанина, поэтому "NN" было заменено
на "аg".
Участок, представленный на Рис. 4, оказался "богатым на проблемы", поэтому мне показалось интересным использовать для иллюстрации проблем, описанных выше, именно его. На всех рисунках, содержащих участки 2-х хроматограмм, сверху изображена хроматограмма прямой цепи, снизу - обратной комплиментарной.
Проблема 4. Слишком сильный шум. Из-за того, что уровень шума прямой цепи сильно выше среднего и почти достигает уровня сигнала, программа не смогла прочесть 2 нуклеотида подряд. На цепи, комплементарной обратной, такой проблемы нет, поэтому можно определить, что это за нуклеотиды. Иллюстрация поблемы приведена на Рис. 5.
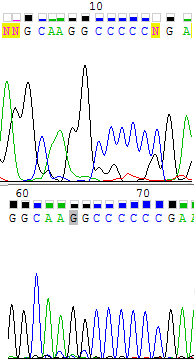
Рис. 4. Иллюстрация проблем 1-3. На позициях 2-3 прямой цепи видно 2 непрочитанных нуклеотида подряд и наложение пиков на хроматограмме (3 и 1 проблемы). Hа позициях 10-15 показан широкий кратный пик цитозина; на позиции 15 виден непрочитанный нуклеотид (проблема 2). |
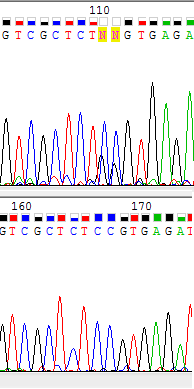
Рис. 5. Проблема 4. Высокий уровень шума |
Участок без проблем. А есть и участки, на которых никаких проблем нет (Рис. 6).
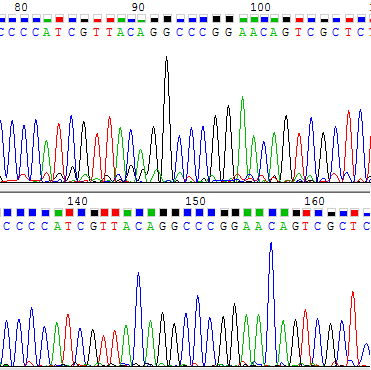
Рис. 6. Беспроблемный участок
После замены нуклеотидов в прямой цепи осталось 5 буквы "N". Нуклеотид на позиции 231 не удалось определить, т.к. он не прочитался в обеих
хроматограммах. Остальные 4 (позиции 321, 324, 330, 354) не удалось заменить из-за того, что они отсутствовали в последовательности, комплементарной обратной.
На позиции 231 было решено поставить y, т.к. там мог стоять тимин или цитозин; на позиции 321, 324, 330 - также y, на позицию 354 - k, т.к. там
могл стоять гуанин или тимин. Все замены нуклеотидов обозначены маленькими буквами.
Ссылки на отредактированные последовательности (все замены произведены маленькими буквами): прямая цепь и комплементарная обратной
цепь.
Далее в программе JalView было проведено выравнивание двух отредактированных последовательностей. Выравнивание было сделано вручную, чтобы маленькие буквы, отмечающие редактирование последовательностей ,не превратились в большие. Результат выравнивания представлен на Рис. 7.

Рис. 7. Выравнивание 2-х цепей с раскраской по нуклеотидам
Итоговые результаты задания: ссылка на "чистую" консенсусную последовательность и ссылка на проект JalView.
Задание 2.
Для примера нечитаемой хроматограммы был взят этот файл. На участке этой хроматограммы, представленном на Рис. 8, видны пятна краски, пиков слишком много и шум настолько силен, что его не удается отличить от сигнала. Причиной этого может служить ошибка программы либо загрязненность используемого образца.