Гены эукариот
Задание 1.
В этом задании нужно было предсказать гены X5 с помощью AUGUSTUS, при этом выбрав один из контигов из сборки Х5 длиной от 20 kb до 100 kb. Для получения
длин контигов в сборке была использована команда infoseq X5.fasta -outfile con.fasta -only -name -snucleotide1 -length пакета EMBOSS
(ссылка на файл).
Для работы был выбран контиг unplaced-982 с длиной 23575 п.н., его нуклеотидная последовательность была получена при
помощи команды seqret X5.fasta:unplaced-982 unplaced-982.fasta была получена fasta-последовательность этого
контига.
Далее требовалось предсказать гены с помощью AUGUSTUS.
Для начала нужно было выбрать организм, параметры генома которого будут взяты для предсказания генов, либо проведя "обучение" (training) программы на
хороших примерах (используя гомологи хорошо изученных генов или анализируя транскриптом, если таковой имеется), либо использовав геном ближайшего организма,
для которого уже было ранее выполнено предсказание генов. В данной работе был выбран второй вариант. Последовательность контига была отправлена в blastx для
нахождения родственных организмов и проведения таксономического анализа.
Для облегчения поиска в некоторые параметры были внесены изменения: максимальное количество находок было уменьшено до 50 (ведь в любом случае для работы
потребуются только сведения о лучших находках), а область поиска была ограничена эукариотами. На Рис. 1 представлен результат выдачи blast.
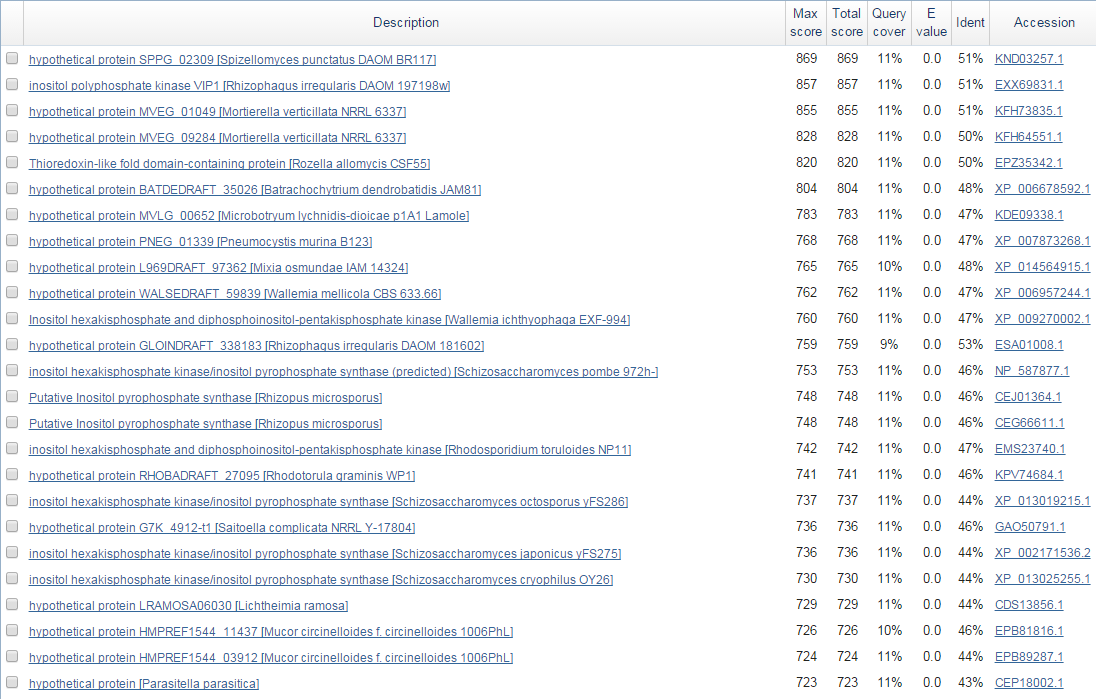
Рис. 1. Выдача blastx при поиске находок для контига unplaced-982
Лучшая находка принадлежит Spizellomyces punctatus DAOM BR117. Однако при просмотре таксономии оказалось, что больше всего находок принадлежит организму Rhizopus microsporus (Рис. 2), поэтому несмотря на то, что эта находка не являлась лучшей, для работы с AUGUSTUS была выбрана она.
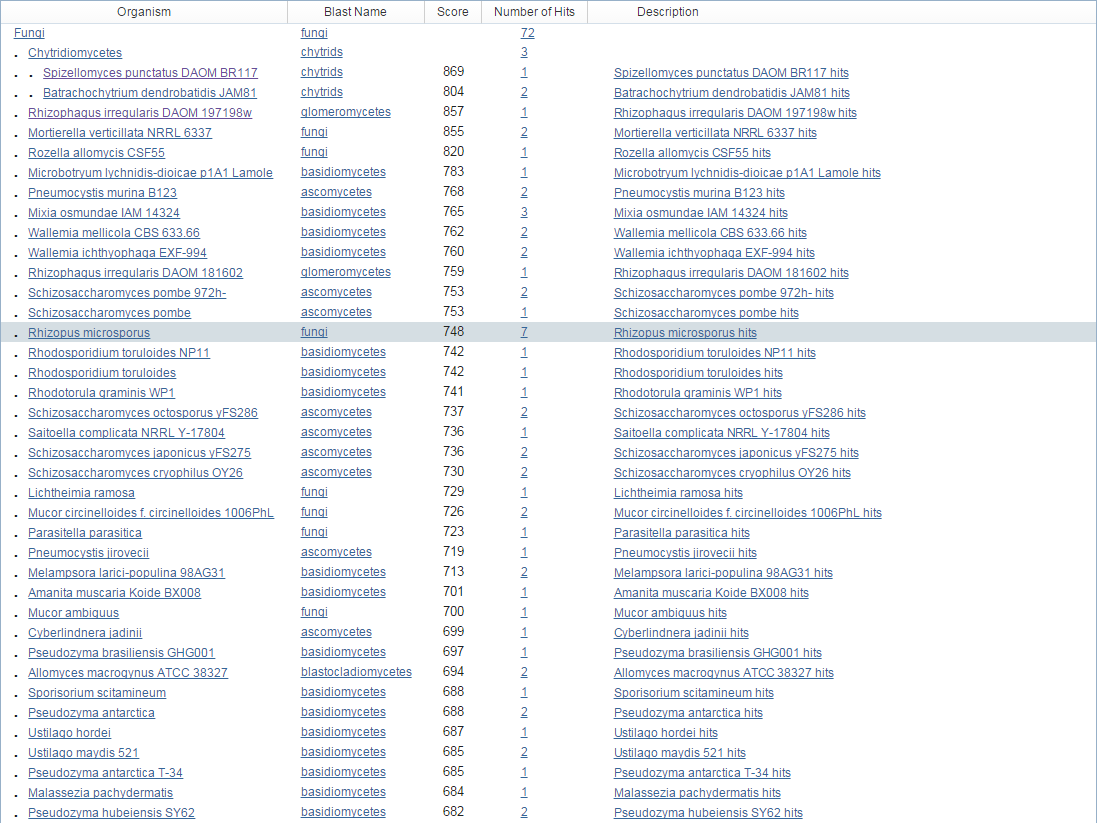
Рис. 2. Таксономия находок blastx
Далее нужно было зайти на сайт AUGUSTUS. Наиболее близким организмом к выбранному оказался Rhizopus oryzae, поэтому он был использован при работе ресурса. AUGUSTUS выдал архив с несколькими файлами как результат (страница с описанием статуса задания).
- augustus.gtf с предсказанием генов в формате .gtf
- augustus.gff с общими результатами (содержит информацию из всех файлов)
- augustus.gbrowse с информацией (координаты в геноме, прямая или обратная цепь) о структурных элементах пре-мРНК, предшествующей каждому из предсказанных генов (предсказание геномного браузера GBrowse)
- augustus.codingseq с кодирующими нуклеотидными последовательностями предсказанных генов целиком (все экзоны одного гена вместе в формате fasta)
- augustus.csdexons с нуклеотидными последовательностями предсказанных экзонов (по отдельности для каждого экзона в формате fasta)
- augustus.aa с аминокислотными последовательностями белков (в формате fasta) - продуктов каждого из предсказанных генов
Далее была проведена проверка предсказания при помощи blast. В файле с расширением .аа находятся последовательности одиннадцати предсказанных генов в fasta-формате. Для проверки были выбраны первые 5 из списка. Предсказанная экзон-интронную структура данных генов была извлечена из файла с расширением .gff и обработана в Excel. Получившаяся таблица доступна по ссылке и представлена на Рис. 3.
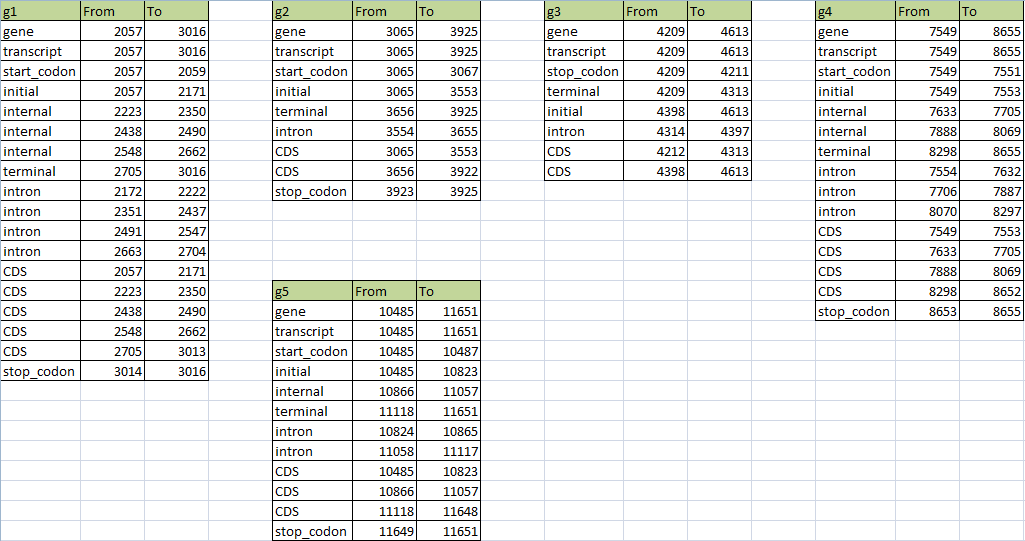
Рис. 3. Предсказанные при помощи AUGUSTUS экзон-интронные структуры генов g1-g5
Поиск был проведен алгоритмом blastp по БД Swissprot, область поиска была ограничена таксоном Fungi.
G1
Для g1 было сделано всего 2 находки (Рис. 4). Обе относятся к белку inositol hexakisphosphate and diphosphoinositol-pentakisphosphate kinase.
Для лучшей находки
есть запись, что он имеет подтверждение на уровне белка (/UniProtKB_evidence="Evidence at protein level")
Функция: Bifunctional inositol kinase that acts in concert with
the IP6K kinases to synthesize the diphosphate group-containing
inositol pyrophosphates diphosphoinositol pentakisphosphate,
PP-InsP5, and bis-diphosphoinositol tetrakisphosphate, (PP)2-InsP4.
Phosphorylates inositol hexakisphosphate (InsP6) at positions 1 or
3 to produce PP-InsP5 which is in turn phosphorylated by IP6Ks to
produce (PP)2-InsP4. Alternatively, phosphorylates at position 1 or
3 PP-InsP5, produced by IP6Ks from InsP6, to produce (PP)2-InsP4
(By similarity). Required for maintaining cellular integrity,
normal growth and interactions with the ARP complex. Acts as a
regulator of the PHO80-PHO85 cyclin/cyclin-dependent kinase (CDK)
complex, thereby regulating signaling of phosphate availability (By
similarity). Required for the function of the cortical actin
cytoskeleton, possibly by participating in correct F-actin
localization and ensuring polarized growth.
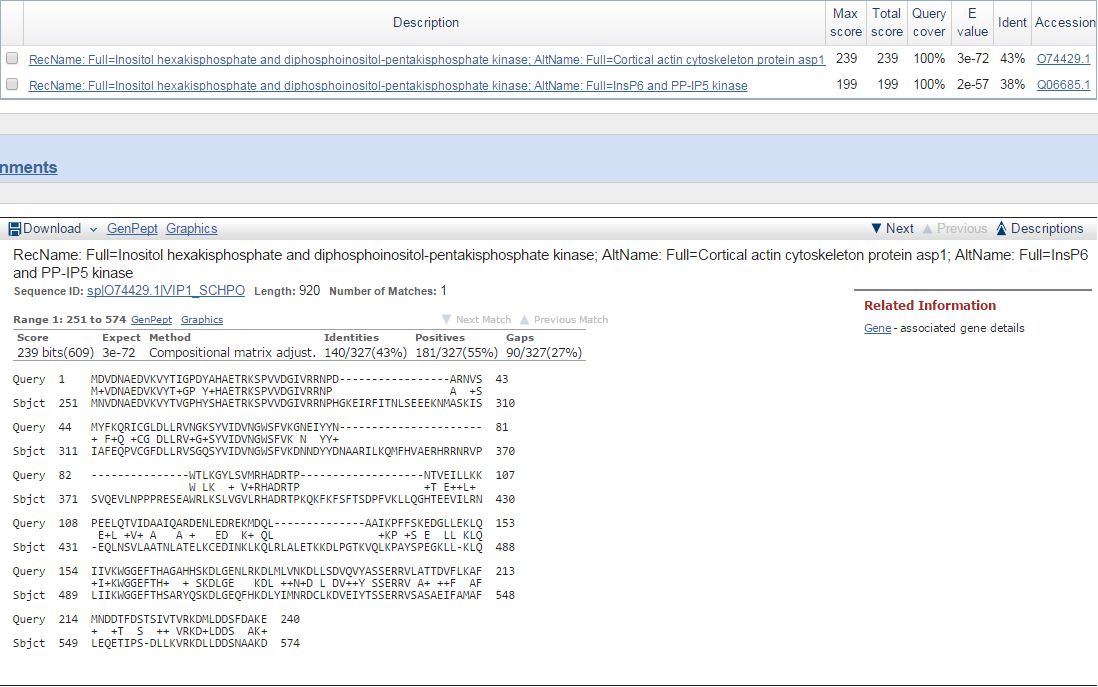
Рис. 4. Находки blastp для гена g1
G2
Удивительно, но для гена g2 blast выдал те же самые находки с немного другими параметрами. Сами находки представлены на Рис. 5. Остальное аналогично написанному выше для g1.
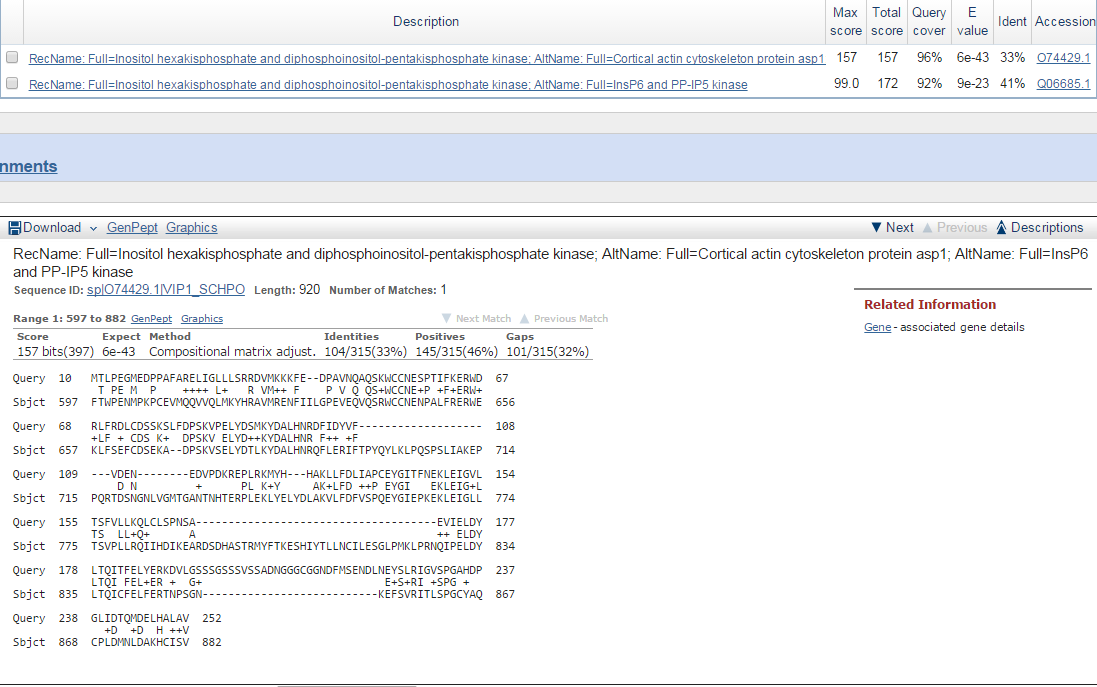
Рис. 5. Находки blastp для гена g2
G3
Для g3 было сделано 5 находок, но скорее всего только одна (лучшая)
может претендовать на достоверность. У 3 из 4 остальных находок слишком большое E-value (у худшей оно равно 5), оставшаяся же уступает лучшей находке в идентичности
входной последовательности и score. На основании этого было заключено .что скорее всего ген g3 кодирует белок transport protein particle subunit trs31
Его функция: TRAPP plays a key role in the late stages of endoplasmic
reticulum to Golgi traffic.
Однако, как оказалось, подтверждение для этой находки есть только на уровне гомологии (/UniProtKB_evidence="Inferred from homology"), поэтому была
рассмотрена вторая находка. Для нее уже
имеется подтверждение на белковом уровне (/UniProtKB_evidence="Evidence at protein level")
Функция: Component of the TRAPP I, TRAPP II and TRAPP III
complexes which act as guanine nucleotide exchange factors (GEF)
for YPT1. TRAPP I plays a key role in the late stages of
endoplasmic reticulum to Golgi traffic. TRAPP II plays a role in
intra-Golgi transport. TRAPP III plays a role in autophagosome
formation.
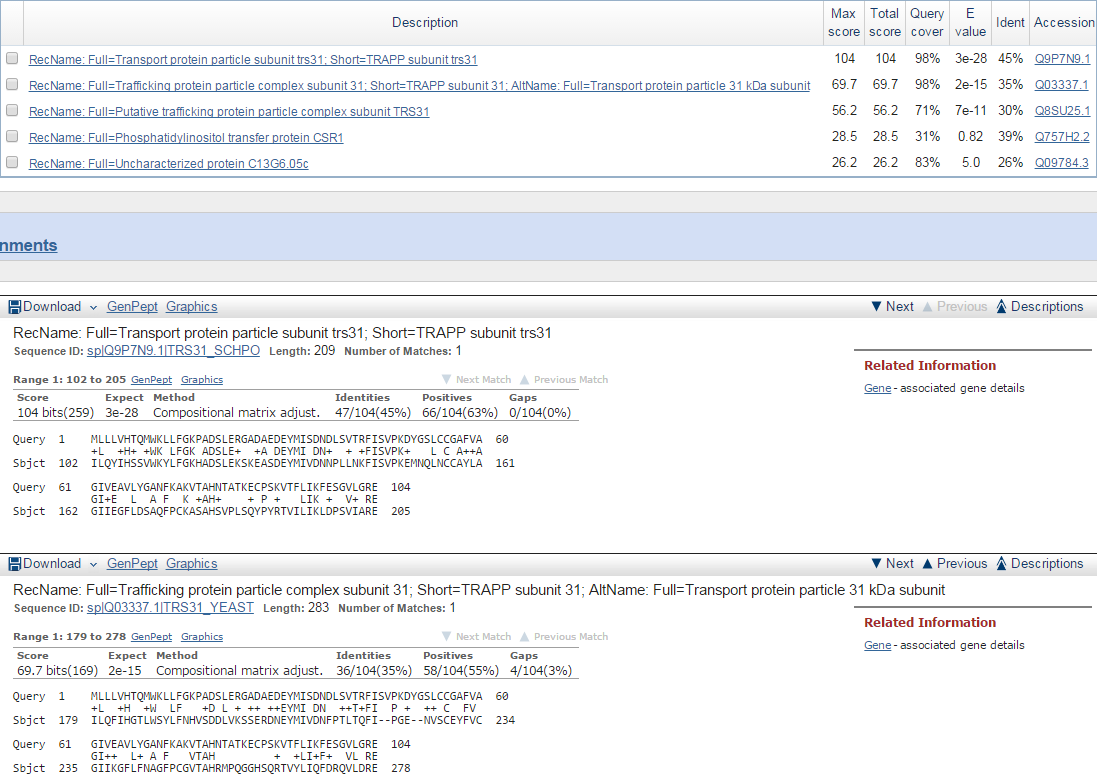
Рис. 6. Находки blastp для гена g3
G4
Для g4 было сделано 143 находки. Все находки являются плохими, но лучшая находка
может претендовать на достоверность, т.к. имеет подтверждение на белковом уровне. Из этого можно заключить, что ген g4 кодирует белок Ribosome assembly protein 4.
Функция: Involved in ribosome biogenesis. Required for processing
and efficient intra-nuclear transport of pre-60S ribosomal
subunits. Interacts with the AAA-ATPase Midasin, which is essential
for the ATP-dependent dissociation of a group of nonribosomal
factors from the pre-60S particle.
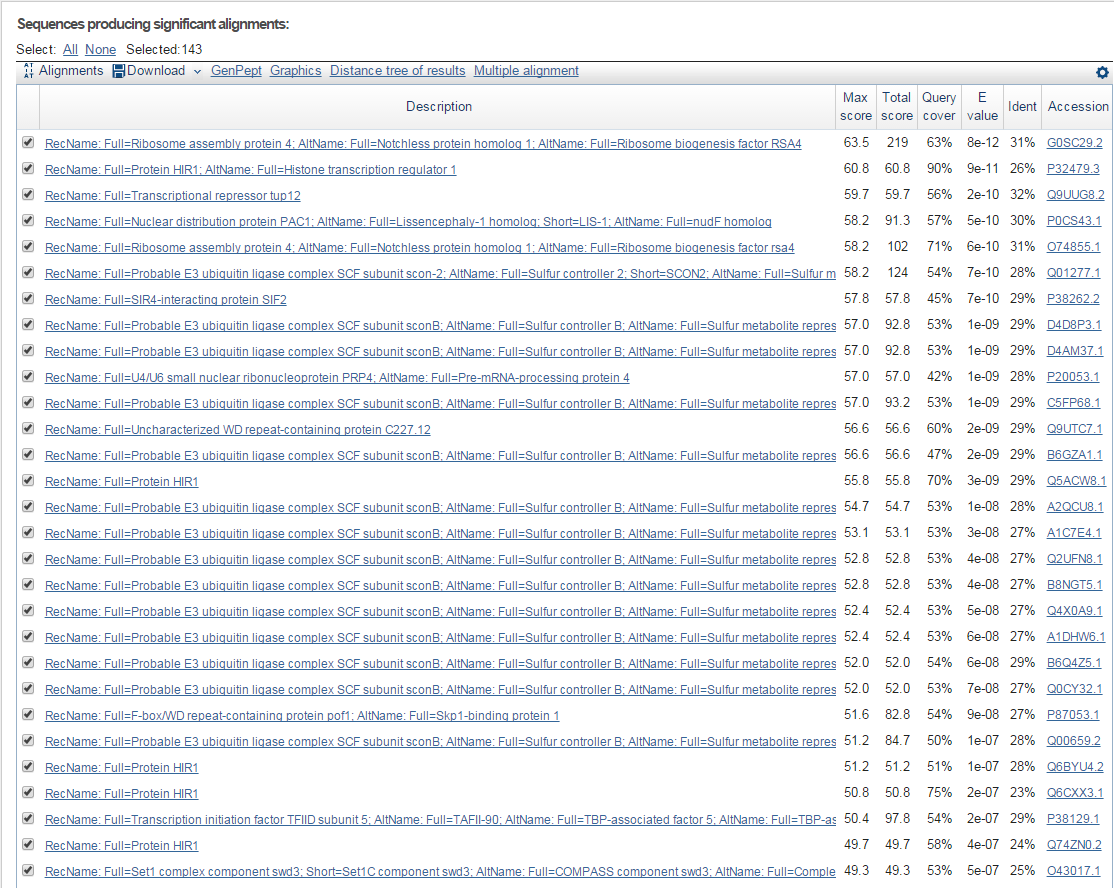
Рис. 7. Находки blastp для гена g4
G5
Для гена g5 было сделано 12 находок. Неожиданным после плохого качества предыдущих находок оказалось то, что у двух лучших находок данного запроса E-value=0.0 и
Query cover=99%. Также можно заметить, что почти все находки относятся к одному и тому же белку - 60S ribosomal protein L3. Это, а также наличие подтверждения
лучшей
находки на белковом уровне, позволяют утверждать, что ген g5 действительно кодирует этот белок.
Его функция: The L3 protein is a component of the large subunit of
cytoplasmic ribosomes.
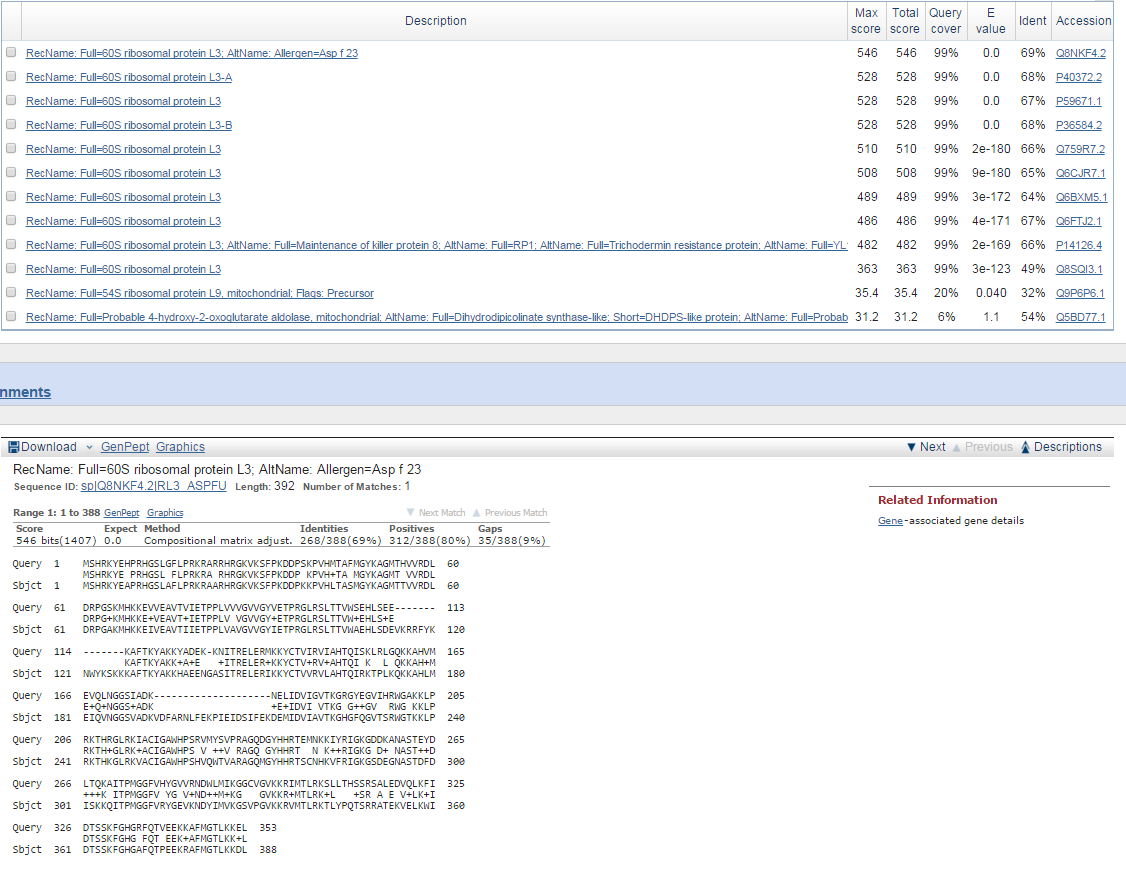
Рис. 8. Находки blastp для гена g5
Задание 2.
В данном задании требовалось сравнить аннотацию Refseq и AUGUSTUS одного гена человека. Для выполнения задания был использован UCSC Genome Browser. В нем была выбрана последняя сборка генома hg38. Для работы был выбран ген ACE (angiotensin I converting enzyme). Этот ген кодирует фермент, вовлеченный в катализ преобразования ангиотензина I в физиологически активный белок ангиотензин II. Ангиотензин II - важный вазопрессорный и альдостерон-стимулирующий белок, контролирующий кровяное давление и водно-солевой баланс.
Координаты гена: хромосома 17, 63477061-63498380 п.н. по версии NCBI, 63477071-63497620 п.н. по версии Genome Browser.
Длина: 21320 п.н. по версии NCBI, 20550 п.н. по версии Genome Browser.
Нужный ген в ресурсе UCSC Genome Browser был найден в последней сборке генома человека при помощи опции Genome Browser. Далее было оставлено только три трека: base position, RefSeq и AUGUSTUS. На Рис. 9 приведен скриншот окна браузера с двумя аннотациями гена ACE.
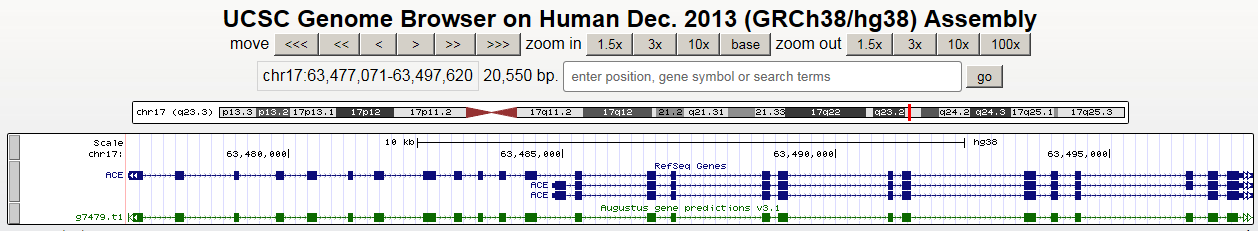
Рис. 9. Скриншот окна браузера с двумя аннотациями гена ACE
Затем были по отдельности получены предсказания экзон-интронной структуры, выполненные RefSeq и AUGUSTUS, в виде таблиц: таблица RefSeq и таблица AUGUSTUS. Также таблицы представлены на Рис. 10-11 соответственно.
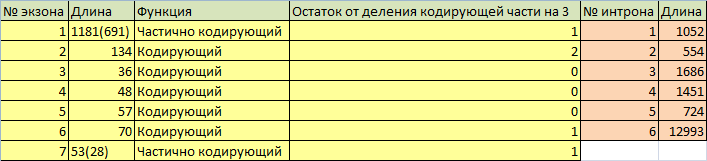
Рис. 10. Экзон-интронная структура гена гена ACE, аннотация RefSeq
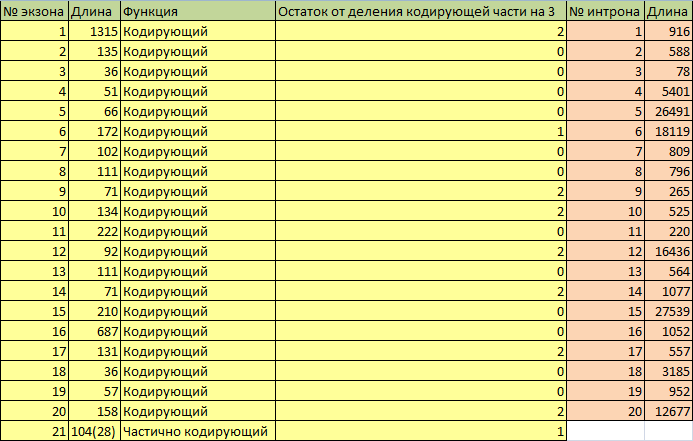
Рис. 11. Экзон-интронная структура гена гена ACE, аннотация AUGUSTUS
Из Рис.10-11 видно, что аннотации сильно различаются. AUGUSTUS нашел в 3 раза больше экзонов, чем RefSeq. Гены в двух аннотациях начинаются и заканчиваются в разном положении.