Выравнивание геномов
Задание 1.
В данном задании требовалось построить карту сходства хромосом двух родственных бактерий. Для работы были взяты геномы Brucella abortus 104M chromosome 1, complete sequence и Brucella suis bv. 2 strain Bs143CITA chromosome I, complete sequence.
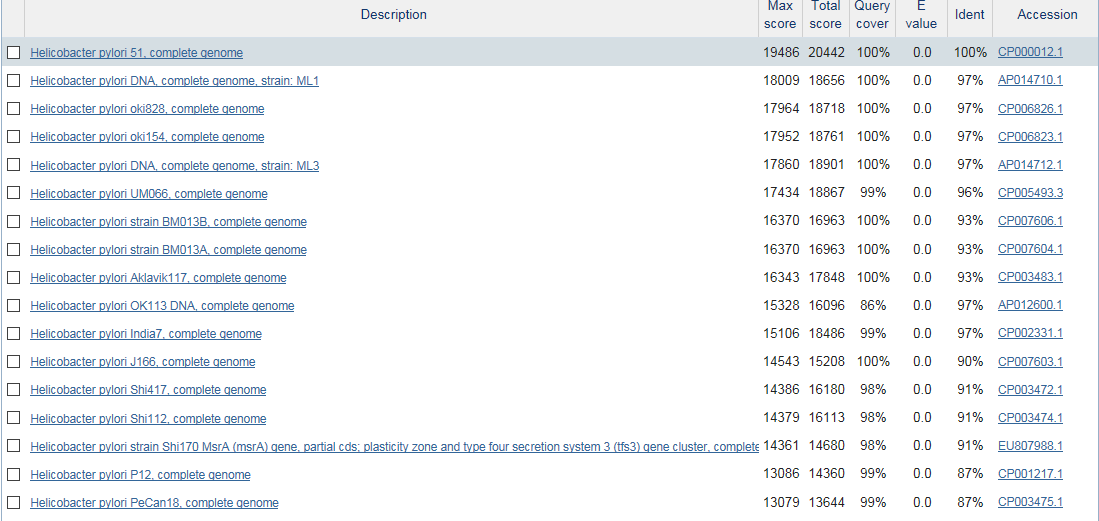
Для сравнения двух геномов было необходимо было запустить blastn для выравнивания 2 последовательностей (blast2seq) с сайта NCBI. Нужным результатом работы ресурса являлась карта локального сходства, представленная на Рис. 1.
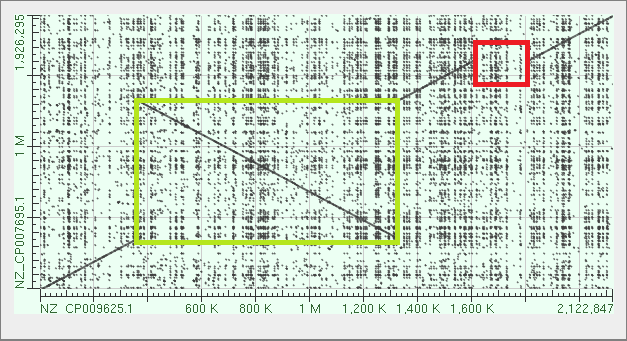
Рис. 1. Карта локального сходства для геномов двух бактерий: Brucella abortus 104M chromosome 1 и Brucella suis bv. 2 strain Bs143CITA chromosome I. Зеленой рамкой обозначено место, где произошла инверсия, красной - место, где произошла вставка (делеция).
Задание 2.
В этом задании надо было описать сходство и различие геномов близкородственных бактерий. При выполнении задания использовался метод построения нуклеотидного пангенома (NPG) с помощью пакета NPG-explorer. Нуклеотидный пангеном - специальный формат для множественного выравнивания геномов, ориентированный на геномы близкородственных бактерий или архей. Для работы были выбраны 3 штамма бактерии Helicobacter pylori: 26695, J99, 51.
Для работы пакета NPG-explorer было необходимо создать файл genomes.tsv, в которых содержится информация об источниках последовательностей
геномных ДНК и аннотаций генов.
Затем при помощи команды npge -g npge.conf был создан файл npge.conf, содержащий параметры программы. Параметр MIN_IDENTITY
был изменен на рекомендованное программой значение 0.877.
Информация о блоках в формате Excel

Рис. 2. Участок выравнивания блоков
Немного о блоках.
- s-блоки - стабильные (коровые) блоки, по одному фрагменту из каждого генома
- h-блоки - "полустабильные" блоки - по одному фрагменту из части геномов
- u-блоки - и не блоки вовсе, а уникальные последовательности из одного генома, у них нет гомологов среди всех геномов, кроме самой себя
- r-блоки - блоки с повторами, по крайней мере, в одном геноме
- m-блоки - минорные блоки - короткие ( Список глобальных блоков - синтений - см. в global-blocks/blocks.gbi g-блоки (глобальные блоки) состоят из последовательно идущих во всех геномах s-блоков, перемежающихся блоками других типов (r-, h-, u- и m-)
Описание g-блоков (синтеничных участков).
Для работы был использован файл global-blocks/blocks.gbi. Всего есть 42 g-блока.
Описание s-блоков (стабильных).
Информация о блоках была взята из файла pangenome/pangenome.info. Число блоков = 517, суммарная длина блоков 1331210 (69.15%), процент от длины генома в среднем для каждого отдельного блока (длина s-блоков / кол-во s-блоков) = 0.19%, сходство геномов (процент консервативных позиций в объединенном выравнивании s-блоков) = 0.92436
Описание повторов на примере r-блоков.
Были использованы данные из файла pangenome/pangenome.bi.
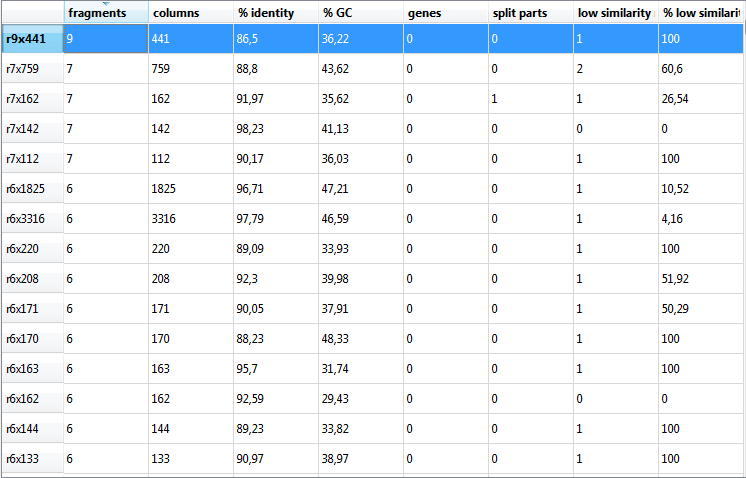
Рис. 3. Таблица r-блоков
Для примера был взят блок r9x441. Видно, что в нем 9 фрагментов и 441 позиция в выравнивании блока. На Рис. 4 представлен участок выравнивания последовательностей, составляющих этот блок.

Рис. 4. Участок выравнивания последовательностей, составляющих блок r9x441
Описание h-блоков.
H-блоки - это "полустабильные" блоки, включающие по одному фрагменту из части геномов. То есть у какого-то из организмов данная последовательность отсутствует. На Рис. 5 представлены 4 самых больших h-блока, соответствующие крупным делециям.
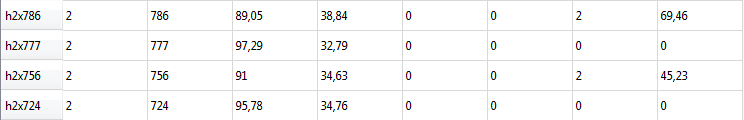
Рис. 5. Самые большие h-блоки
Описание u-блоков.
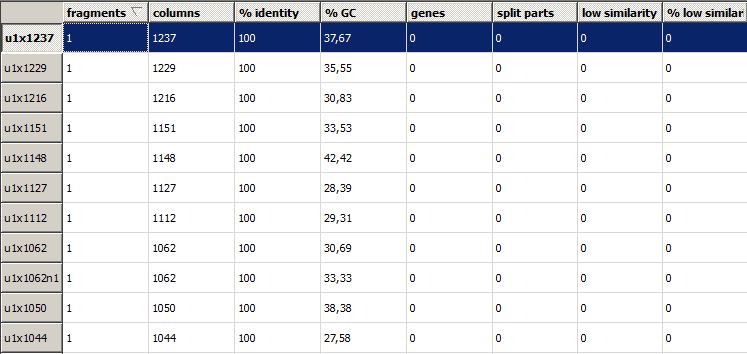
Рис. 6. Примеры уникальных последовательностей
Для аннотирования был выбран самый крупный u-блок: u1x10805. На Рис. 7 представлена выдача blastn для последовательности этого блока. Видно, что этот блок есть у штамма 51 изучаемой бактерии. Все хорошие находки для данной последовательности принадлежали разным штаммам бактерии Helicobacter pylori, поэтому скорее всего последовательность не является результатом горизонтального переноса из бактерий другого вида.