Поиск по сходству (blast)
Задание 1.
Данное задание является Заданием 4 предыдущего практикума. Ссылка на страницу с выполненным заданием: ссылка.
Задание 2.
В данном задании требовалось сравнить списки находок нуклеотидной последовательности 3-я разными алгоритмами blast. Для выполнения задания была взята
последовательность из практикума "Прочтение последовательностей по Сэнгеру" (она же использовалась при выполнении предыдущего практикума).
Поскольку при выполнении Задания 1 (Задания 4 предыдущего практикума) лучшая находка принадлежала виду Glycera capitata, а последним общим таксоном у
5/6 выбранных лучших находок яляется класс Polychaeta, было решено при выполнении задания ограничиться данным таксоном.
Был проведен поиск 3-мя алгоритмами blast: blastn, megablast, discontiguous megablast. Максимальное число находок было увеличено до 10000. В Таблице 1 приведены некоторые данные о находках при поиске по каждому из алгоритмов. Некоторые из находок при 3-х поисках можно увидеть на Рис. 1-3.
| Таблица 1. Сравнение находок при поиске по алгоритмам blastn, megablast, discontiguous megablast | ||||
|---|---|---|---|---|
| Алгоритм | Число находок | E-value худшей находки | Query cover худшей находки, % | Сходство худшей находки, % |
| blastn | 517 | 3е-56 | 85 | 81 |
| megablast | 517 | 2е-56 | 85 | 81 |
| discontiguous megablast | 782 | 5е-18 | 40 | 76 |
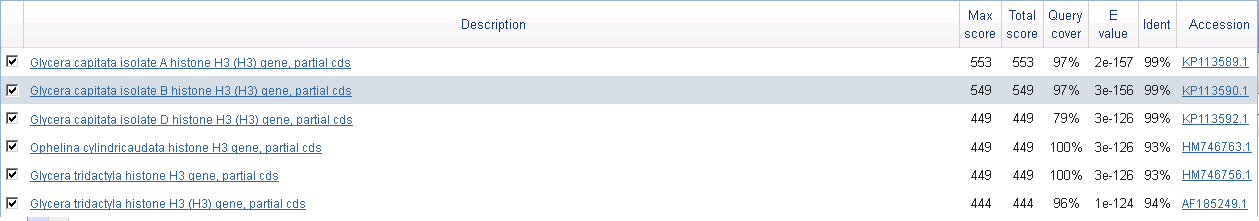
Рис. 1. Некоторые находки при поиске по алгоритму blastn
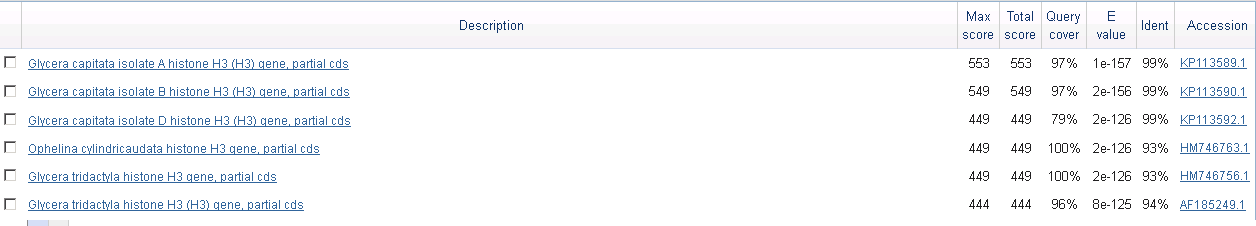
Рис. 2. Некоторые находки при поиске по алгоритму megablast
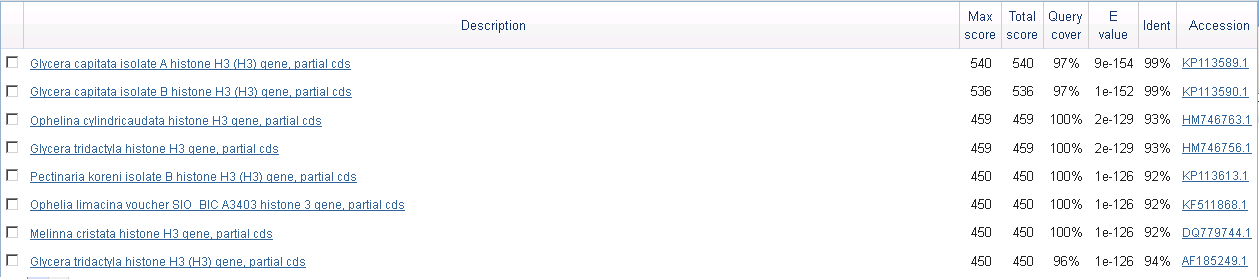
Рис. 3. Некоторые находки при поиске по алгоритму discontiguous megablast
Blastn используется для поиска всех сходных последовательностей, он ищет любые сходные участки. Megablast позволяет найти последовательности,
обладающие очень высоким сходством с исходной. Discontiguous megablast нужен для поиска дивергированных последовательностей, возможно являющихся
близкими гомологами. Megablast ищет сходные последовательности по паттерну в 28 нуклеотидов, а другие 2 алгоритма - по паттерну в 11 нуклеотидов,
поэтому у megablast выше вероятность попадания замены.
В находках blastn и megablast было выбрано по 6 лучших находок, которые оказались одинаковыми. Как видно из Таблицы 1, число находок и параметры
худших находок у этих 2-х алгоритмов одинаковы, за исключением E-value. Больше всего находок получилось при поиске по алгоритму discontiguous
megablast, что странно, т.к. исходя из сути этих 3-х алгоритмов меньше всег онаходок должен был сделать megablast, а больше всех - blastn.
Задание 3.1.
В этом задании требовалось проверить наличие гомологов пяти белков в геноме одного из организмов. Для работы был взят организм, использовавшийся в предыдущем практикуме: беркут (Aquila chrysaetos). Проверяемые белки: HSP7C_HUMAN, TERT_HUMAN, CISY_HUMAN, RPB1_HUMAN, PABP2_HUMAN.
HSP7C_HUMAN: Heat shock cognate 71 kDa protein. AC - P11142. Последовательность в формате fasta.
Действует как репрессор транскрипционной активации. Ингибирует
транскрипционную коактиваторную активность CITED1 при Smad-медиированной транскрипции. Шаперон. Компонент комплекса PRP19-CDC5, формирующего неотъемлемую часть
сплайсингсомы и обязательного для активации pre-mRNA сплайсинга. Может играть роль скэффолда в сплайсингосомной сборке, т.к. контактирует со всеми другими
компонентами ядерного комплекса. Связывает бактериальные липополисахариды (LPS), медиирующие LPS-индуцированный воспалительный ответ, включающий выработку TNF
моноцитами. Участвует в процессе контроля качества ER-ассоциированной деградации (ERAD) вместе с содержащими домен J ко-шаперонами и Е3 лигазой CHIP.
TERT_HUMAN: Telomerase reverse transcriptase. AC - O14746. Последовательность в формате fasta.
Теломераза - рибонуклеопротеиновый фермент, необходимый для репликации хромосомных сайтов терминации
транскрипции у большинства эукариот. Активный в клетках-предшественниках и опухолевцх клетках. Неактивный или проявляющий очень низкую активность в нормальных
соматических клетках. Катализирующий компонент теломеразного холоферментного комплекса, чья основная деятельность заключается в элонгации теломеров, действуя как
обратная транскриптаза, которая добавляет элементарные повторяющиеся последовательности в конец хромосомы копированием матричной последовательности компонента РНК
фермента. Катализирует РНК-зависимую достройку 3'-хромосомных сайтов терминации транскрипции 6-нуклеотидными теломерными единицами повтора, 5'-TTAGGG-3'.
Каталитический цикл включает в себя связывание праймера, достройку праймера и отщепление продукта, как только достигается граница матрицы, либо транслокация
появляющегося продукта, за которой следует дальнейшая достройка. Более активен на субстратах, содержащих 2 или 3 теломерных повтора. Активность
теломеразы регулируется некоторым количеством факторов, включающим ассоциированные с теломеразным комплексом белки, шапероны и полипептидные
модификаторы. Модулирует подачу сигналов Wnt. Играет важную роль в старении и антиапоптозе.
Каталитическая активность: Deoxynucleoside triphosphate + DNA(n) = diphosphate + DNA(n+1).
CISY_HUMAN: Citrate synthase, mitochondrial protein. AC - O75390. Последовательность в формате fasta.
Каталитическая активность: Acetyl-CoA + H2O + oxaloacetate = citrate + CoA.
Цикл Кребса: этот белок вовлечен в 1-ый этап субпроцесса, синтезирующего изоцитрат из оксалоацетата. Белки, которые, как известно, вовлечены в
2 этипа этого субпроцесса в организме:
1-ый этап. Citrate synthase, mitochondrial (CS)
2-ой этап. Aconitate hydratase, mitochondrial (ACO2)
Этот субпроцесс является частью цикла Кребса, который, в свою очередь, является частью углеводного обмена веществ
RPB1_HUMAN: DNA-directed RNA polymerase II subunit RPB1. AC - P24928. Последовательность в формате fasta.
ДНК-зависимая РНК полимераза катализирует транскрипцию ДНК в РНК, используя 4 рибонуклеозидных
трифосфата как субстраты. Наибольший каталитический компонент РНК полимеразы II синтезирует предшественников мРНК и много функциональных некодирующих РНК.
Формирует полимеразный активный центр со второй наибольшей субъединицей. Является центральным компонентом основного механизма транскрипции РНК полимеразы II.
Состоит из мобильных элементов, перемещающихся относительно друг друга.
Каталитическая активность: Nucleoside triphosphate + RNA(n) = diphosphate + RNA(n+1).
PABP2_HUMAN: Polyadenylate-binding protein 2. AC - Q86U42. Последовательность в формате fasta.
Вовлечен в 3'-концевое формирование pre-mRNA добавлением хвоста poly(A) из 200-250нк к расположенному
против хода транскрипции продукта расщепления. Стимулирует poly(A) полимеразу, придавая процессивность реакции элонгации poly(A) хвоста и контролируя его длину.
Повышает сродство poly(A) полимеразы к РНК. Взаимодействует со SKIP, чтобы синергически активировать E-box-медиированную транскрипцию на основе MYOD1, и может
регулировать экспрессию специфических для мышц генов. Связывается с poly(A) и poly(G) с высокой степенью сродства. Может защищать poly(А) хвост от разрушения.
Для поиска находок использовался tblastn, поиск велся по БД Refseq, область поиска была ограничена данным организмом (Aquila chrysaetos).Сведения о находках представлены в Таблице 2.
| Таблица 2. Находки tblastn | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Белок | Число находок (хорошие/все) | Лучшая находка | Query cover лучшей находки, % | E-value лучшей находки | Идентичность лучшей находки, % | Ссылка на выдачу blast | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| HSP7C_HUMAN | 2/10 | Scaffold91 | 94 | 0.0 | 83 | Ссылка | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| TERT_HUMAN | 1/2 | Scaffold88 | 69 | 7e-38 | 41 | Ссылка | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| CISY_HUMAN | 1/2 | Scaffold161 | 97 | 2e-72 | 70 | Ссылка | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| RPB1_HUMAN | 1/6 | Scaffold271 | 76 | 1e-50 | 94 | Ссылка | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| PABP2_HUMAN | 1/5 | Scaffold61 | 39 | 3e-25 | 81 | Ссылка | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
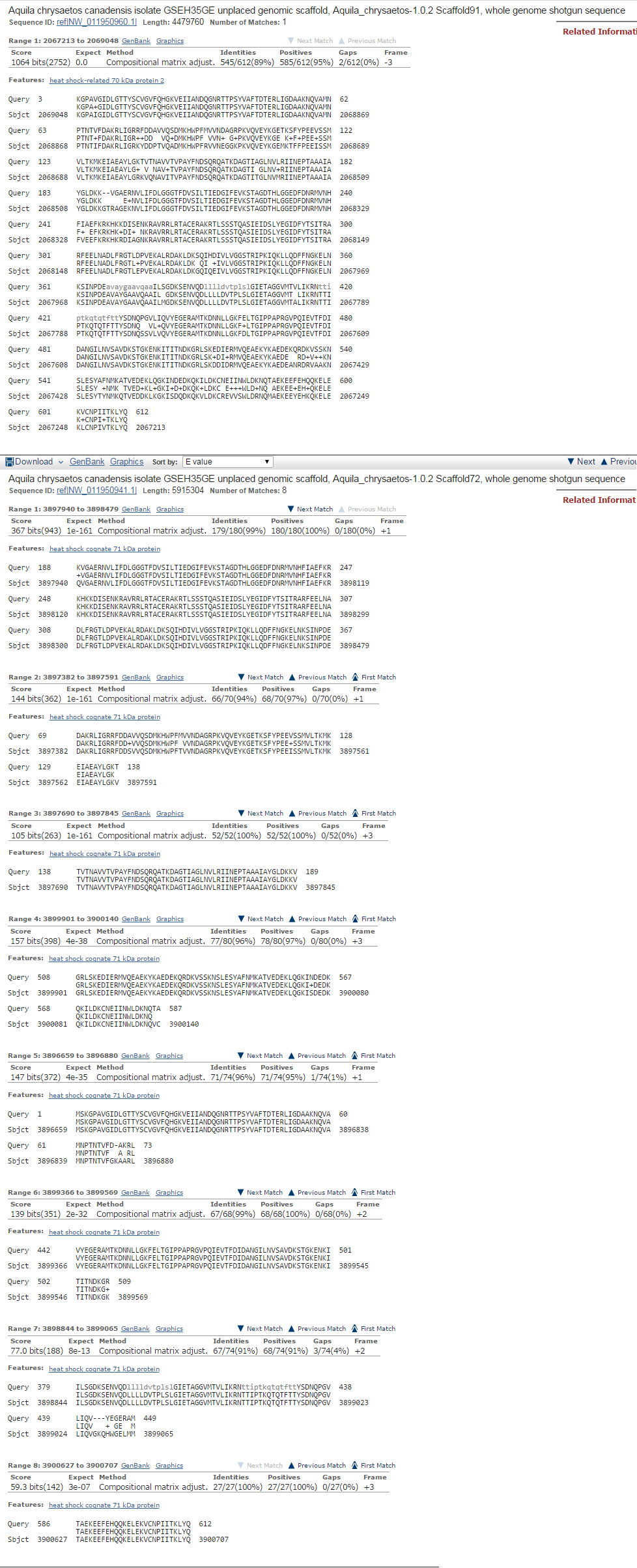
"Хорошими" при поиске по возможности считались находки с идентичностью более 80%. Однако для TERT_HUMAN и CISY_HUMAN таких не нашлось, поэтому было решено взять за "хорошую" лучшую находку (в обоих случаях было всего 2 находки, и вторая была значительно хуже первой). В ссылках на выдачу blast написано большее количество находок, чем в таблице, т.к. за отдельные находки считались различные участки сходства для одной и той же последовательности. Что интересно, для последователности белка HSP7C_HUMAN blast выдал 2 "хорошие" находки, причем иднетичность второй была выше, чем у первой (99% против 83%). Но, поскольку E-value второй "хорошей" был ниже (1e-161), было решено внести в таблицу параметры первой находки. Параметры обеих находок для сравнения можно посмотреть на Рис. 4.

Рис. 4. "Хорошие" находки для последовательности белка HSP7C_HUMAN
То, что для HSP7C_HUMAN было найдено 2 предполагаемых гомолога, может объясняться тем, что из всех найденных скэффолдов данные 2 (91 и 72) наиболее близки друг к другу. К тому же, для первой находки (скэффолд 91) blast выдал один большой участок сходства, а для второй - восемь маленьких. Участки сходства обеих для обеих находок можно посмотреть на Рис. 5.