A- и В- формы ДНК. Структура РНК
|
1. Запустить скрипт: 2. Продолжить исполнение скрипта: |
Задание 1
Для выполнения задания требовалось построить А-, В- и Z-формы ДНК с помощью программы fiber пакета 3DNA (пакета программ для анализа и простейшего
моделирования структур нуклеиновых кислот). Данный пакет программ работает под операционной системой Linux, поэтому для работы с ним было необходимо
использовать программу Putty. Были построены А- и В-формы дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз повторенную
последовательность "gatc". Программа fiber позволяет только Z-форму ДНК, представленную 5 раз повторенной парой G-C, поэтому построить Z-форму ДНК с
базовой последовательностью "gatc" не представлялось возможным.
Результаты выполнения задания представлены файлами с полученными структурами форм ДНК в формате .pdb: А-форма,
B-форма, Z-форма.
Задание 2
Упражнение 1
В данном упражнении целью было научиться выделять разные атомы и химические группировки, используя предопределенные множества JMol. Работа велась с А-формой ДНК, построенной в Задании 1. При выполнении задания для большей наглядности был использован Jmol-апплет, поcледовательно выделяющий следующие структуры:
- сахарофосфатный остов ДНК (выделен зеленым цветом, моделью backbone);
- все нуклеотиды( А выделен красным цветом, Т - желтым, G - зеленым, с - синим, все изображены в cartoon);
- все нуклеотиды, содержащие основание аденин (выделены зеленым и моделью wireframe);
- атом N7 во всех гуанинах и только в первом по последовательности (все атомы крупного размера, все, кроме первого, покрашены красным, первый - желтым)
Упражнение 2
В данном упражнении требовалось получить файлы PDB структур т-РНК и ДНК-белкового комплекса с сайта PDB и отобрать из предложенных вариантов структуры подходящую. Результаты: полученные файлы со структурами тРНК (Glutamyl-tRNA synthetase from Thermus thermophilus in complex with tRNA(Glu) and L-glutamate) и ДНК-белкового комплекса (Crystal structure of restriction endonuclease BGLII complexed with DNA 16-mer)
Упражнение 3
В данном упражнении требовалось проверить полученные структуры ДНК и РНК на наличие разрывов. Для этого нужно было рассмотреть проволочные модели (backbone) только нуклеиновых кислот из файлов, полученных с сайта PDB. Ни в тРНК, ни в ДНК разрывов обнаружено не было.

Рис. 1.Изображение тРНК из Thermus thermophilus. Изображение получено с помощью Jmol
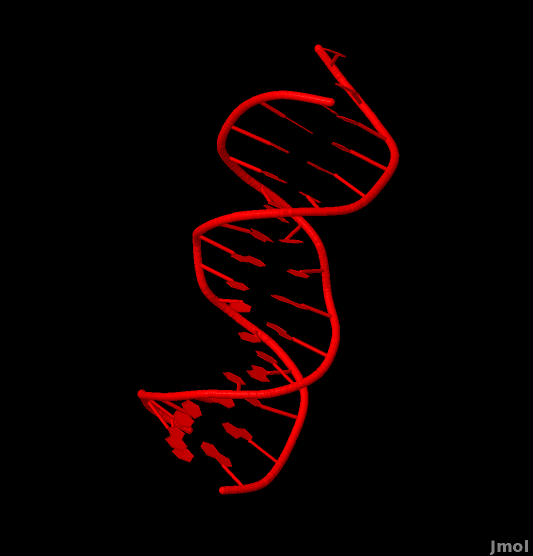
Рис. 2.Изображение ДНК. Изображение получено с помощью Jmol
Задание 3
В данном задании требовалось сравнить структуры трех форм ДНК, используя средства Jmol.
Упражнение 1
Целью упражнения было научиться находить большую и малую бороздки в структуре ДНК, для чего были использованы структуры А- и В-форм ДНК из Задания 1.
B-форма. Выбранное азотистое основание - десятый аденин A10.
В сторону малой бороздки смотрят синие: A10.C2 A10.N3
В сторону большой – красные: A10.N6 A10.C6 A10.C5 A10.C8 A10.N7
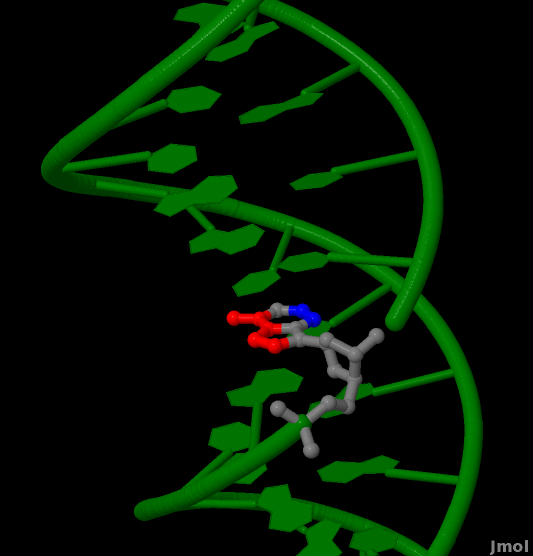
Рис. 3a. B-форма ДНК
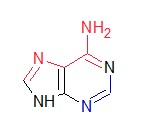
Рис. 3b. Аденин, ChemSketch
A-форма. Азотистое основание то же.
В сторону малой бороздки смотрят синие: A10.N7 A10.C6 A10.N6 A10.C5
В сторону большой бороздки смотрят красные: A10.N3 A10.C2 A10.N1
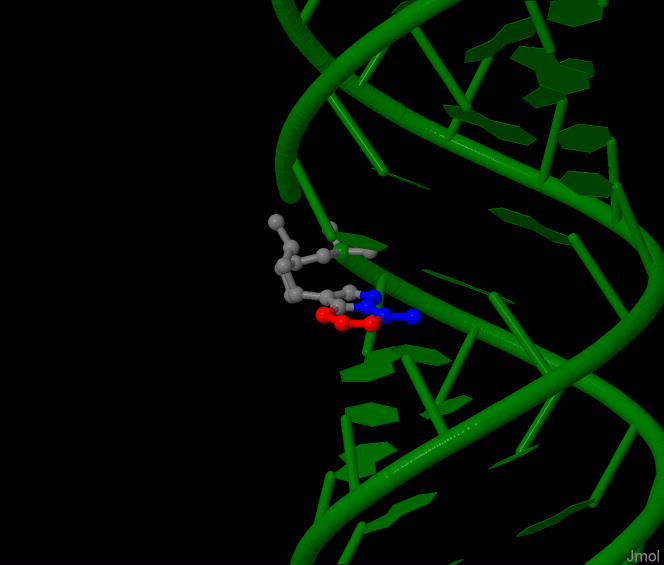
Рис. 4a. A-форма ДНК
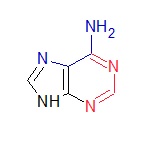
Рис. 4b. Аденин, ChemSketch
Упражнение 2
| Таблица 1. Сравнение основных спиральных параметры разных форм ДНК | |||
|---|---|---|---|
| Свойство | A-форма | В-форма | Z-форма |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (Å) | 28.03 | 33.75 | 43.50 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (Å) | 7.98 | 17.21 | 18.3 |
| Ширина малой бороздки (Å) | 16.81 | 11.69 | 7.2 |
Упражнение 3
| Таблица 2а. Сравнение торсионных углов в структурах А- и В-форм. Данные из презентации. | ||
|---|---|---|
| Угол | A-форма | В-форма |
| α | 62 | 63 |
| β | 173 | 171 |
| γ | 52 | 54 |
| δ | 178 | 123 |
| ε | 88 | 155 |
| ζ | -50 | -90 |
| χ | -160 | -117 |
| Таблица 2b. Сравнение торсионных углов в структурах А- и В-форм. Полученные данные в Jmol. | ||
|---|---|---|
| Угол | A-форма | В-форма |
| α | 64.1 | -60.0 |
| β | 174.8 | -163.4 |
| γ | 41.7 | 31.1 |
| δ | 79.1 | 143.4 |
| ε | -147.8 | -140.8 |
| ζ | -75.1 | -160.5 |
| χ | -157.2 | -98 |
Задание 4
Упражнение 1
Таблицы с торсионными углами:
ДНК:
DNA, самый отклоняющийся 4-й тимин
тРНК:
tRNA, самый отклоняющийся 11-й гуанин
Упражнение 2
Водродные связи:
Акцепторный стебель: Антикодоновый стебель:
(0.011) C:.501_:[..G]G——-C[..C]:.572_:C (0.008) (0.007) C:.539_:[..G]G——-C[..C]:.531_:C (0.003)
(0.008) C:.502_:[..G]G-*—-U[..U]:.571_:C (0.007) (0.009) C:.540_:[..G]G——-C[..C]:.530_:C (0.007)
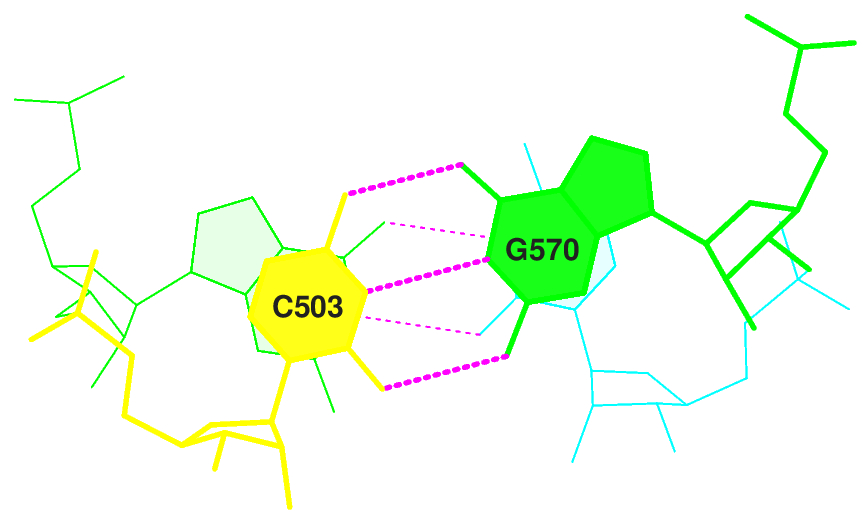
(0.005) C:.503_:[..C]C——-G[..G]:.570_:C (0.004) (0.003) C:.541_:[..C]C——-G[..G]:.529_:C (0.006)
(0.005) C:.504_:[..C]C——-G[..G]:.569_:C (0.008) (0.008) C:.542_:[..C]C——-G[..G]:.528_:C (0.007)
(0.007) C:.505_:[..C]C——-G[..G]:.568_:C (0.009) (0.007) C:.543_:[..G]G——-C[..C]:.527_:C (0.009)
(0.006) C:.506_:[..C]C——-G[..G]:.567_:C (0.004) (0.011) C:.544_:[..A]Ax*—-G[..G]:.526_:C (0.009)
(0.004) C:.507_:[..A]Ax——U[..U]:.566_:C (0.004)
Стабилизация третичной структуры:
Т-стебель: (0.009) C:.555_:[..U]Ux**+xG[..G]:.518_:C (0.014)
(0.008) C:.549_:[..G]G——-C[..C]:.565_:C (0.003) (0.024) C:.515_:[..G]Gx**+xC[..C]:.548_:C (0.013)
(0.006) C:.550_:[..G]G——-C[..C]:.564_:C (0.005) (0.012) C:.519_:[..G]Gx—-xC[..C]:.556_:C (0.003)
(0.005) C:.551_:[..G]G——-C[..C]:.563_:C (0.002)
(0.003) C:.552_:[..G]G——-C[..C]:.562_:C (0.004)
(0.008) C:.553_:[..G]G——xC[..C]:.561_:C (0.004)
D-стебель:
(0.012) C:.510_:[..G]G——-C[..C]:.525_:C (0.005)
(0.005) C:.511_:[..U]U——-A[..A]:.524_:C (0.006)
(0.005) C:.512_:[..C]C——-G[..G]:.523_:C (0.010)
(0.007) C:.513_:[..U]U-*—xG[..G]:.522_:C (0.013)
Упражнение 3
Наибольший вес имеет строчка:
2 GC/GU 7.14( 4.30) 0.00( 0.00) 0.00( 0.00) 6.63( 4.10) 13.76( 8.40)