Выравнивание и гомология
Задание 1
Для построения выравнивания я использовала последовательности девяти белков.
Последовательности можно посмотреть в fasta-файле.
Проект JalView можно скачать по ссылке.
Последовательности были отсортированы по сходству при помощи дерева, построенного методом Neihbour Joining using BLOSUM62 (Рис. 1).
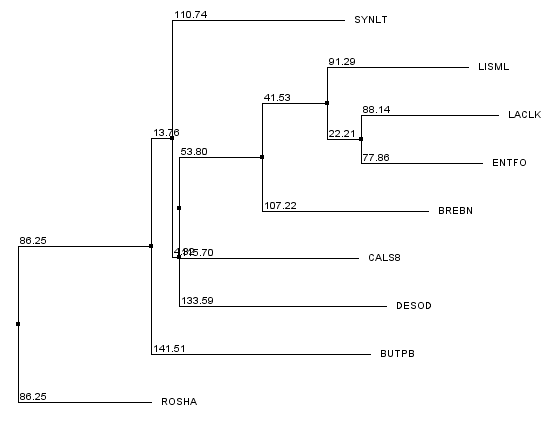
Рис. 1. Дерево для последовательностей из файла "align_06.fasta", получено методом Neihbour Joining using BLOSUM62
Были сделаны изображения выравниваний с раскрасками BLOSUM62 с порогом консервативности 30% и ClustalX. Они представлены на Рис. 2a и Рис. 2b соответственно.
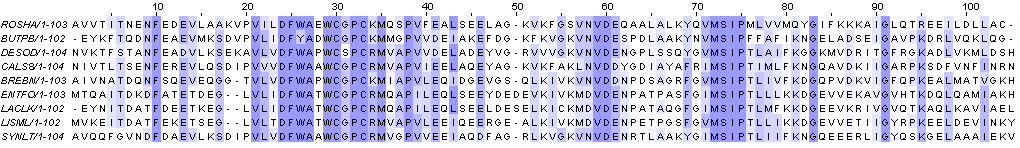
Рис. 2a. Выравнивание с раскраской BLOSUM62, порог консервативности 30%
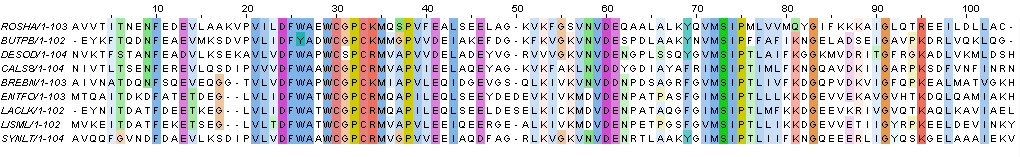
Рис. 2b. Выравнивание с раскраской ClustalX
Задание 2
Была одбавлена строка разметки Clusters, в которой отмечены участки, где можно ожидать гомологию между всеми последовательностями.
Было найдено 3 блока: позиции 21-46, 52-61 и 69-83. Два последних можно объединить в кластер, т.к. между ними нет гэпов.
Самые длинные участки, не входящие в блоки или кластеры, расположены на позициях 1-20 и 84-105 и имеют длину 20 и 21 а.о. соответственно. Выравнивание с разметкой приведено на Рис. 3.

Рис. 3. Выравнивание с раскраской BLOSUM62, порог консервативности 30%.
Обозначения: В - блоки, С - кластеры, Х - самые длинные участки, не входящие в состав блоков или кластеров.
Были выделены 3 последовательности, которые, сокрее всего, составляют отдельную эволюционную ветвь: ENTFO, LACLK и LISML.
Последовательности были объединены в группу (обведены черной рамкой на Рис. 4).
Был выбран участок (позиции 14-16 и 18-20), нельзя говорить о гомологии всех последовательностей выранивания, однако выбранные
последовательности имеют высокое сходство, и можно считать, что аминокислотные остатки в соответствующих позициях для данных
последовательностей гомологичны. На эволюционном дереве (Рис. 1) также видно, что эти последовательности составляют отдельную эволюционную группу.
Задание 3
Для первого блока (обозначен на Рис. 3) были подсчитаны посчитала число и процент абсолютно консервативных позиций, абсолютно функционально консервативных, консервативных и функционально консервативных на 70%. Результат записан в Таблице 1.
| Таблица 1. Число и процент позиций различного типа в первом блоке | |||
|---|---|---|---|
| Позиция | Число | Процент | Номера позиций |
| Абсолютно консервативные | 10 | 38,46% | 21, 24, 25, 27, 29, 30, 32, 33, 35, 38 |
| Абсолютно функционально консервативные | 9 | 34,62% | 22, 26, 34, 39, 40, 41, 43, 45, 46 |
| Консервативные на 70% | 6 | 38,46% | 22, 26, 31, 39, 41, 46 |
| Функционально консервативные на 70% | 2 | 7,69% | 23, 42 |
Задание 4
Была добавлена новая последовательность EUBR3. Были добавлены 2 гэпа на позиции 18, 19. Новое выравнивание представлено на Рис. 4, новая последовательность расположена внизу.
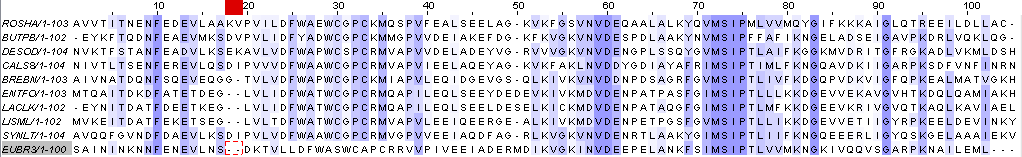
Рис. 4. Новое выравнивание с раскраской BLOSUM62, порог консервативности 30%.
Новая последовательность EUBR3 расположена внизу, добавленные гэпы обведены красным пунктиром.
Задание 5
Была сделана попытка добавить к выравниванию заведомо негомологичную последоваельность (последовательность белка Elongation factor 1-alpha). Эта последовательность намного длиннее, чем все, входящие в исходное выравнивание, поэтому был взят фрагмент длиной 105 а.о. из начала последовательности белка. Результат выравнивания представлен на Рис. 5а и Рис. 5b (раскраски BLOSUM62 с порогом консервативности 30% и ClustalX соответственно).

Рис. 5а. Новое выравнивание с раскраской BLOSUM62, порог консервативности 30%.
Новая последовательность расположена внизу и обозначена YP_003816619.

Рис. 5а. Новое выравнивание с раскраской ClustalX.
Новая последовательность расположена внизу и обозначена YP_003816619.
Как видно из Рис. 5b, некоторые позиции новой негомологичной последовательности совпадают с консервативными позициями в исходном выравнивании (раскрашены одинаково).
Таких позиций 13, т.е. всего совпадений с негомологичным белком 12,38%.
Совпадающие позиции: 10, 22, 27, 57, 59, 60, 72, 74, 77, 79, 95, 98, 102.
Если учитывать сходство по функциональным группам (все гидрофильные и все гидрофобные аминокислоты и сходные по структуре доноры или акцепторы водородных связей),
то число сходств увеличивается до 15, т.е. 14,28%. Совпадающие позиции: 23, 34, 75. Однако такие совпадения можно считать случайными, т.к.
консервативные позиции нельзя объединить в блоки, поэтому участки последовательностей друг другу не гомологичны.
Задание 6
Была сделана попытка создать "выравнивание" заведомо негомологичных белков. Для этого были использованы 5 последовательностей из данного списка белков. Их АС в базе данных UniProt: B8D2N2, I0A0C8, H6Q874, C1DWK1, F0QTE2. "Выравнивание" было построено с помощью команды "Muscle with Default". Результат представлен на Рис. 6. Совпадающих позиций мало, т.к. последовательности негомологичны друг другу. Наилучшие блоки (в которых аминокислотные остатки гомологичны более, чем в половине последовательностей) выделены на Рис. 6 черной рамкой (позиции 130-138, 143-146, 276-282). Наличие таких совпадений можно считать случайным, т.к. их количество достаточно мало.
