EMBOSS
Упражнения
Упражнение 1. (seqret) Несколько файлов в формате fasta собрать в единый файл.
В качестве объединяемых файлов были взяты последовательности белков из Задания 3.1 предыдущего практикума.
Был создан файл со списком названий (команда ls > list), затем при помощи команды seqret @list ex1.fasta последовательности были объединены
в один файл.
Упражнение 2. (seqretsplit) Один файл в формате fasta с несколькими последовательностями разделить на отдельные fasta файлы.
В качестве входного файла был использован файл, полученный в Упражнении 1. При помощи команды seqretsplit ex1.fasta ex2.fasta файл был разделен на 5
отдельных файлов, в каждом из которых хранится последовательность из входного файла.
Упражнение 3. (seqret) Из файла с хромосомой в формате .gb вырезать три кодирующих последовательности по указанным координатам "от", "до",
"ориентация" и сохранить в одном fasta файле.
Последовательность хромосомы, взятой для работы, имеет AC KC784951 в БД GenBank. В этой последовательности были выбраны 3 CDS. Далее был создан listfile с
координатами этих CDS:
- genbank:KC784951[11492:12301]
- genbank:KC784951[13231:13956]
- genbank:KC784951[14117:14644]
Затем при помощи команды seqret @list ex3.fasta три последовательности были объединены в один файл.
Упражнение 4. (transeq) Транслировать кодирующие последовательности, лежащие в одном fasta файле, в аминокислотные, используя указанную
таблицу генетического кода. Результат - в одном fasta файле.
Для работы был взят fasta-файл с кодирующими последовательностями, полученный в Упражнении 3. При помощи команды transeq ex3.fasta ex4.fasta -frame 1
был получен файл с белковыми последовательностями. Стоп-кодон был транслирован как *.
Упражнение 5. (transeq) Транслировать данную нуклеотидную последовательность в шести рамках.
Для работы был использован этот fasta-файл. При помощи команды transeq seq.fasta ex5.fasta -frame 6 был получен
файл с шестью белковыми последовательностями, полученными при транслировании входной последовательности в 6 рамках.
Упражнение 6. (seqret) Перевести выравнивание и из fasta формате в формат .msf
Для работы было взято выравниваниe последовательностей из практикума второго семестра.
При помощи команды seqret align.fasta msf::align.msf был изменен формат файла align.fasta и получен файл, в котором указана
дополнительная информация о выравниваемых последовательностях и выравнивание всех последовательностей.
Сравнение аннотации генов белков в одной хромосоме археи с трансляциями длинных открытых рамок считывания
Для выполнения данного задания была взята архея Acidilobus saccharovorans 345-15, изученная в практикуме первого семестра.
Хромосома у этой археи всего одна, АС ее записи в GenBank - CP001742. Последовательность ее хромосомы в формате fasta можно посмотреть здесь.
Ссылка на файл в формате GenBank здесь.
Задание 1.
В данном задании требовалось получить список трансляций открытых рамок с помощью команды getorf пакета EMBOSS. Для этого сперва была использована
команда getorf CP001742.fasta orfs.fasta -table 11 -minsize 180 -find 0 -circular.
Опция -table задает таблицу генетического кода (здесь 11, т.е. бактериальная таблица), опция -minsize указывает минимальную длину открытой рамки опция,
-find 0 позволяет транслировать найденные рамки от старт-кодона до стоп-кодона, а опция -circular показывает, что данная молекула ДНК имеет кольцевую
структуру.
Результат: fasta-файл с с трансляциями открытых рамок.
Далее был получен список координат и ориентаций найденных открытых рамок с помощью команды infoseq orfs.fasta -outfile orfs.txt -only -name -length -description. Результатом стал текстовый файл, из которого после обработки в Excel и удалении лишней информации была получена таблица, строки которой были отсортированы по началу открытой рамки в геноме (столбец "From").
Задание 2.
Здесь нужно было получить список аннотированных генов белков. Для этого были скачаны файлы в форматах ptt (хромосомная таблица со списком генов белков) и faa (аминокислотные последовательности всех белков в формате fasta). Таблица генов белков была отредактирована в Excel, строки были отсортированы по началу в геноме (столбец "From"). Результат.
Задание 3.
В этом задании было необходимо сравнить полученные ранее таблицы. Для этого таблицы были объединены в одну.
При анализировании получившейся таблицы стало видно, что аннотированных белков почти в 6 раз меньше, чем открытых рамок (1499 против 8788). Возможно, это связано
с тем, что не каждая рамка что-то кодирует (так могло получиться из-за заданной минимальной длины рамки в 180п.н. или из-за перекрывания рамок). Ниже приведены
некоторые конкретные различия:
- Смещение открытой рамки относительно аннотированного гена на 3 нуклеотида (последовательность открытой рамки заканчивается раньше, чем последовательность гена.
Это может быть связано с тем, что открытые рамки учитывают стоп-кодоны, а аннотированные последовательности - нет. На Рис. 1 представлен пример такого сдвига.

Рис. 1. Смещение границ ORF относительно Annotation на 3 нуклеотида на прямой цепи
- Значительное различие в длине у аннотированного гена и открытой рамки. Может быть вызвано с наличием у прокариот альтернативных старт-кодонов: GTG, CTG,
TTG, ATT. Тогда если ген начинается с нестандартного кодона, getorf ищет ближайший кодон AUG, и рамка оказывается длиннее аннотированного гена. На Рис. 2a-b
приведены примеры для прямой и обратной цепи.

Рис. 2a. Различие в длине у аннотированного гена и открытой рамки на прямой цепи

Рис. 2b. Различие в длине у аннотированного гена и открытой рамки на обратной цепи
- Различие в длине аннотированного гена и открытой рамки на 3 нуклеотида. Это может объясняться различием в учитывании стоп-кодона рамкой и геном. Пример
представлен на Рис. 3.

Рис. 3. Различие в длине аннотированного гена и открытой рамки на 3 нуклеотида
- Для аннотированного белка не нашлось открытой рамки. Возможная причина - маленькая длина гена при установленной минимальной длине открытой рамки в 180 п.н.
Пример представлен на Рис. 4.
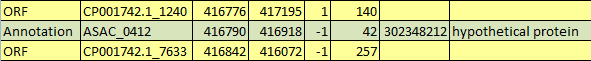
Рис. 4. Аннотированный белок без открытой рамки
- Для длинного аннотированного белка нет рамки. Отличие данного случая от предыдущего в том, что минимальная учтановленная длина открытой рамки не может служить
причиной отсутствия рамки у гена, ведь его длина составляет 1151 п.н. Пример данного явления можно увидеть на Рис. 5.

Рис. 5. Длинный аннотированный белок без открытой рамки
- Пересечение антипараллельных аннотированного белка и открытой рамки. Эти рамки расположены на прямой и на обратной цепи и перекрываются более чем на 150
п.н. Наиболее часто встречающееся явление в таблице. Примере показан на Рис. 5.

- Несколько рамок внутри одного гена. На Рис. 7 видно, что 11 рамок находятся внутри гена ASAC_0317, причем рамки как прямой, так и обратной цепи.
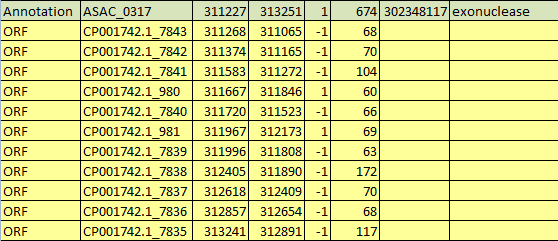
Рис. 7. Несколько рамок внутри одной рамки